Protocollo per HER2 pescare con un fissativo tessuto-cross-linking, privo di formalina per combinare i vantaggi di crioconservazione e fissazione in formalina
Summary
Ibridazione in situ di fluorescenza (pesce) è spesso necessaria in combinazione con l’istopatologia e diagnostica molecolare per la selezione della terapia in medicina personalizzata. Un fissativo di romanzo-cross-linking, privo di formalina tessuto che permette alta qualità morfologiche, molecolare e analisi dei pesci dallo stesso campione tramite l’aggiunta di un passaggio di post-fissazione prima di pesce è presentato.
Abstract
Valutazione morfologica dei campioni di tessuto formalina-fisse, paraffina (FFPE) è stato il gold standard per la diagnostica del cancro per decenni a causa della sua ottima conservazione della morfologia. Medicina personalizzata fornisce sempre più individualmente adattate e mirate terapie per malattie individuali caratterizzati abilitati da tecnologie combinate di analisi morfologiche e molecolare e diagnostica. Prestazioni di analisi morfologiche e molecolari dallo stesso campione di FFPE sono impegnativo a causa dell’impatto negativo di formalina a causa di modificazione chimica e cross-linking di acidi nucleici e proteine. Un fissativo tessuto-cross-linking, privo di formalina è stato recentemente sviluppato per soddisfare entrambi i requisiti, vale a dire, per preservare la morfologia come FFPE e biomolecole come crio-conservazione. Poiché il pesce è spesso necessaria in combinazione con l’istopatologia e diagnostica molecolare, abbiamo testato l’applicabilità dei protocolli di pesce sui tessuti trattati con questo nuovo fissativo. Abbiamo trovato quella post-fissazione formalina di sezioni istologiche dei non-cross-linking, privo di formalina e seno per (NCFPE) a paraffina cancro del tessuto generato risultati equivalenti a quelli con tessuto FFPE nel recettore del fattore di crescita epidermico umano 2 (HER2) Analisi dei pesci. Questo protocollo descrive come un saggio FISH originariamente sviluppato e validato per tessuto FFPE può essere utilizzato per tessuti NCFPE da una semplice fase di post-fissazione delle sezioni istologiche.
Introduction
Medicina personalizzata si basa sempre più su test multi-parametro di analisi morfologica e molecolare del tessuto. Fissazione in formalina di tessuti come il gold standard fornisce eccellenti qualità morfologiche1,2. Tuttavia, modificazione chimica formalina-indotta e cross-linking delle proteine e acidi nucleici3,4,5 negativamente interferisce con l’analisi molecolare6. Queste modificazioni molecolari limitare la qualità di acidi nucleici e proteine e potrebbero gene sequenza manufatti7 o ridotta sensibilità della reazione a catena della polimerasi (PCR)-base di saggi8. Anche se gli sforzi principali sono state prese per ottimizzare i test molecolari per tessuto FFPE, cryo-conservazione di tessuti è in generale superiore alla fissazione in formalina, rendendo necessario dividere i campioni di tessuto per le procedure di conservazione diversi. Per evitare la necessità di cryo-conservazione per analisi molecolari, un non-cross-linking, privo di formalina fissativo, tessuto PAXgene è stato sviluppato. Questo sistema disponibile in commercio è costituito da una fissazione e una soluzione di stabilizzazione contenente diversi alcoli, acido acetico e un composto organico solubile. Buona conservazione degli acidi nucleici, proteine (defosforilata) e morfologia è stata indicata in parecchi studi6,9,10,11,12,13.
Una particolare applicazione nella diagnostica del cancro è pesce rilevare un’ampia gamma di alterazioni cromosomiche, come ad esempio traslocazioni, delezioni submicroscopiche e amplificazioni 14. Ad esempio, il venti per cento dei tumori del cancro al seno invasivo mostrano amplificazione del recettore del fattore di crescita epidermico umano 2 (HER2) gene15,16,17, che è associata con la prognosi difficile18 ,19. Determinazione dello status di sovraespressione e amplificazione di HER2 nel cancro al seno dai pesci è necessario per selezionare i pazienti per la terapia anti-HER2-diretto ed è uno strumento diagnostico elencato dalla Federal Drug Association (FDA) (http://www.fda.gov/ MedicalDevices/ProductsandMedicalProcedures/InVitroDiagnostics/ucm301431.htm)17. Al fine di consentire una vasta applicazione di test diagnostici di accompagnamento a base di pesce nella cura della salute, le analisi sono state sviluppate e approvate per tessuti FFPE.
In uno studio precedente, abbiamo dimostrato che i tessuti NCFPE non possono essere utilizzati per analisi di pesce che sono state approvate per tessuto FFPE. Tuttavia, test di tempo serie di post differenti procedure di fissazione dei tessuti NCFPE con formaldeide 4% tamponato ha mostrato che pubblica i tempi di fissazione di 18 h o più delle sezioni del tessuto NCFPE raggiunti risultati equivalenti a quelli con FFPE tessuti14.
In questo studio, forniamo un protocollo dettagliato e dimostrare che l’impatto del tessuto-cross-linking, privo di formalina fissazione e 24h 4% tamponata la post-fissazione formaldeide su sezioni usato per qualità del pesce di HER2 e RNA dallo stesso campione di tessuto primario.
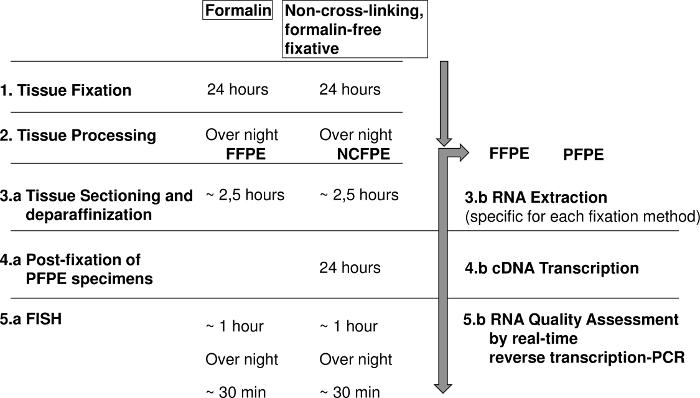
Figura 1: diagramma che mostra i passaggi per ibridazione in situ di fluorescenza (pesce) e analisi di RNA di formalina riparata paraffina (FFPE) e non-cross-linking, privo di formalina fisso paraffina incastonato tessuti (NCFPE). Clicca qui per visualizzare una versione più grande di questa figura.
Protocol
Representative Results
Discussion
I risultati dimostrano due risultati principali. In primo luogo, un passo di post-fissazione Raffaele semplice 24h di sezioni tagliate dai tessuti NCFPE è sufficiente per ottenere risultati equivalenti a quelli con FFPE in analisi dei pesci usando le analisi di pesce approvate per tessuti FFPE (veda inoltre riferimento14). Questo protocollo ha il vantaggio di usando le analisi pesce originariamente sviluppato e approvato per FFPE senza la necessità di riconvalida completa (ad es., ottimizzando le condizioni di pre-ibridazione per tessuto non-cross-linked). Il protocollo di pesce può essere utilizzato esattamente come descritto nelle istruzioni del produttore.
Modifiche minori dalle istruzioni del produttore descritte in questo risultato di protocollo (ad esempio tessuto sezione diametro e incubazione periodi presso i passaggi di sparaffinatura) da precedenti adattamenti, che è esplicitamente raccomandato dal produttore Grazie all’utilizzo di diversi tipi di tessuto e metodi di fissazione.
Il passaggio di post-fissazione è fondamentale perché richiede un tempo di reazione chimico di 18-24 h, che estende il tempo di analisi di un giorno, ma permette il flusso di lavoro quotidiano stesso come sviluppato per tessuti FFPE (Figura 1). Raffaele è segnalato per penetrare il tessuto ad un tasso medio di 1 millimetro al ore22 a 5 mm a 2 h a seconda del tipo di tessuto23,24. L’osservazione che solo lunghi periodi post-fissazione di più di 18 h potrebbero modificare le proprietà di NCFPE di sezioni FFPE, indica che non la penetrazione del fissativo ma il tempo di reazione chimico è fondamentale per raggiungere i risultati desiderati1 ,14.
In secondo luogo, il tessuto restante del NCFPE che non viene utilizzato per la post-fissazione utilizzabile per ulteriori analisi molecolari come le biomolecole sono ben conservate. Questo è stato dimostrato da trascrizione d’inversione Real-Time PCR (Figura 3). Il test è più sensibile e specifico rispetto al valore RIN elettroferogramma-derivato (numero di integrità di RNA) in materia di qualità di RNA (cioè, modificazione chimica e frammentazione) per mRNA isolato da tessuti paraffina-incastonati8. Controllo di qualità è stato limitato in questo studio a PCR in tempo reale poiché gruppi indipendenti hanno dimostrato che oltre a RNA, la qualità di DNA e proteine isolate dal NCFPE è migliore di quella da FFPE tessuti 8,9, 13.
Il protocollo presentato segue, ove applicabile (ad es., ricetta di Raffaele, condizioni di fissazione, controllo di qualità di RNA, convalida), i requisiti delle specifiche tecniche CEN per le procedure pre-analitiche per diagnostica in vitro (IVD), che sono stati recentemente rilasciati dal Comitato europeo di normalizzazione (CEN) (ad es. CEN/TS 16827-1:2015 per isolamento del RNA dai tessuti FFPE che si riferisce allo standard ISO 15189).
Il metodo è limitato a questo fissativo specifico. Anche se la buona conservazione delle biomolecole e morfologia utilizzando il fissativo-cross-linking, formalina-free è stata presentata in diversi studi, è usato principalmente nella ricerca, ma non in applicazioni mediche di routine. Uno dei motivi è che sostituendo il gold standard Raffaele fissazione da parte di un altro fissativo richiederebbe una varietà di studi di validazione come non tutti i protocolli ottimizzati per materiale FFPE possono essere utilizzati per materiali fissate con fissativi-cross-linking. Altri metodi di fissazione del tessuto non sono stati testati per questo protocollo di pesce.
Il semplice passaggio di post-fissazione descritto in questo manoscritto presenta il vantaggio di usando le analisi IVD approvato sia per tessuti FFPE e NCFPE.
Disclosures
The authors have nothing to disclose.
Acknowledgements
Ringraziamo il team del laboratorio di patologia molecolare presso l’Istituto di patologia dell’Università medica di Graz per la loro competenza e supporto. Inoltre, si ringraziano Iris Kufferath e Daniela Pabst per assistenza tecnica, Bernadette Rieger e Sylvia Eidenhammer per l’elaborazione di casi HER2 come pure Kinga Szurian (patologo) e Tamas Regényi (3DHISTEC). Vi ringraziamo di correzione di bozze del manoscritto Penelope Kungl. Questo lavoro è stato sostenuto finanziariamente da Christian Doppler ricerca del fondo, il Ministero federale austriaco della scienza, ricerca ed economia e Fondazione nazionale per ricerca e sviluppo tecnologico.
Materials
| SAV LP GmbH, Flintsbach a. I., Germany | www.sav-lp.de | FN-200L-4-1 | Tissue fixative |
| Simport Plastic Ltd., Beloeil, Canada | www.simport.com | M-498 | Safekeeping device providing labelling, fixation and paraffin embedding for tissue samples. |
| Qiagen GmbH, Hilden Germany | www.qiagen.com | 765112 | For collection, fixation, and stabilization of tissue samples: 10 Prefilled Reagent Containers, containing PAXgene Tissue FIX and PAXgene Tissue STABILIZER. |
| ThermoFisher Scientific | www.thermofisher.com | 813150 | Processing of biological specimens from chemical fixation to paraffin infiltration |
| Merck Millipore, Munich, Germany | www.analytics-shop.com/de/hersteller/millipore.html | MC1009834000 | Dehydration of tissue samples |
| VWR Chemicals, Darmstadt, Germany | https://at.vwr.com | 10293EP | Dehydration of tissue samples |
| ACM Herba Chemosan Apotheker AG, Vienna, Austria | www.herba-chemosan.at | 2549662 32 | Paraffin used in a tissue processing device |
| Leica Mikrosysteme Handels GmbH, Vienna, Austria | www.LeicaBiosystems.com | 39602004 | Paraffin embedding medium |
| Sanova Diagnostik, Vienna, Austria | www.sanova.at | 5229 | Paraffin embedding instrument for tissues for histology |
| Histocom Medizintechnik Vertriebs GmbH, Wiener Neudorf, Austria | www.histocom.info | M 910010 | This device is used for especially sophisticated paraffin sectioning techniques in biology and medicine. Only skilled or specially trained personnel must operate the microtome, i.e. clamping the specimen, trimming, sectioning and taking off the sections from the instrument. |
| Dako Denmark A/S, Glostrup, Denmark | www.chem.agilent.com | K802021-2 | Coated glass slides, intended for mounting formalin-fixed, paraffin-embedded tissue sections. |
| Qiagen GmbH, Hilden Germany | www.qiagen.com | 73504 | For purification of total RNA from formalin-fixed, paraffin-embedded tissue sections |
| Qiagen GmbH, Hilden Germany | www.qiagen.com | 765134 | For purification of total RNA from PAXgene-fixed, paraffin-embedded tissue sections |
| ZytoVision GmbH, Bremerhaven, Germany | www.zytovision.com | Z-2015-200 | For the detection of ERBB2 (a.k.a HER2) gene amplification frequently observed in solid malignant neoplasms e.g. breast cancer samples. |
| 3DHISTEC, Budapest, Hungary | www.3dhistech.com | Digital device for scanning tissue slides equipped with a 40x/1.2 NA objective, Quad band filters (DAPI/FITC/TRITC/Cy5) filters and a 5.5Mpx, 16 bit, cooled scientific CMOS (Complementary Metal Oxide Semiconductor) camera. | |
| ThermoFisher Scientific | www.thermofisher.com | 4368814 | The High Capacity cDNA Reverse Transcription Kit contains all components necessary for the quantitative conversion of up to 2 µg of total RNA to single-stranded cDNA in a single 20 µL reaction. |
| ThermoFisher Scientific | www.thermofisher.com | 4367659 | PCR Master Mix containing polymerase, nucleotides and SYBR Green for PCR and quanitification of amplicons. |
| ThermoFisher Scientific, Wilmington, DE | www.thermofisher.com | Ser. Nr. F239 | Spectrophotometer for nucleic acid quantification. |
| QuantStudio 7 Flex Real-time PCR System | www.thermofisher.com | 4485701 | PCR machine, |
| Vysis/Abbott Laboratories. Abbott Park, Illinois, U.S.A | www.molecular.abbott/us/en/products/instrumentation/thermobrite#order | e.g. (ThermoBrite) 07J91-020 | Temperature controlled slide processing system for in-situ denaturation/hybridization procedures. |
References
- Fox, C. H., Johnson, F. B., Whiting, J., Roller, R. P. Formaldehyde fixation. J. Histochem.Cytochem. 33 (8), 845-853 (1985).
- Oosterhuis, J. W., Coebergh, J. W., van Veen, E. B. Tumour banks: well-guarded treasures in the interest of patients. Nat. Rev. Cancer. 3 (1), 73-77 (2003).
- Masuda, N., Ohnishi, T., Kawamoto, S., Monden, M., Okubo, K. Analysis of chemical modification of RNA from formalin-fixed samples and optimization of molecular biology applications for such samples. Nucleic Acids Res. 27 (22), 4436-4443 (1999).
- Metz, B., Kersten, G. F., Hoogerhout, P., Brugghe, H. F., Timmermans, H. A., de Jong, A., et al. Identification of formaldehyde-induced modifications in proteins: reactionswith model peptides. J. Biol. Chem. 279 (8), 6235-6243 (2003).
- Evers, D. L., Fowler, C. B., Cunningham, B. R., Mason, J. T., O’Leary, T. J. The effect of formaldehyde fixation on RNA: optimization of formaldehyde adduct removal. J.Mol. Diagn. 13 (3), 282-288 (2011).
- Groelz, D., Sobin, L., Branton, P., Compton, C., Wyrich, R., Rainen, L. Non-formalin fixative versus formalin-fixed tissue: A comparison of histology and RNA quality. Exp. Mol. Pathol. 94 (1), 188-194 (2013).
- Do, H., Dobrovic, A. Sequence artifacts in DNA from formalin-fixed tissues: causes and strategies for minimization. Clin. Chem. 61, 64-71 (2015).
- Kashofer, K., Viertler, C., Pichler, M., Zatloukal, K. Quality Control of RNA Preservation and Extraction from Paraffin-Embedded Tissue: Implications for RT-PCR and Microarray Analysis. PloS One. 8 (7), e70714 (2013).
- Viertler, C., Groelz, D., Gündisch, S., Kashofer, K., Reischauer, B., Riegman, P. H., et al. A new technology for stabilization of biomolecules in tissues for combined histological and molecular analyses. J. Mol. Diagn. 14 (5), 458-466 (2012).
- Gundisch, S., Slotta-Huspenina, J., Verderio, P., Maura, C., Ciniselli, S. P., Schott, S., et al. Evaluation of colon cancer histomorphology: A comparison between formalin and PAXgene tissue fixation by an international ring trial. Virchows Arch. 465 (5), 509-519 (2014).
- Ergin, B., Meding, S., Langer, R., Kap, M., Viertler, C., Schott, C., et al. Proteomic analysis of PAXgene-fixed tissues. J. Proteome Res. 9 (10), 5188-5196 (2010).
- Kap, M., Smedts, F., Oosterhuis, W., Winther, R., Christensen, N., Reischauer, B., et al. Histological Assessment of PAXgene Tissue Fixation and Stabilization Reagents. PLoS ONE. 6 (11), e27704 (2011).
- Mathieson, W., Marcon, N., Antunes, L., Ashford, D. A., Betsou, F., Frasquilho, S. G., et al. A Critical Evaluation of the PAXgene Tissue Fixation System: Morphology, Immunohistochemistry, Molecular Biology, and Proteomics. Am J Clin Pathol. 146, 25-40 (2016).
- Oberauner-Wappis, L., Loibner, M., Viertler, C., Groelz, D., Wyrich, R., Zatloukal, K. Protocol for HER2 FISH determination on PAXgene-fixed and paraffin-embedded tissue in breast cancer. Int J. Exp. Path. 97 (2), 202-206 (2016).
- Pauletti, G., Godolphin, W., Press, M. F., Slamon, D. J. Detection and quantitation ofHER-2/neu gene amplification in human breast cancer archival material using fluorescence in situ hybridization. Oncogene. 13 (1), 63-72 (1996).
- Owens, M. A., Horten, B. C., Da Silva, M. M. HER2 amplification ratios by fluorescence in situ hybridization and correlation with immunohistochemistry in a cohort of 6556 breast cancer tissues. Clin.Breast Cancer. 5 (1), 63-69 (2004).
- Wolff, A. C., Hammond, M. E., Schwartz, J. N., Hagerty, K. L., Allred, D. C., Cote, R. J., et al. American Society of Clinical Oncology/College of American Pathologists guideline recommendations for human epidermal growth factor receptor 2 testing in breast cancer. Arch Pathol Lab Med. 131 (1), 18-43 (2007).
- Press, M. F., Bernstein, L., Thomas, P. A., Meisner, L. F., Zhou, J. Y., Ma, Y., et al. HER-2/neu gene amplification characterized by fluorescence in situ hybridization: poor prognosis in node-negative breast carcinomas. J. Clin. Oncol. 15 (8), 2894-2904 (1997).
- Yamauchi, H., Stearns, V., Hayes, D. F. When is a tumor marker ready for prime time? A case study of c-erbB-2 as a predictive factor in breast cancer. J. Clin. Oncol. 19 (8), 2334-2356 (2001).
- Paulik, R., Micsik, T., Kiszler, G., Kaszál, P., Székely, J., Paulik, N., Várhalmi, E., Prémusz, V., Krenács, T., Molnár, B. An optimized image analysis algorithm for detecting nuclear signals in digital whole slides for histopathology. Cytometry A. , (2017).
- Applied Biosystems. . QuantStudio 6 and 7 Flex Real-Time PCR System Software. , (2013).
- NCCLS. . Quality Assurance for Immunocytochemistry. , (1999).
- Baker, J. R. . Principles of Biological Microtechnique. , (1958).
- Hewitt, S. M., Lewis, F., Cao, Y., Conrad, R. C., Cronin, M., Danenberg, K. D., et al. Tissue handling and specimen preparation in surgical pathology: issues concerning the recovery of nucleic acids from formalin-fixed, paraffin-embedded tissue. Arch Pathol. Lab Med. 132 (12), 1929-1935 (2008).

