הערכה סימולטני של DNA Cardiomyocyte סינתזה ploidy: שיטה Assist כימות Cardiomyocyte רגנרציה מחזור
Summary
Quantification of cardiomyocyte turnover is challenging. The protocol described here makes an important contribution to this challenge by enabling accurate and sensitive quantification of neo-cardiomyocyte nuclei generation and nuclei ploidy.
Abstract
למרות זאת הוא קיבל את זה בלב יש פוטנציאל מוגבל להתחדש cardiomyocytes בעקבות פציעה וכי רמות נמוכות של מחזור cardiomyocyte להתרחש במהלך הזדקנות נורמלית, כימות של אירועים אלה נותר מאתגר. מדובר בין השאר בשל הנדירות של תהליך והעובדה מקורות תאיים רבים לתרום תחזוקה שריר הלב. יתר על כן, שכפול ה- DNA בתוך cardiomyocytes לעתים קרובות מוביל cardiomyocyte polyploid ורק לעיתים רחוקות מוביל cardiomyocytes חדש על ידי חטיבת הסלולר. כדי לכמת במדויק אפליה מחזור cardiomyocyte בין תהליכים אלה היא חיונית. הפרוטוקול המתואר כאן מעסיקה תיוג nucleoside לטווח ארוך כדי לתייג כל הגרעינים שהתעוררו כתוצאה גרעינים שכפול דנ"א cardiomyocyte מזוהה על ידי ניצול בידוד הגרעינים ואת immunolabeling PCM1 שלאחר מכן. יחד זה מאפשר זיהוי מדויק ורגיש של תיוג nucleoside של גardiomyocyte אוכלוסיית גרעינים. יתר על כן, 4 ', 6-diamidino-2-phenylindole תיוג וניתוח ploidy גרעינים, מאפשרת אפליה של גרעינים הניאו-cardiomyocyte מגרעינים אשר שילבו nucleoside במהלך polyploidization. אמנם שיטה זו לא יכולה לשלוט על cardiomyocyte binucleation, היא מאפשרת כימות מהיר ויציב של גרעינים הניאו-cardiomyocyte תוך התחשבות polyploidization. לשיטה זו מספר היישומים במורד זרם כוללים בחינה של הרפוי הפוטנציאלי כדי לשפר התחדשות cardiomyocyte או שבדק את ההשפעה של מחלת לב על מחזור cardiomyocyte ו ploidy. טכניקה זו היא גם תואמת עם טכניקות immunohistological במורד נוספות, המאפשרות כימותי התאגדות nucleoside בכל סוגי תאי הלב.
Introduction
בשנים האחרונות חלה ולראיות תיגר על ההנחה כי הלב הוא איבר 1,2 הבדיל סופני, שלאחר mitotic. עם זאת, כימות של מחזור cardiomyocyte והתחדשות נותרת מאתגרת.
הקשיים בזיהוי דור cardiomyocyte נדיר במדויק תוך שימוש בטכניקות immunohistochemical סטנדרטיים גם מדווחים 3. בנוסף, מקור הסלולר של דור cardiomyocyte נשאר לא ברור עם ראיות לתרומות על ידי התפשטות cardiomyocyte וכן התמיינות תאי גזע 4-6. לכן, שימוש במודלים התחקות השושלת אשר דורשים ידע של פנוטיפ cardiomyocyte אבי הוא בלתי אפשרי וכימות של התפשטות מאוכלוסיה אחת, כולל שריר הלב, אינו ראוי. יתר על כן cardiomyocyte יש פוטנציאל endoreplication ללא karyokinesis (וכתוצאה מכך המכונית polyploiddiomyocyte) או karyokinesis בהעדר cytokinesis (וכתוצאה מכך cardiomyocyte binucleated) 7,8. כימות מדויק של מחזור cardiomyocyte תלויה ביכולת להבחין בינם ובין הדור הניאו-cardiomyocyte נכון. זה יוצר סיבוכים ייחודיים משום שכפול הדנ"א וביטוי של קינאזות תלויות ציקלין ב cardiomyocytes לא בלעדי להפגין חלוקת תא נכון 9,10.
כדי לסייע כימות של הדור הניאו-cardiomyocyte, יש לנו בשילוב טכניקה בידוד גרעינים הוקמה, וסימון אימונולוגיים של pericentriolar חומר 1 (PCM1) לזיהוי גרעינים cardiomyocyte כפי שתואר על ידי ברגמן et al. 7,11 עם שיטות חדשניות של טווח ארוך תיוג DNA וניתוח ploidy. PCM-1 הוא חלבון centrosome שמצטבר על פני שטח הגרעין של מיוציטים בדיל, הלא אופניים. מחקרים קודמים הראו כי נוגדנים נגדPCM-1 תווית במיוחד גרעינים cardiomyocyte 7,11 וכפי PCM1 נוצל בעבר על ידי מספר קבוצות עצמאיות לזהות 1,12,13 cardiomyocytes כזה. בנוסף, אנו הראינו כי ביטוי PCM1 מפות כדי גרעיני cardiomyocyte שכותרתו גנטית במודל העכבר המהונדס-cre TnT 14 (איור 1 משלים).
הפרוטוקול המתואר כאן מאפשר זיהוי המדויק ורגיש של דור גרעיני cardiomyocyte ניאו בלב העכבר, ללא קשר המקור התאי (איור 1 א 'וב') תוך הדרת תיוג nucleoside זמנית בשל polyploidization מהניתוח (איור 1 ג ו-ד). אמנם שיטה זו לא יכולה לשלוט על cardiomyocyte binucleation, היא מאפשרת כימות מהיר ויציב של גרעינים הניאו-cardiomyocyte אשר נדרש עבור כימות מדויק של מחזור cardiomyocyte. יתר על כן,הוא מספק כלי מיון מהירים להעריך שינויים פוטנציאליים הדינמיקה של דור cardiomyocyte.
בעוד תיוג DNA בדרך כלל 5-Bromo-2'-deoxyuridine (BrdU) כמו אנלוגי thymidine, פרוטוקול המתואר כאן משתמשת assay מבוסס 5 ethynyl-2'-deoxyuridine (EDU) כפי שהוא דורש מדרגות עיבוד פחות עבור יותר מהיר דרך-לשים ואינו דורש denaturing של DNA כדי לאפשר גילוי חיסוני, מה שהופך אותו תואם פרוטוקולי immunostaining אחרים ובכך הגדיל את היישומים במורד הפוטנציאל של השיטה.
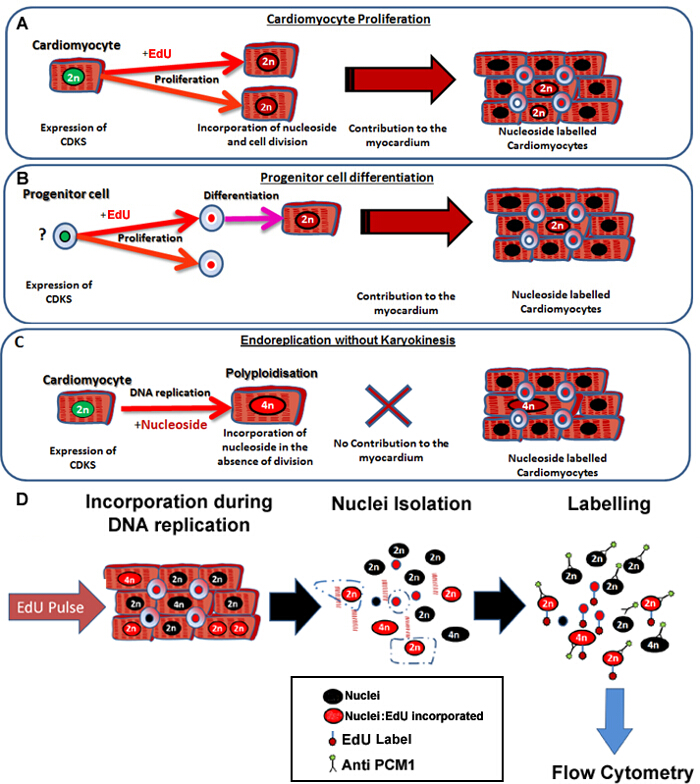
איור 1: פועם רציף עם edu תוויות הניאו-cardiomyocytes ללא קשר אבות שלהם. (א) edu הוא שולב DNA של cardiomyocytes בעת חלוקת התא. הפצת נשק באוכלוסייה cardiomyocyte יהיה Result לעלייה, או החלפה של cardiomyocytes ולכן הוא סינתזה של DNA פרודוקטיבי (תורם תחזוקה ותיקון רקמות). (ב) edu הוא שולב DNA של ובתאים לב בעת חלוקת התא. זה יישמר בתא במהלך בידול אל שושלת cardiomyocyte. התמיינות תאי גזע זה תביא גם לעלייה במספר cardiomyocytes ולכן תורמת תחזוקה ותיקון רקמות. (C) cardiomyocytes יש פוטנציאל לעבור "לא פרודוקטיביים" שכפול הדנ"א וכתוצאה מכך ploidy cardiomyocyte גדל, אשר מזוהה עם היפרטרופיה cardiomyocyte שיפוץ שריר הלב, אך אינו מחליף cardiomyocytes האבוד. תהליך polyploidization נבדל binucleation כמו התוצאה היא cardiomyocyte עם גרעינים יחידים אשר מכיל ארבעה או יותר סטים של שני כרומוזומים הומולוגיים (> 2N). (ד) בעקבות p גרעינים רציףulse, פרוטוקול זה מתאר בידוד גרעינים וזיהוי של גרעינים cardiomyocyte ידי ביטוי PCM1 לאפשר כימות של שני ploidy cardiomyocyte ושילוב edu. ביטוי PCM1 ושילוב edu זוהה באמצעות cytometry הזרימה. אנא לחץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
Protocol
Representative Results
Discussion
כדי לכמת במדויק מחזור cardiomyocyte מבחני התחדשות יש להבחין בין הדור cardiomyocyte נכון וחילוק DNA פרודוקטיבי. מחקרים רבים ממשיכים פשוט להתעלם אירועים בלתי יצרניים אלה, לכימות התפשטות cardiomyocyte אך ורק דרך הביטוי של נִיעוּת cyclin וסמני מחזור התא. עד כה שיטה אחת המאפשרת הכימות המדויק של מחזור cardiomyocyte תוך פיקוח על אירועים בלתי יצרניים אלה נשאר מרומז. בפרט הוא נשאר קשה להסביר polyploidization cardiomyocyte התורמת עד ~ 65% של שכפול הדנ"א cardiomyocyte 13. לכן כדי לסייע כימות המדויק של דור cardiomyocyte פתחנו פרוטוקול המאפשר כימותים החזקים של שיעורי גרעיני ניאו cardiomyocyte תוך הדרת שכפול הדנ"א שתוצאתו ploidy המוגברת. למרות בפרוטוקול זה לא יכול אפליה בין סוגים הניאו-cardiomyocytetion ו cardiomyocyte דו-התגרענות, ניתן להשתמש בו במהירות ובאופן מדויק לחשב את הגבול העליון (והיוו ploidy) של הדור cardiomyocyte. פרוטוקול זה ולכן מספק כלי הקרנה להעריך שינויים אפשריים בשיעורי דור cardiomyocyte ו polyploidization במודלי מחלה או להעריך את היעילות הפוטנציאלית של רפויים. לאחר שינויים בשער של דור גרעינים-cardiomyocyte ניאו מזוהה באמצעות פרוטוקול זה מחקרים מאוחרים יותר ניתן להשתמש כדי לבדוק אם זה עקב שינויים דורות cardiomyocyte של מספר התגרענות cardiomyocyte, כפי שתוארו לעיל 2,13,17,18. אלה כוללים את שימוש דינמיקת התגרענות cardiomyocyte כימות היסטולוגית בתקופת הדופק או מנתח סעיפי רקמות המתקבל חי פעמיו edu כדי להשוות התאגדות edu באוכלוסיות cardiomyocyte mononucleated ו multinucleated.
בשל הרמות הנמוכות של מחזור cardiomyocyte זהפרוטוקול עושה שימוש זריקות מרובות של edu פני תקופה של 7 ימים. זה גם מאפשר "רודף" של כל מקורות הסלולר הפוטנציאל של דור cardiomyocyte ומתיר כימות של דור גרעיני cardiomyocyte המצטבר במשך פרק זמן זה. בהתאם המחקר, זמן זה עשוי להיות מותאם כדי להתאים את הרמות החזויות של דור cardiomyocyte. עבור כימות מדויק ההתאגדות edu בגרעינים cardiomyocyte, קיים הכרח כי אין תיוג הלא ספציפית של גרעינים עם נוגדנים משני המשמשים לאיתור תגובתיות PCM-1. לכן יהיה זה נבון לבצע ניסויי טיטרציה נוגדנים משני נוספים על מנת לייעל את ההיבט הזה של הפרוטוקול, במיוחד אם נוגדנים משני חוץ מזה הציעו בפרוטוקול זה הוא לשמש. הפרוטוקול המתואר כאן משתמש בביטוי PCM-1 לזהות גרעיני cardiomyocyte. אמנם זה מהווה סמן cardiomyocyte הוקמה, סמנים חלופיים שניתן להשתמש בהם כדי לאמתנתונים; אלה כוללים נוגדנים ספציפיים עבור לב Troponin T אשר זוהה בחלקו מקומי גרעיני cardiomyocyte 1. בדומה לכך, חלבונים מקומיים גרעיניים חלופיים ניתן להשתמש כדי לזהות ולכמת התאגדות edu באוכלוסיות גרעינים אחרות מזה של cardiomyocytes. חשוב כי כל הגרעינים cardiomyocyte כי עוברים פעיל מיטוזה מודרים מן הניתוח, כמו גורלם של סינתזת ה- DNA הזה אינו ידוע ועלול לגרום גם חלוקת התא או ploidy מוגברת. PCM1 הוא מפורק בשלב M של מחזור התא ולכן cardiomyocytes שעברו מיטוזה לא יזוהה על ידי ביטוי PCM1. בנוסף, כל הגרעינים בשלב S של מחזור התא צריך להיות מחוץ לניתוח שלאחר מכן. זו יכולה להיות מושגת על ידי gating את כל הגרעינים בעוצמה DAPI מעל האוכלוסייה 2N כולל אלה עם עוצמת DAPI בין 2N ו 4N אוכלוסיות.
למרות זאת הוא יותר ויותרקיבל שהלב את היכולת להחליף cardiomyocytes במהלך הזדקנות נורמלית לבין פגיעה חריפה הבאה, את המקור ואת מידת הפוטנציאל הזה נשאר שנוי במחלוקת. בנוסף, שיעורים מפורד ממחזור cardiomyocyte דווחו 1,7,20-22. זה עשוי להיות בין השאר בשל קשיים טכניים לאתר במדויק וכימות הדור הניאו-cardiomyocyte 19. עד כה רוב המחקרים התבססו רק על השימוש בניתוח היסטולוגית והזיהוי של cardiomyocytes באמצעות ביטוי של חלבונים cytoplasmic, כולל חלבונים של sarcomere, כימות של מחזור cardiomyocyte והתחדשות 2,4,23,24. השימוש בשיטות אלה כדי לזהות את הביטוי של סמני התפשטות, או כפי שהודגם כאן, השילוב של אנלוגים thymidine יכול לגרום בקלות מזיהוי השגויה של סוגי תאים לבביים אחרים כמו cardiomyocytes. בעוד השימוש הדמיה confocal 3D יכול לעזור כדי להקל על בעיות אלה השיטות אסה יקרות זמן רב. מעניין לציין, כי הפרוטוקול המתואר כאן מדגים הדור גרעינים-cardiomyocyte ניאו מתרחשת בקצב של 0.17% בשבוע. זה עולה בקנה אחד עם זרימת אחרים cytometry מחקרים המבוססים הוכחת שיעורי תחלופה שבועית של עד 0.13% 5. למרות שזה מפתה להסיק שיעורי מחזור שנתיים על פי נתונים אלו, כמו במחקרים קודמים 2,5,25,26, זו אינה מתאימה כמו ושיעור העזיבה הם דינמיים במהלך זמן חיים של חיה 13.
לשיטה זו מספר היישומים הפוטנציאליים כוללים בחינה של הרפוי הפוטנציאלי כדי לשפר התחדשות cardiomyocyte או שבדק את ההשפעה של מחלת לב על מחזור cardiomyocyte ושיעורי polyploidization cardiomyocyte.
Disclosures
The authors have nothing to disclose.
Acknowledgements
This work was supported by the British Heart Foundation, project grant PG/13/69/30454.
Materials
| 0.32 M sucrose | Sigma | 84100 | |
| 10 mM Tris-HCl (pH = 8) | Sigma | T3253 | |
| 5 mM CaCl2 | Sigma | c5086 | |
| 5 mM magnesium acetate | sigma | M-5661 | |
| 2.0 mM EDTA | Sigma | E5134 | |
| 0.5 mM EGTA | Sigma | 63779 | |
| 1 mM DTT | Sigma | D0632 | |
| 70 mM KCl | Sigma | P9541 | |
| 10 mM MgCl2 | Sigma | M8266 | |
| 1.5 mM spermine | Sigma | 85590 | |
| Isotype rabbit IgG- ChIP Grade | abcam | abc7415 | |
| Rabbit anti-PCM-1 antibody | Sigma | HPA023374 | |
| Alexa Fluor 488 F(ab')2 Fragment of Goat Anti-Rabbit IgG (H+L) Antibody | Life technologies | A-11070 | |
| cell strainers 70 μm and 100μm | Fisher scientific | 11597522, 11517532 | |
| Glass dounce (40 ml) and pestle large clearance | Sigma | D9188-1SET | |
| EdU (5-ethynyl-2’-deoxyuridine) | Life technologies | A10044 | |
| Click-iT Plus EdU Alexa Fluor 647 Flow Cytometry Assay Kit | Life technologies | C10634 | This kit inlcudes reagents required for section, EdU reaction buffer, EdU fixative, saponin-based permeabilization solution and the reagents required for the EdU labelling cocktail. |
| CyStain DNA 2 step kit, | Sysmex Partec | 05 5005 | This kit inlcudes reagents required for DAPI labelling (DNA staining solution) |
| Probe homogeniser e.g. TissueRuptor | Qiagen | 9001273 | |
| TissueRuptor Disposable Probes | Qiagen | 990890 | |
| ultracentrifuge | Sorvall | ||
| Facscanto II | BD Biosciences | ||
| Ultracentrifuge Tube, Thinwall, Polypropylene. 38.5 mL, 25 x 89 mm | Beckman Coulter | 326823 | |
| Bovine serum albumin | Sigma | A2153 |
References
- Bergmann, O., et al. Evidence for cardiomyocyte renewal in humans. Science. 324, 98-102 (2009).
- Senyo, S. E., et al. Mammalian heart renewal by pre-existing cardiomyocytes. Nature. 493, 433-436 (2013).
- Soonpaa, M. H., Rubart, M., Field, L. J. Challenges measuring cardiomyocyte renewal. Biochim Biophys Acta. 1833, 799-803 (2013).
- Loffredo, F. S., Steinhauser, M. L., Gannon, J., Lee, R. T. Bone marrow-derived cell therapy stimulates endogenous cardiomyocyte progenitors and promotes cardiac repair. Cell Stem Cell. 8, 389-398 (2011).
- Malliaras, K., et al. Cardiomyocyte proliferation and progenitor cell recruitment underlie therapeutic regeneration after myocardial infarction in the adult mouse heart. EMBO Mol Med. 5, 191-209 (2013).
- Hsieh, P. C., et al. Evidence from a genetic fate-mapping study that stem cells refresh adult mammalian cardiomyocytes after injury. Nat Med. 13, 970-974 (2007).
- Bergmann, O., et al. Identification of cardiomyocyte nuclei and assessment of ploidy for the analysis of cell turnover. Exp Cell Res. 317, 188-194 (2011).
- Herget, G. W., Neuburger, M., Plagwitz, R., Adler, C. P. DNA content, ploidy level and number of nuclei in the human heart after myocardial infarction. Cardiovasc Res. 36, 45-51 (1997).
- Scholzen, T., Gerdes, J. The Ki-67 protein: from the known and the unknown. J Cell Physiol. 182, 311-322 (2000).
- Carmena, M., Earnshaw, W. C. The cellular geography of aurora kinases. Nat Rev Mol Cell Biol. 4, 842-854 (2003).
- Bergmann, O., Jovinge, S. Isolation of Cardiomyocyte Nuclei from Post-mortem Tissue. J. Vis. Exp. , e4205 (2012).
- Gilsbach, R., et al. Dynamic DNA methylation orchestrates cardiomyocyte development, maturation and disease. Nat Commun. 5, 5288 (2014).
- Richardson, G., Laval, S., Owens, W. A. Cardiomyocyte regeneration in the mdx mouse model of non-ischemic cardiomyopathy. Stem Cells Dev. , (2015).
- Jiao, K., et al. An essential role of Bmp4 in the atrioventricular septation of the mouse heart. Genes Dev. 17, 2362-2367 (2003).
- Bergmann, O., et al. Cardiomyocyte renewal in humans. Circ Res. 110, e17-e18 (2012).
- Prigge, J. R., et al. Nuclear double-fluorescent reporter for in vivo and ex vivo analyses of biological transitions in mouse nuclei. Mamm Genome. , (2013).
- Naqvi, N., et al. A proliferative burst during preadolescence establishes the final cardiomyocyte number. Cell. 157, 795-807 (2014).
- Liu, Z., Yue, S., Chen, X., Kubin, T., Braun, T. Regulation of cardiomyocyte polyploidy and multinucleation by CyclinG1. Circ Res. 106, 1498-1506 (2010).
- Ang, K. L., et al. Limitations of conventional approaches to identify myocyte nuclei in histologic sections of the heart. Am J Physiol Cell Physiol. 298, C1603-C1609 (2010).
- Kajstura, J., et al. Myocyte turnover in the aging human heart. Circ Res. 107, 1374-1386 (2010).
- Kajstura, J., et al. Cardiomyogenesis in the adult human heart. Circ Res. 107, 305-315 (2010).
- Walsh, S., Ponten, A., Fleischmann, B. K., Jovinge, S. Cardiomyocyte cell cycle control and growth estimation in vivo–an analysis based on cardiomyocyte nuclei. Cardiovasc Res. 86, 365-373 (2010).
- Anversa, P., Leri, A., Kajstura, J. Cardiac regeneration. J Am Coll Cardiol. 47, 1769-1776 (2006).
- Gonzalez-Valdes, I., et al. Bmi1 limits dilated cardiomyopathy and heart failure by inhibiting cardiac senescence. Nat Commun. 6, 6473 (2015).
- Kimura, W., et al. Hypoxia fate mapping identifies cycling cardiomyocytes in the adult heart. Nature. 523, 226-230 (2015).
- Soonpaa, M. H., Field, L. J. Assessment of cardiomyocyte DNA synthesis in normal and injured adult mouse hearts. Am J Physiol. 272, H220-H226 (1997).

