Gelijktijdig Beoordeling van Cardiomyocyte DNA Synthese en Ploïdie: een methode om Assist Kwantificering van Cardiomyocyte Regeneration en Omzet
Summary
Quantification of cardiomyocyte turnover is challenging. The protocol described here makes an important contribution to this challenge by enabling accurate and sensitive quantification of neo-cardiomyocyte nuclei generation and nuclei ploidy.
Abstract
Hoewel wordt aangenomen dat het hart heeft een beperkte mogelijkheden om hartspiercellen na letsel en dat lage niveaus van de omzet cardiomyocyt situaties zich voordoet tijdens de normale veroudering te regenereren, kwantificering van deze gebeurtenissen blijft uitdagend. Dit is gedeeltelijk te wijten aan de zeldzaamheid van het proces en het feit dat meerdere cellulaire bronnen dragen bij aan het myocard onderhoud. Bovendien DNA duplicatie in cardiomyocyten leidt vaak tot een polyploïde cardiomyocyten en slechts zelden leidt tot nieuwe hartspiercellen door celdeling. Om nauwkeurig te kwantificeren cardiomyocyt omzet onderscheid tussen deze processen is essentieel. De hier beschreven protocol maakt gebruik nucleoside etikettering lange termijn om alle kernen die zijn ontstaan als gevolg van DNA-replicatie en cardiomyocyten kernen geïdentificeerd door gebruik kernen isolatie en vervolgens PCM1 immunokleuring labelen. Samen toelaat nauwkeurige en gevoelige identificatie van de nucleoside etikettering van de cardiomyocyte kernen bevolking. Verder 4 ', 6-diamidino-2-fenylindol labeling en analyse van kernen ploïdie, maakt de discriminatie van neo-cardiomyocyt kernen van nuclei die in polyploïdisatie nucleoside hebben opgenomen. Hoewel deze methode niet kan controleren voor cardiomyocyt binucleation, is het mogelijk een snelle en robuuste kwantificering van neo-cardiomyociet kernen, terwijl goed voor polyploïdisatie. Deze methode heeft een aantal afgeleide toepassingen zoals het beoordelen van mogelijke therapieën voor regeneratie cardiomyocyten verhogen of onderzoeken van de effecten van hartziekten op cardiomyocyten omzet en ploïdie. Deze techniek is ook geschikt voor de extra benedenstroomse immunohistologische technieken, waardoor kwantificatie van nucleoside incorporatie in alle cardiale celtypen.
Introduction
In de afgelopen jaren is er een opeenstapeling van bewijs tegen de veronderstelling dat het hart is een terminaal gedifferentieerde, post-mitotische orgel 1,2 geweest. Echter, kwantificering van cardiomyocyt omzet en regeneratie blijft uitdagend.
De moeilijkheden bij het nauwkeurig identificeren van zeldzame cardiomyocyt generatie met behulp van standaard immunohistochemische technieken zijn goed gerapporteerd 3. Bovendien, de cellulaire bron van cardiomyocyten generatie blijft onzeker bewijs bijdrage per cardiomyocyt proliferatie en differentiatie van stamcellen 4-6. Daarom is het gebruik van lineage tracing modellen waarbij kennis van de cardiomyocyt voorlopercellen fenotype vereist onmogelijk en kwantificering van proliferatie in één populatie, zoals cardiomyocyten, ongepast is. Verder een cardiomyocyten het potentieel voor endoreplicatie zonder karyokinesis (resulterend in een polyploïde autodiomyocyte) of karyokinesis bij afwezigheid van cytokinese (resulterend in een binucleaire cardiomyocyten) 7,8. De kwantificatie van de omzet cardiomyocyten afhankelijk van het vermogen om onderscheid te maken tussen deze gebeurtenissen en waar neo-cardiomyocyt generatie. Dit creëert unieke complicaties omdat DNA replicatie en expressie van cycline-afhankelijke kinasen in cardiomyocyten niet alleen tonen waar celdeling 9,10.
Te helpen bij de kwantificering van neo-cardiomyocyt generatie, hebben we een gevestigde kernen isolatietechniek en immunologische kenmerken van pericentriolar materiaal 1 (PCM1) voor cardiomyocyten kernen identificatie gecombineerd zoals beschreven door Bergmann et al. 7,11 met nieuwe werkwijzen voor langdurige DNA-etikettering en ploïdie analyse. PCM-1 is een centrosome eiwit dat accumuleert in de kern oppervlak van gedifferentieerde, niet-myocyten fietsen. Eerdere studies hebben aangetoond dat antilichamen tegenPCM-1 specifiek label cardiomyocyten kernen 7,11 en als zodanig PCM1 is gebruikt door een aantal onafhankelijke groepen cardiomyocyten 1,12,13 identificeren. Bovendien hebben we aangetoond dat expressie PCM1 kaarten genetisch gelabeld cardiomyocyten kernen in de TnT-Cre transgene muismodel 14 (aanvullende figuur 1).
De hier beschreven protocol maakt de nauwkeurige en gevoelige identificatie van neo cardiomyocyt nucleatie in het muizenhart, ongeacht de cellulaire oorsprong (Figuur 1A en B), terwijl tegelijkertijd uitzondering nucleoside labeling door polyploïdisatie van de analyse (figuur 1C en D). Hoewel deze methode niet kan worden bediend cardiomyocyten binucleation, is het mogelijk een snelle en gefundeerde kwantificering neo-cardiomyocyt kernen die nodig is voor de kwantificatie van de omzet cardiomyocyt. Voortshet biedt een snelle screening tool om potentiële veranderingen in de dynamiek van cardiomyocyt generatie te beoordelen.
Terwijl DNA labeling omvat gewoonlijk 5-Broom-2'-deoxyuridine (BrdU) als thymidine analoge, de hier beschreven protocol maakt gebruik van een 5-ethynyl-2'-deoxyuridine (EDU) gebaseerde assay zoals minder verwerkingsstappen voor een snellere vereist doorvoercapaciteit en vereist geen denaturering van het DNA voor immunodetectie, waardoor compatibel met andere immunokleuring protocollen en daarmee de mogelijke verdere toepassingen van de werkwijze toeneemt.
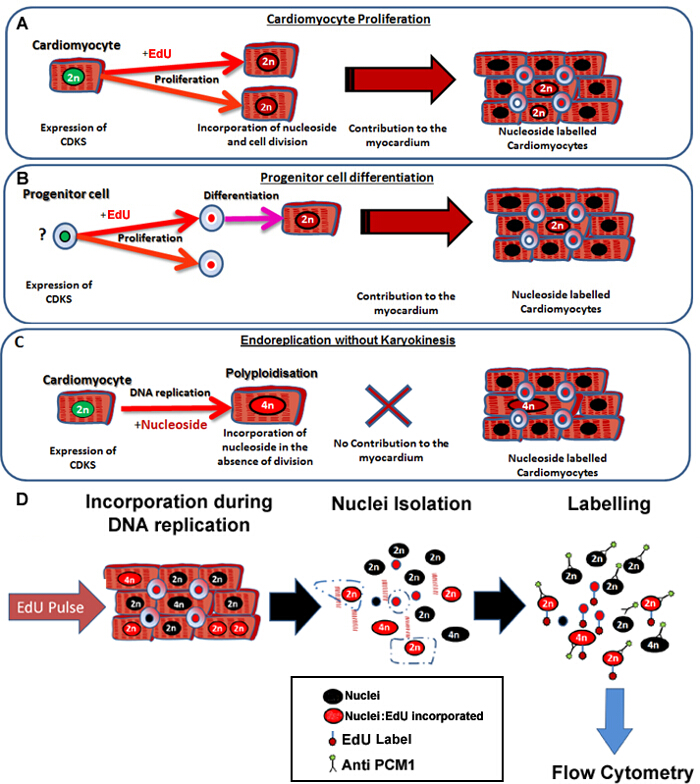
Figuur 1: Continue pulseren met Edu labels neo-hartspiercellen, ongeacht hun voorouders. (A) EDU wordt geïncorporeerd in het DNA van cardiomyocyten tijdens de celdeling. Proliferatie in de cardiomyocyt bevolking zal Result een verhoging van, of vervanging van cardiomyocyten en derhalve productieve DNA synthese (draagt weefsel onderhoud en reparatie). (B) EDU is opgenomen in het DNA van cardiale progenitorcellen tijdens de celdeling. Dit zal in de cel behouden tijdens differentiatie de cardiomyocyt lijn. Deze stamcel differentiatie leidt ook tot een toename van het aantal cardiomyocyten en levert dus weefsel onderhoud en reparatie. (C) Cardiomyocytes het potentieel hebben om "niet-productieve" DNA replicatie resulteert in verhoogde ploïdie cardiomyocyten, die wordt geassocieerd met cardiomyocyt hypertrofie en myocardiale remodeling ondergaan, maar niet verloren cardiomyocyten vervangen. Werkwijze polyploïdisatie verschilt binucleation aangezien zij leidt tot een cardiomyocyt met één kernen die vier of meer sets van twee homologe chromosomen (> 2 N) bevat. (D) Naar aanleiding van een continue kernen pulse Dit protocol beschrijft kernen isolatie en identificatie van de cardiomyocyt kernen door PCM1 uitdrukking kwantificering van zowel cardiomyocyt ploïdie en Edu integratie mogelijk te maken. PCM1 expressie en Edu integratie gedetecteerd met behulp van flowcytometrie. Klik hier om een grotere versie van deze figuur te bekijken.
Protocol
Representative Results
Discussion
Om nauwkeurig te kwantificeren omzet cardiomyocyt en regeneratie assays moeten onderscheid maken tussen echte cardiomyocyt generatie en niet-productieve DNA divisie. Vele studies blijven deze niet-productieve gebeurtenissen gewoon negeren, het kwantificeren van cardiomyocyt proliferatie uitsluitend via de expressie van cycline kinesis en celcyclus markers. Om een enkele methode die de nauwkeurige kwantificering van de omzet cardiomyocyt maakt terwijl de controle voor deze niet-productieve gebeurtenissen zinspelend blijft dateren. In het bijzonder blijft het moeilijk om rekening te cardiomyocyt polyploïdisatie die tot bijdraagt tot ~ 65% cardiomyocyt DNA replicatie 13. Derhalve helpen bij de accurate kwantificering van cardiomyocyten generatie hebben we een protocol dat de gefundeerde kwantificering van de tarieven van neo cardiomyocyt kernen maakt met uitsluiting van DNA-replicatie die leidt tot verhoogde ploïdie ontwikkeld. Hoewel dit protocol kan geen discriminatie tussen de neo-cardiomyociet generatie en cardiomyocyten bi-nucleatie, kan het snel worden gebruikt en nauwkeurig te berekenen de bovengrens (goed voor ploïdie) van cardiomyocyt generatie. Dit protocol geeft daarom een screening tool om mogelijke veranderingen in de tarieven van de cardiomyocyt generatie en polyploïdisatie in ziektemodellen te beoordelen of om het potentieel rendement van therapieën te evalueren. Nadat de wijzigingen in de snelheid van neo-cardiomyocyt nucleatie worden geïdentificeerd met dit protocol latere studies kunnen worden gebruikt om vast te stellen of deze door veranderingen in cardiomyocyten generatie cardiomyocyten nucleatie nummer, zoals hiervoor beschreven 2,13,17,18. Deze omvatten het gebruik van histologische kwantificatie cardiomyocyt nucleatie dynamiek tijdens de pulsperiode of analyses van weefselsecties verkregen van EDU gepulste dieren om de EDU opname vergelijken de mononucleaire en multinucleated cardiomyocyten populaties.
Vanwege de lage cardiomyocyt deze omzetprotocol maakt gebruik van meerdere injecties van Edu over een periode van 7 dagen. Dit maakt ook de "jagen" alle potentiële cellulaire bronnen van cardiomyocyten generatie en maakt kwantificering van cumulatieve cardiomyocyt nucleatie in deze periode. Afhankelijk van de studie, kan deze periode worden aangepast aan de verwachte niveaus van cardiomyocyten generatie passen. Voor de kwantificatie van edu opname in cardiomyocyten kernen, is het noodzakelijk dat er geen niet-specifieke labeling van de kernen met het secundaire antilichaam gebruikt om PCM-1 reactiviteit detecteren. Het zou daarom verstandig zijn om aanvullende secundair antilichaam titratie-experimenten uitvoeren om dit aspect van het protocol optimaliseren, vooral als een secundair antilichaam dan die voorgesteld in dit protocol wordt gebruikt. De hier beschreven protocol gebruikt PCM-1 expressie cardiomyocyten kernen identificeren. Hoewel dit een gevestigd cardiomyocyten marker kunnen alternatieve merkers worden gebruikt om te validerengegevens; deze omvatten antilichamen specifiek voor cardiaal troponine T die is geïdentificeerd als gedeeltelijk gelokaliseerd in cardiomyocyten kernen 1. Evenzo kunnen alternatieve nucleair gelokaliseerde eiwitten worden gebruikt voor het identificeren en kwantificeren EDU verwerking in andere dan de cardiomyocyten kernen populaties. Het is belangrijk dat alle cardiomyocyt kernen die actief mitose ondergaan zijn uitgesloten van de analyse, aangezien het verdere verloop van dit DNA synthese is onbekend en kan ofwel celdeling of verhoogde ploïdie. PCM1 wordt gedemonteerd in de M fase van de celcyclus dan cardiomyocyten ondergaat mitose niet worden opgespoord door PCM1 expressie. Bovendien moeten alle kernen in de S-fase van de celcyclus worden uitgesloten van verdere analyse. Dit kan worden bereikt door gating alle kernen met DAPI intensiteit boven de 2N bevolking waaronder die met DAPI intensiteit tussen 2N en 4N populaties.
Hoewel het steedsaanvaard dat het hart heeft de capaciteit om cardiomyocyten vervangen tijdens normale veroudering en na acuut letsel, de bron en de mate van het mogelijke blijft controversieel. Daarnaast zijn uiteenlopende percentages van de omzet cardiomyocyt gemeld 1,7,20-22. Dit kan voor een deel te wijten aan de moeilijkheden bij het nauwkeurig identificeren en kwantificeren van neo-cardiomyocyt generatie 19. Tot op heden de meeste studies hebben alleen gebaseerd op het gebruik van histologische analyse en identificatie van cardiomyocyten via de expressie van cytoplasmatische eiwitten, waaronder eiwitten van het sarcomeer, voor de kwantificering van cardiomyocyten omzet en vernieuwing 2,4,23,24. Het gebruik van deze werkwijzen om de expressie van proliferatiemerkers detecteren, of zoals hier getoond, kan de opname van thymidine analogen gemakkelijk leiden tot het onterecht overige cardiale celtypen zoals cardiomyocyten. Hoewel het gebruik van 3D confocale beeldvorming kan helpen om deze problemen verlichten these methoden zijn duur en tijdrovend. Interessant is dat de hier beschreven protocol toont neo-cardiomyocyt nucleatie optreedt met een snelheid van 0,17% per week. Dit is consistent met andere flowcytometrie op basis van studies aantonen van de wekelijkse omzet van maximaal 0,13% 5. Hoewel het verleidelijk om jaaromzet tarieven te extrapoleren op basis van deze gegevens, zoals in eerdere studies 2,5,25,26, dit is ongepast als de tarieven van de omzet zijn dynamisch tijdens de levensduur van een dier 13.
Deze methode heeft een aantal potentiële toepassingen, waaronder de beoordeling van de mogelijke therapieën voor de cardiomyocyt regeneratie verbeteren of het onderzoek naar de gevolgen van hart-en vaatziekten op de omzet cardiomyocyten en de tarieven van de cardiomyocyt polyploïdisatie.
Disclosures
The authors have nothing to disclose.
Acknowledgements
This work was supported by the British Heart Foundation, project grant PG/13/69/30454.
Materials
| 0.32 M sucrose | Sigma | 84100 | |
| 10 mM Tris-HCl (pH = 8) | Sigma | T3253 | |
| 5 mM CaCl2 | Sigma | c5086 | |
| 5 mM magnesium acetate | sigma | M-5661 | |
| 2.0 mM EDTA | Sigma | E5134 | |
| 0.5 mM EGTA | Sigma | 63779 | |
| 1 mM DTT | Sigma | D0632 | |
| 70 mM KCl | Sigma | P9541 | |
| 10 mM MgCl2 | Sigma | M8266 | |
| 1.5 mM spermine | Sigma | 85590 | |
| Isotype rabbit IgG- ChIP Grade | abcam | abc7415 | |
| Rabbit anti-PCM-1 antibody | Sigma | HPA023374 | |
| Alexa Fluor 488 F(ab')2 Fragment of Goat Anti-Rabbit IgG (H+L) Antibody | Life technologies | A-11070 | |
| cell strainers 70 μm and 100μm | Fisher scientific | 11597522, 11517532 | |
| Glass dounce (40 ml) and pestle large clearance | Sigma | D9188-1SET | |
| EdU (5-ethynyl-2’-deoxyuridine) | Life technologies | A10044 | |
| Click-iT Plus EdU Alexa Fluor 647 Flow Cytometry Assay Kit | Life technologies | C10634 | This kit inlcudes reagents required for section, EdU reaction buffer, EdU fixative, saponin-based permeabilization solution and the reagents required for the EdU labelling cocktail. |
| CyStain DNA 2 step kit, | Sysmex Partec | 05 5005 | This kit inlcudes reagents required for DAPI labelling (DNA staining solution) |
| Probe homogeniser e.g. TissueRuptor | Qiagen | 9001273 | |
| TissueRuptor Disposable Probes | Qiagen | 990890 | |
| ultracentrifuge | Sorvall | ||
| Facscanto II | BD Biosciences | ||
| Ultracentrifuge Tube, Thinwall, Polypropylene. 38.5 mL, 25 x 89 mm | Beckman Coulter | 326823 | |
| Bovine serum albumin | Sigma | A2153 |
References
- Bergmann, O., et al. Evidence for cardiomyocyte renewal in humans. Science. 324, 98-102 (2009).
- Senyo, S. E., et al. Mammalian heart renewal by pre-existing cardiomyocytes. Nature. 493, 433-436 (2013).
- Soonpaa, M. H., Rubart, M., Field, L. J. Challenges measuring cardiomyocyte renewal. Biochim Biophys Acta. 1833, 799-803 (2013).
- Loffredo, F. S., Steinhauser, M. L., Gannon, J., Lee, R. T. Bone marrow-derived cell therapy stimulates endogenous cardiomyocyte progenitors and promotes cardiac repair. Cell Stem Cell. 8, 389-398 (2011).
- Malliaras, K., et al. Cardiomyocyte proliferation and progenitor cell recruitment underlie therapeutic regeneration after myocardial infarction in the adult mouse heart. EMBO Mol Med. 5, 191-209 (2013).
- Hsieh, P. C., et al. Evidence from a genetic fate-mapping study that stem cells refresh adult mammalian cardiomyocytes after injury. Nat Med. 13, 970-974 (2007).
- Bergmann, O., et al. Identification of cardiomyocyte nuclei and assessment of ploidy for the analysis of cell turnover. Exp Cell Res. 317, 188-194 (2011).
- Herget, G. W., Neuburger, M., Plagwitz, R., Adler, C. P. DNA content, ploidy level and number of nuclei in the human heart after myocardial infarction. Cardiovasc Res. 36, 45-51 (1997).
- Scholzen, T., Gerdes, J. The Ki-67 protein: from the known and the unknown. J Cell Physiol. 182, 311-322 (2000).
- Carmena, M., Earnshaw, W. C. The cellular geography of aurora kinases. Nat Rev Mol Cell Biol. 4, 842-854 (2003).
- Bergmann, O., Jovinge, S. Isolation of Cardiomyocyte Nuclei from Post-mortem Tissue. J. Vis. Exp. , e4205 (2012).
- Gilsbach, R., et al. Dynamic DNA methylation orchestrates cardiomyocyte development, maturation and disease. Nat Commun. 5, 5288 (2014).
- Richardson, G., Laval, S., Owens, W. A. Cardiomyocyte regeneration in the mdx mouse model of non-ischemic cardiomyopathy. Stem Cells Dev. , (2015).
- Jiao, K., et al. An essential role of Bmp4 in the atrioventricular septation of the mouse heart. Genes Dev. 17, 2362-2367 (2003).
- Bergmann, O., et al. Cardiomyocyte renewal in humans. Circ Res. 110, e17-e18 (2012).
- Prigge, J. R., et al. Nuclear double-fluorescent reporter for in vivo and ex vivo analyses of biological transitions in mouse nuclei. Mamm Genome. , (2013).
- Naqvi, N., et al. A proliferative burst during preadolescence establishes the final cardiomyocyte number. Cell. 157, 795-807 (2014).
- Liu, Z., Yue, S., Chen, X., Kubin, T., Braun, T. Regulation of cardiomyocyte polyploidy and multinucleation by CyclinG1. Circ Res. 106, 1498-1506 (2010).
- Ang, K. L., et al. Limitations of conventional approaches to identify myocyte nuclei in histologic sections of the heart. Am J Physiol Cell Physiol. 298, C1603-C1609 (2010).
- Kajstura, J., et al. Myocyte turnover in the aging human heart. Circ Res. 107, 1374-1386 (2010).
- Kajstura, J., et al. Cardiomyogenesis in the adult human heart. Circ Res. 107, 305-315 (2010).
- Walsh, S., Ponten, A., Fleischmann, B. K., Jovinge, S. Cardiomyocyte cell cycle control and growth estimation in vivo–an analysis based on cardiomyocyte nuclei. Cardiovasc Res. 86, 365-373 (2010).
- Anversa, P., Leri, A., Kajstura, J. Cardiac regeneration. J Am Coll Cardiol. 47, 1769-1776 (2006).
- Gonzalez-Valdes, I., et al. Bmi1 limits dilated cardiomyopathy and heart failure by inhibiting cardiac senescence. Nat Commun. 6, 6473 (2015).
- Kimura, W., et al. Hypoxia fate mapping identifies cycling cardiomyocytes in the adult heart. Nature. 523, 226-230 (2015).
- Soonpaa, M. H., Field, L. J. Assessment of cardiomyocyte DNA synthesis in normal and injured adult mouse hearts. Am J Physiol. 272, H220-H226 (1997).

