Caractérisation des protéines par chromatographie d'exclusion couplée à multi-angle Light Scattering (SEC-MALS)
Summary
Ce protocole décrit la combinaison de la chromatographie d’exclusion de taille avec la diffusion de lumière multi-angle (SEC-MALS) pour la caractérisation absolue des protéines et des complexes dans la solution. SEC-MALS détermine le poids moléculaire et la taille des protéines pures, des oligomères indigènes, des hétérocomplexes et des protéines modifiées telles que les glycoprotéines.
Abstract
La chromatographie analytique d’exclusion de taille (SEC), couramment utilisée pour déterminer le poids moléculaire des protéines et des complexes protéines-protéines en solution, est une technique relative qui repose sur le volume d’élution de l’analyte pour estimer les poids. Lorsque la protéine n’est pas globulaire ou subit des interactions de colonne non idéales, la courbe d’étalonnage basée sur les normes de protéines est invalide, et le poids moléculaire déterminé à partir du volume d’élution est incorrect. La diffusion de la lumière multi-angle (MALS) est une technique absolue qui détermine le poids moléculaire d’un analyte dans la solution à partir d’équations physiques de base. La combinaison de SEC pour la séparation avec MALS pour l’analyse constitue un moyen polyvalent et fiable pour caractériser les solutions d’une ou plusieurs espèces protéiques, y compris les monomères, oligomères indigènes ou agrégats, et les hétérocomplexes. Étant donné que la mesure est effectuée à chaque volume d’élution, SEC-MALS peut déterminer si un pic d’élution est homogène ou hétérogène et faire la distinction entre une répartition du poids moléculaire fixe par rapport à l’équilibre dynamique. L’analyse de protéines modifiées telles que les glycoprotéines ou les lipoprotéines, ou des conjugués tels que les protéines membranaires détergent-solubilisées, est également possible. Par conséquent, SEC-MALS est un outil essentiel pour le chimiste de protéine qui doit confirmer les propriétés biophysiques et le comportement de solution des molécules produites pour la recherche biologique ou biotechnologique. Ce protocole pour SEC-MALS analyse le poids moléculaire et la taille des monomères et des agrégats de protéines pures. Les données acquises servent de base à d’autres analyses SEC-MALS, y compris celles de complexes, de glycoprotéines et de protéines membranaires liées aux surfactants.
Introduction
Une analyse fiable du poids moléculaire (MW) des protéines en solution est essentielle pour la recherche biomoléculaire1,2,3,4. L’analyse de MW informe le scientifique si la protéine correcte a été produite et si elle est appropriée pour l’usage dans l’expérimentation plus loin5,6. Comme décrit sur les sites Web des réseaux de recherche sur les protéines P4EU7 et ARBRE-Mobieu8, le contrôle de la qualité des protéines doit caractériser non seulement la pureté du produit final, mais aussi son état oligomérique, son homogénéité, son identité, sa conformation, structure, les modifications post-traduction et d’autres propriétés.
La mesure MW dans la solution non dénaturante identifie la forme de la protéine qui est présente dans un environnement aqueux, qu’il soit monomérique ou oligomérique. Alors que pour de nombreuses protéines l’objectif est de produire la forme monomérique, pour d’autres un oligomère indigène spécifique est la clé de l’activité biologique9,10,11,12. D’autres oligomères et agrégats non indigènes ne sont pas souhaitables et entraîneront des défauts dans la détermination structurelle par la cristallographie, la résonance magnétique nucléaire (RMN) ou la diffusion de rayons X à petit angle, ainsi que des artefacts ou des inexactitudes dans les analyses fonctionnelles quantifier la liaison par calorimétrie de titration isothermique ou résonance plasmon de surface2,13.
Dans le cas des biothérapeutiques tels que les anticorps monoclonaux (mAbs), l’analyse MW basée sur des solutions sert un but similaire de contrôle de la qualité et de caractérisation du produit. Les agrégats et les fragments excessifs sont indicatifs d’un produit instable qui n’est pas adapté à une utilisation humaine. Les organismes de réglementation exigent une caractérisation minutieuse, non seulement de la molécule thérapeutique, mais aussi des dégradants potentiels qui peuvent être présents dans le produit final14,15,16,17.
Certaines des méthodes les plus répandues pour analyser les protéines MW sont l’électrophores de gel de sulfate de sulfate de sodium polyacrylamide (SDS-PAGE), l’électrophorèse capillaire (CE), la PAGE indigène, la spectrométrie de masse (MS), la chromatographie d’exclusion de taille (SEC) et l’analyse l’ultracentrifugation (AUC). Parmi ceux-ci, SDS-PAGE, CE et MS ne sont pas exécutés dans l’état indigène et conduisent généralement à la dissociation des oligomères et des agrégats, donc ne sont pas appropriés pour déterminer l’oligomère indigène ou quantifier les agrégats. Bien que page indigène ne, théoriquement, conserver l’état natif, dans notre expérience, il est difficile d’optimiser pour de nombreuses protéines, et les résultats ne sont pas très fiables. L’AUC, que ce soit par vitesse de sédimentation ou équilibre de sédimentation, est quantitative et peut déterminer MW à partir des premiers principes, mais il est assez lourd, nécessitant beaucoup de travail manuel et une expertise significative dans l’interprétation des données, le temps d’expérience long et un instrument très coûteux.
La SEC analytique est une méthode quantitative et relativement robuste et simple qui sépare les macromolécules pendant le flux à travers une colonne emballée. Les principes et les applications de la SEC sont bien présentés dans plusieurs revues18,19,20 et dans le manuel “Size Exclusion Chromatography: Principles and Methods”21. Les différences de rétention sont dues à différentes quantités de temps passé à se répandre dans et hors des pores dans la phase stationnaire avant d’élavoir de la fin de la colonne. Les différences proviennent (nominalement) des tailles relatives et des coefficients de diffusion des molécules22. Une courbe d’étalonnage est construite à l’aide d’une série de molécules de référence, reliant le MW de la molécule au volume d’élution. Pour les protéines, les molécules de référence sont généralement des protéines globulaires bien élevées qui n’interagissent pas avec la colonne par charge ou résidus de surface hydrophobes. Le volume d’élution est mesuré à l’aide d’un détecteur d’absorption ultraviolet (UV). Si le coefficient d’extinction UV est connu- souvent calculé à partir de la séquence, la masse totale maximale de protéines peut également être quantite.
Notamment, l’analyse de MW par SEC repose sur deux hypothèses clés concernant les protéines à caractériser : 1) elles partagent avec les normes de référence la même conformation et le même volume spécifique (en d’autres termes, la même relation entre les propriétés de diffusion et MW) et 2) comme les normes de référence, ils n’interagissent pas avec la colonne, sauf par des propriétés stériques, ils n’adhèrent pas à l’emballage de la colonne par charge ou interactions hydrophobes. Les déviations par rapport à ces hypothèses invalident la courbe d’étalonnage et conduisent à des déterminations erronées du MW. C’est le cas pour les protéines intrinsèquement désordonnées qui ont de grands radii de Stokes en raison de leurs régions non structurées étendues23,24 ou non sphériques/linéaires assemblages oligomeric10. Les protéines glycosylated auront généralement un plus grand rayon de Stokes que la forme non-glycosylated, même quand la masse ajoutée de hydrate de carbone est prise en compte19. Les protéines membranaires stétrifiantes s’élifient différemment des protéines d’étalonnage parce que leur suppression de sec dépend de la taille totale du complexe polypeptide-détergent-lipides plutôt que de l’état oligomeric et de la masse molaire de la protéine25 ,26. La chimie des colonnes, le pH et les conditions de sel affectent tous les volumes d’élution des protéines avec des résidus de surface chargés ou hydrophobes27,28.
SEC devient beaucoup plus polyvalent et fiable pour la détermination MW lorsqu’il est combiné avec la diffusion de lumière multi-angle (MALS) et différentiel réfractive index (dRI) détecteurs3,4,11,29, 30,31,32. Un détecteur d’IRD détermine la concentration en fonction de la variation de l’indice de réfraction de la solution en raison de la présence de l’analyte. Un détecteur MALS mesure la proportion de lumière dispersée par un analyte en plusieurs angles par rapport au faisceau laser incident. Collectivement connu sous le nom SEC-MALS, cet instrumentation détermine MW indépendamment du temps d’élution puisque MW peut être calculé directement à partir des premiers principes en utilisant l’équation 1,
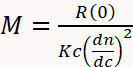 ①
①
où M est le poids moléculaire de l’analyte, R(0) le rapport réduit de Rayleigh (c.-à-d., la quantité de lumière dispersée par l’analyte par rapport à l’intensité laser) déterminée par le détecteur MALS et extrapolée à l’angle zéro, c concentration de poids déterminée par le détecteur UV ou dRI, dn/dc l’incrément de l’indice de réfraction de l’analyte (essentiellement la différence entre l’indice de réfraction de l’analyte et le tampon), et K une constante optique qui dépend de la propriétés du système telles que la longueur d’onde et l’indice de réfraction des solvants29.
Dans SEC-MALS, la colonne SEC est utilisée uniquement pour séparer les différentes espèces en solution afin qu’elles pénètrent individuellement dans les cellules du MALS et du détecteur de concentration. Le temps de rétention réel n’a aucune signification pour l’analyse, sauf dans la mesure où il résout les espèces protéiques. Les instruments sont calibrés indépendamment de la colonne et ne reposent pas sur des normes de référence. Par conséquent, SEC-MALS est considéré comme une méthode «absolue» pour la détermination MW à partir d’équations physiques de base. Si l’échantillon est hétérogène et non complètement séparé par la colonne, alors la valeur fournie à chaque volume d’élution sera une moyenne de poids des molécules dans chaque volume d’élution qui circule à travers la cellule d’écoulement par tranche de temps, environ 75 L.
En anadisant la variation angulaire de l’intensité de diffusion, MALS peut également déterminer la taille (rayon carré racine-moyen, Rg) des macromolécules et des nanoparticules avec un rayon géométrique supérieur à environ 12,5 nm29. Pour les espèces plus petites comme les protéines monomériques et les oligomères, un module dynamique de diffusion de la lumière (DLS) peut être ajouté à l’instrument MALS afin de mesurer les radii hydrodynamiques à partir de 0,5 nm et jusqu’à33.
Bien que l’analyse de la concentration des UV ou de l’IRD puisse fournir la valeur de c dans l’Eq. 1, l’utilisation de l’IRD est préférable pour deux raisons : 1) dRI est un détecteur de concentration universel, adapté à l’analyse de molécules telles que les sucres ou les polysaccharides qui ne contiennent pas d’UV chromophore34; et 2) la réponse de concentration dn/dc de presque toutes les protéines pures dans le tampon aqueux est la même à dans un ou deux pour cent (0.185 mL/g)35,ainsi il n’est pas nécessaire de connaître le coefficient d’extinction UV.
L’utilisation de SEC-MALS dans la recherche sur les protéines est assez étendue. De loin les applications les plus courantes sont d’établir si une protéine purifiée est monomeric ou oligomeric et le degré d’oligomerisation, et l’évaluation des agrégats3,10,11,17, 31,36,37,38. La capacité de le faire pour les protéines membranaires détergent-solubilisées qui ne peuvent pas être caractérisées par des moyens traditionnels est particulièrement prisée, et des protocoles détaillés pour ceci ont été édités31,39,40 , 41 Ans, états-unis ( , 42 Ans, états-unis ( , 43. D’autres applications courantes comprennent l’établissement du degré de modification post-traduction nacrée et de polydispersity de la glycoprotéine, des lipoprotéines et des conjugues similaires4,31,44 , 45 Annonces , 46 Annonces , 47; la formation (ou l’absence de celle-ci) et la stoichiométrie absolue (par opposition au rapport stoichiométrique) des hétérocomplexes, y compris les protéines-protéines, l’acide nucléique-protéine et les complexes protéique-polysaccharide24,46, 48,49,50,51,52; déterminer la dissociation d’équilibre monomère-dimer constante49,53,54; et l’évaluation de la conformation des protéines55,56. Au-delà des protéines, SEC-MALS est inestimable pour la caractérisation des peptides57,58, polymères naturels largement hétérogènes tels que les héparines59 et chitosans60,61, petits virus62 et la plupart des types de polymères synthétiques ou transformés63,64,65,66. Une bibliographie étendue peut être trouvée dans la littérature67 et en ligne (à http://www.wyatt.com/bibliography).
Ici, nous présentons un protocole standard pour l’exécution et l’analyse d’une expérience SEC-MALS. L’albumine bovine de sérum (BSA) est présentée comme exemple pour la séparation et la caractérisation des monomères et des oligomères de protéine. Le protocole BSA détermine certaines constantes du système qui servent de base à d’autres analyses SEC-MALS, y compris celles des complexes, des glycoprotéines et des protéines membranaires liées aux surfactants.
Nous notons que SEC-MALS peut être effectué en utilisant la chromatographie liquide haute performance standard (HPLC) ou l’équipement de chromatographie liquide à protéines rapides (FPLC) de nombreux fournisseurs. Ce protocole décrit l’utilisation d’un système FPLC que l’on trouve couramment dans les laboratoires qui produisent des protéines pour la recherche et le développement (voir Tableau des matériaux). Avant d’exécuter le protocole, le système FPLC, les détecteurs MALS et dRI auraient dû être installés, ainsi que leurs progiciels respectifs pour le contrôle, l’acquisition et l’analyse de données selon les instructions des fabricants et les constantes d’étalonnage requises. ou d’autres paramètres entrés dans le logiciel. Un filtre en ligne doit être placé entre la pompe et l’injecteur avec une membrane hydrophile de 0,1 m pore installée.
Protocol
Representative Results
Discussion
L’expérience SEC-MALS a fourni une bonne séparation du monomère, du dimère et du trimère, ainsi que des résultats quantitatifs pour les masses molaires et les tailles hydrodynamiques de chaque pic. Cela identifie et caractérise clairement chaque espèce présente, ainsi que la quantification de la pureté. Habituellement, les résultats obtenus sont précis à moins de 5%, et précis et répétable à moins de 1-2%3,69. Ce niveau de précision et de répétabilité permet de distinguer en toute confiance les espèces qui peuvent être proches en MW, tant qu’elles sont séparées par la SEC (peut se chevaucher partiellement dans le même pic). Les avantages pour le contrôle de la qualité des protéines et la caractérisation biophysique fondamentale sont évidents.
La vérification de l’absence de particules est très importante pour les mesures sensibles et répétables SEC-MALS. Les pics de particules apparaissent généralement sous forme de signaux MALS importants non accompagnés de signaux UV ou RI comparables. La phase mobile et l’échantillon doivent être préparés avec soin pour éliminer ces particules. Utilisation de réactifs de qualité HPLC ou mieux, filtration de la phase mobile et tampon de dilution à 0,1 – 0,2 m (pré-lavage du filtre pour éliminer les particules qui sont toujours présents sur les membranes sèches), maintien de bouteilles de phase mobile extra-propre et autres verreries pour SEC-MALS et l’équilibrage prolongé de colonne sous courant (pour enlever les particules et les agrégats qui peuvent s’être accumulés lorsque le débit a été arrêté ou une colonne non utilisée) sont tous recommandés. L’échantillon doit être filtré à la plus petite taille de pores qui n’enlève pas le matériau d’intérêt, généralement pas plus grand que 0,1 m et, si possible, 0,02 m. Si le filtre obstrue rapidement, l’échantillon peut être centrifugé, filtré à des stades de taille descendante des pores et/ou repurifié. Lorsque les systèmes sont constamment maintenus avec une phase mobile de haute qualité, fraîche et filtrée, que les chocs de colonne sont évités, que les échantillons sont propres et qu’ils n’adhèrent pas à la colonne, les mesures n’exhiberont pas le bruit de particules susmentionné et fourniront données de haute qualité.
MALS est agnostique aux tampons typiques de SEC pour des protéines ; beaucoup d’autres systèmes tampons en plus de PBS peuvent être utilisés pour optimiser la séparation et la stabilité, y compris une variété d’excipients71. MALS est également agnostique à la colonne spécifique, qui doit être sélectionnée pour une séparation et une récupération optimales. Les principales préoccupations des excipients en ce qui concerne l’analyse sont des changements significatifs dans l’indice de réfraction, conduisant à la modification de l’indice réfractif spécifique dn/dc, et les excipients qui absorbent 280 nm lorsque l’analyse UV est nécessaire. Par exemple, l’arginine est une excipiente commune de réduction de l’agrégation qui peut affecter considérablement le dn/dc d’une protéine typique, même en l’amenant dans le régime négatif (une protéine avec dn/dc négatif peut encore être analysée par SEC-MALS si dn/ dc est déterminé empiriquement, mais si dn/dc 0 l’intensité de la lumière dispersée par la protéine sera nulle et l’analyse DE MW sera impossible). Le sujet des valeurs dn/dc pour les protéines est longuement discuté par Zhao et al.35 où il est démontré que pour les tampons aqueux standard, la grande majorité des protéines non modifiées se situent dans 2-3% de la valeur standard (0,185 ou 0,186 mL/g à 660 nm) , bien que les protéines inférieures à 10 kDa soient plus variables, et il y a quelques espèces qui peuvent aller jusqu’à 0,21 ml/g.
Les profils MW à travers les pics de monomère et de dimère de BSA dans les données présentées étaient tous deux assez homogènes à moins de 2% ou moins, indiquant des espèces monodispersées. Les valeurs MW non uniformes d’un sommet peuvent résulter d’une hétérogénéité ou d’une analyse incorrecte. En particulier, un profil BSA MW qui est concave («sourire») ou convexe («grimace») pourrait résulter de ne pas appliquer correctement la correction de bande-élargissement. Pour d’autres protéines, un profil convexe pourrait également résulter de l’équilibre dynamique entre les monomères et les oligomères, où le rapport de monomère à oligomère – et donc la valeur apparente de MW – dépend de la concentration maximale (BSA ne présente pas ce comportement et est souvent utilisé comme échantillon témoin pour vérifier les paramètres d’élargissement corrects de la bande). Un profil MW qui varie de la tête au bord de fuite et qui ne change pas avec la concentration de l’échantillon est typique d’une répartition des espèces de poids moléculaire qui sont partiellement résolues par la SEC. L’équilibre dynamique se distingue facilement de l’autre les sources de distributions apparemment inhomogènes en injectant différentes quantités totales de protéines -la distribution variera avec l’équilibre dynamique, mais pas avec une distribution fixe ou une correction incorrecte de l’élargissement de la bande.
Compte tenu des contraintes décrites ci-dessus, SEC-MALS n’est pas adapté à diverses tâches. Il ne convient pas à l’analyse d’échantillons bruts; les échantillons doivent être bien purifiés par des méthodes standard d’affinité et de polissage. Il n’a pas suffisamment de précision ou de puissance de résolution pour identifier les mutants et les variantes d’une protéine ou d’un mAb avec la même masse ou très proche, et ne peut pas être utilisé avec des analytes qui ne s’échappent pas ou ne se séparent pas sur une colonne SEC, bien que récemment il a été démontré que ion-exchang e ou la chromatographie en phase inverse peut être combinée avec MALS pour séparer et caractériser les espèces qui ne sont pas résolues par SEC72,73,74. Lorsque les quantités de protéines sont sévèrement limitées, SEC-MALS peut ne pas être faisable car il nécessite généralement 10 – 200 g3 et même plus peut être nécessaire par un système FPLC avec un diamètre intérieur de tuyauterie supérieur à 0,25 mm; cependant, de plus petites quantités peuvent être analysées par UHPLC-SEC-MALS. Les protéines très instables qui s’agrégent lors de l’introduction à la phase mobile ne conviennent pas à l’analyse SEC-MALS, bien que l’optimisation tampon utilisant la diffusion de lumière dynamique hors ligne puisse surmonter ce problème75.
Malgré les efforts supplémentaires que la SEC-MALS implique, il est inestimable pour la recherche sur les protéines et est largement utilisé par les communautés universitaires et biopharmaceutiques. En plus de la caractérisation des monomères, des oligomères et des agrégats tel que décrit dans le protocole ci-dessus, SEC-MALS peut caractériser des protéines modifiées telles que les glycoprotéines (déterminer le MW des composants protéiques et glycanes individuellement), les surfactants ou protéines membranaires sorubilisées (déterminant le MW des composants des protéines et des composants de solubiliseur individuellement), assemblages de protéines tels que particules virales, protéines-protéines et complexes d’acide nucléique protéique, polysaccharides, conjuguées de protéines-polysaccharide, peptides et bien d’autres biomacromolécules.
Disclosures
The authors have nothing to disclose.
Acknowledgements
Nous remercions le Dr Tsafi Danieli (Wolfson Centre for Applied Structural Biology, Hebrew University) pour ses conseils et ses collaborations. Nous remercions également Daniel Biotech Ltd. (Rehovot, Israel) pour l’aide et la mise en place du système analytique FPLC-MALS utilisé dans cette étude.
Materials
| AKTA pure | GE Healthcare | 29-0182-26 | fast protein liquid chromatograph (FPLC) |
| AKTA UNICORN | GE Healthcare | FPLC control software | |
| ASTRA | Wyatt Technology | MALS data acquisition and analysis software | |
| Bovine serum albumine (purity >97%) | Sigma | A1900 | analyte |
| DAWN or miniDAWN | Wyatt Technology | WH2 or WTREOS | multi-angle light scattering (MALS) detector |
| Increase 200 10/300 | GE Healthcare | size exclusion column, 200 Å pores, 10 mm i.d., 300 mm length | |
| Optilab | Wyatt Technology | WTREX | differential refractive index (RI) detector |
| Sodium chloride NaCl | Sigma | 71382 | HPLC grade NaCl |
| Stericup bottle top filter polyether sulfone 0.1 µm 1000 mL | Millipore | SCVPU11RE | mobile phase filter |
| Whatman Anotop 10 syringe filter 0.02 µm | GE Healthcare | 6809-1002 | sample filter |
| Whatman Anotop 10 syringe-tip filter, 0.1 µm pore, 10 mm diameter | GE Healthcare | 6809-1012 | sample filter |
| Whatman Anotop 25 syringe filter 0.1 µm | GE Healthcare | 6809-2012 | mobile phase filter |
| WyattQELS | Wyatt Technology | WIQ | dynamic light scattering detector |
References
- Acton, T. B., et al. Robotic cloning and Protein Production Platform of the Northeast Structural Genomics Consortium. Methods in Enzymology. 394, 210-243 (2005).
- Acton, T. B., et al. Preparation of protein samples for NMR structure, function, and small-molecule screening studies. Methods in Enzymology. 493, 21-60 (2011).
- Folta-Stogniew, E., Williams, K. R. Determination of molecular masses of proteins in solution: Implementation of an HPLC size exclusion chromatography and laser light scattering service in a core laboratory. Journal of Biomolecular Technology. 10 (2), 51-63 (1999).
- Kendrick, B. S., Kerwin, B. A., Chang, B. S., Philo, J. S. Online size-exclusion high-performance liquid chromatography light scattering and differential refractometry methods to determine degree of polymer conjugation to proteins and protein-protein or protein-ligand association states. Analytical Biochemistry. 299 (2), 136-146 (2001).
- Hughes, C. S., Longo, E., Phillips-Jones, M. K., Hussain, R. Quality control and biophysical characterisation data of. Data Brief. 14, 41-47 (2017).
- Muthurajan, U., et al. In Vitro Chromatin Assembly: Strategies and Quality Control. Methods in Enzymology. 573, 3-41 (2016).
- P4EU. . Protein Quality Standard PQS. , (2019).
- Arbre Mobieu. . Guidelines on Protein Quality Control. , (2019).
- Bowman, A., et al. The histone chaperones Vps75 and Nap1 form ring-like, tetrameric structures in solution. Nucleic Acids Research. 42 (9), 6038-6051 (2014).
- Bowman, G. R., et al. Oligomerization and higher-order assembly contribute to sub-cellular localization of a bacterial scaffold. Molecular Microbiology. 90 (4), 776-795 (2013).
- Folta-Stogniew, E. Oligomeric states of proteins determined by size-exclusion chromatography coupled with light scattering, absorbance, and refractive index detectors. Methods in Molecular Biology. 328, 97-112 (2006).
- Vieux, E. F., Wohlever, M. L., Chen, J. Z., Sauer, R. T., Baker, T. A. Distinct quaternary structures of the AAA+ Lon protease control substrate degradation. Proceedings of the National Academy of Sciences of the United States of America. 110 (22), E2002-E2008 (2013).
- Group, N.S.. . use of SEC-MALS in crystallization QC. , (2018).
- Ahrer, K., Buchacher, A., Iberer, G., Josic, D., Jungbauer, A. Analysis of aggregates of human immunoglobulin G using size-exclusion chromatography, static and dynamic light scattering. Journal of Chromatography A. 1009 (1-2), 89-96 (2003).
- Narhi, L. O., Schmit, J., Bechtold-Peters, K., Sharma, D. Classification of protein aggregates. Journal of Pharmaceutical Sciences. 101 (2), 493-498 (2012).
- Spiess, C., et al. Bispecific antibodies with natural architecture produced by co-culture of bacteria expressing two distinct half-antibodies. Nature Biotechnology. 31 (8), 753-758 (2013).
- Philo, J. S. A critical review of methods for size characterization of non-particulate protein aggregates. Current Pharmaceutical Biotechnology. 10 (4), 359-372 (2009).
- Stellwagen, E. Gel filtration. Methods in Enzymology. 463, 373-385 (2009).
- Burgess, R. R. A brief practical review of size exclusion chromatography: Rules of thumb, limitations, and troubleshooting. Protein Expression and Purification. 150, 81-85 (2018).
- Striegel, A. M., Yau, W. W., Kirkland, J. J., Bly, D. D. . Modern Size-Exclusion Liquid Chromatography. , (2009).
- GE Healthcare. . Size Exclusion Chromatography: Principles and Methods. , (2014).
- Uliyanchenko, E. Size-exclusion chromatography-from high-performance to ultra-performance. Analytical and Bioanalytical Chemistry. 406 (25), 6087-6094 (2014).
- Dunker, A. K., Silman, I., Uversky, V. N., Sussman, J. L. Function and structure of inherently disordered proteins. Current Opinion in Structural Biology. 18 (6), 756-764 (2008).
- Hsiao, H. H., Nath, A., Lin, C. Y., Folta-Stogniew, E. J., Rhoades, E., Braddock, D. T. Quantitative characterization of the interactions among c-myc transcriptional regulators FUSE, FBP, and FIR. Biochemistry. 49 (22), 4620-4634 (2010).
- Hayashi, Y., Takagi, T., Maezawa, S., Matsui, H. Molecular weights of alpha beta-protomeric and oligomeric units of soluble (Na+, K+)-ATPase determined by low-angle laser light scattering after high-performance gel chromatography. Biochimica et Biophysica Acta. 748 (2), 153-167 (1983).
- Folta-Stogniew, E. J. Macromolecular Interactions: Light Scattering. Encyclopedia of Life Sciences. , (2009).
- Hong, P., Koza, S., Bouvier, E. S. P. Size-Exclusion Chromatography for the Analysis of Protein Biotherapeutics and their Aggregates. Journal of Liquid Chromatography and Related Technology. 35 (20), 2923-2950 (2012).
- Chakrabarti, A. Separation of Monoclonal Antibodies by Analytical Size Exclusion Chromatography. Antibody Engineering. , (2018).
- Wyatt, P. J. Light scattering and the absolute characterization of macromolecules. Analytica Chimica Acta. 272, (1993).
- Takagi, T. Application of low-angle laser light scattering detection in the field of biochemistry: review of recent progress. Journal of Chromatography A. 506, 51-63 (1990).
- Wen, J., Arakawa, T., Philo, J. S. Size-exclusion chromatography with on-line light-scattering, absorbance, and refractive index detectors for studying proteins and their interactions. Analytical Biochemistry. 240 (2), 155-166 (1996).
- Mogridge, J. Using light scattering to determine the stoichiometry of protein complexes. Methods in Molecular Biology. 1278, 233-238 (2015).
- Larkin, M., Wyatt, P. J. Light-Scattering Techniques and their Application to Formulation and Aggregation Concerns. Formulation and Process Development Strategies for Manufacturing Biopharmaceuticals. , (2010).
- Wolfender, J. L. HPLC in Natural Product Analysis: The Detection Issue. Planta Medica. 75 (07), 719-734 (2009).
- Zhao, H., Brown, P. H., Schuck, P. On the distribution of protein refractive index increments. Biophysical Journal. 100, (2011).
- Serebryany, E., Folta-Stogniew, E., Liu, J., Yan, E. C. Homodimerization enhances both sensitivity and dynamic range of the ligand-binding domain of type 1 metabotropic glutamate receptor. FEBS Letters. 590 (23), 4308-4317 (2016).
- Arakawa, T., Wen, J. Size-exclusion chromatography with on-line light scattering. Current Protocols in Protein Science. Chapter 20, Unit 20.6 (2001).
- Müller, R., et al. High-resolution structures of the IgM Fc domains reveal principles of its hexamer formation. Proceedings of the National Academy of Sciences. 110 (25), 10183-10188 (2013).
- Slotboom, D. J., Duurkens, R. H., Olieman, K., Erkens, G. B. Static light scattering to characterize membrane proteins in detergent solution. Methods. 46 (2), 73-82 (2008).
- Miercke, L. J., Robbins, R. A., Stroud, R. M. Tetra detector analysis of membrane proteins. Current Protocols in Protein Science. 77, 1-30 (2014).
- Gimpl, K., Klement, J., Keller, S. Characterising protein/detergent complexes by triple-detection size-exclusion chromatography. Biological Procedures Online. 18 (1), 4 (2016).
- Korepanova, A., Matayoshi, E. D. HPLC-SEC characterization of membrane protein-detergent complexes. Current Protocols in Protein Science. Chapter 29, 1-12 (2012).
- Roy, A., Breyton, C., Ebel, C. Analytical Ultracentrifugation and Size-Exclusion Chromatography Coupled with Light Scattering for Characterization of Membrane Proteins in Solution. Membrane Proteins Production for Structural Analysis. , (2014).
- Hastie, K. M., et al. Crystal structure of the prefusion surface glycoprotein of the prototypic arenavirus LCMV. Nature Structural & Molecular Biology. 23 (6), 513-521 (2016).
- Pallesen, J., et al. Structures of Ebola virus GP and sGP in complex with therapeutic antibodies. Nature Microbiology. 1 (9), 16128 (2016).
- Micoli, F., Adamo, R., Costantino, P. Protein Carriers for Glycoconjugate Vaccines: History, Selection Criteria, Characterization and New Trends. Molecules. 23 (6), (2018).
- Li, J., et al. Characterizing the Size and Composition of Saposin A Lipoprotein Picodiscs. Analytical Chemistry. 88 (19), 9524-9531 (2016).
- Crichlow, G. V., et al. Dimerization of FIR upon FUSE DNA binding suggests a mechanism of c-myc inhibition. EMBO Journal. 27 (1), 277-289 (2008).
- Kapoor, N., Gupta, R., Menon, S. T., Folta-Stogniew, E., Raleigh, D. P., Sakmar, T. P. Nucleobindin 1 is a calcium-regulated guanine nucleotide dissociation inhibitor of G{alpha}i1. Journal of Biological Chemistry. 285 (41), 31647-31660 (2010).
- Pirruccello, M., Swan, L. E., Folta-Stogniew, E., De Camilli, P. Recognition of the F&H motif by the Lowe syndrome protein OCRL. Nature Structural & Molecular Biology. 18 (7), 789-795 (2011).
- Lockyer, K., Gao, F., Derrick, J. P., Bolgiano, B. Structural correlates of carrier protein recognition in tetanus toxoid-conjugated bacterial polysaccharide vaccines. Vaccine. 33 (11), 1345-1352 (2015).
- Steinbach, T., Wurm, F. R. Degradable Polyphosphoester-Protein Conjugates: "PPEylation" of Proteins. Biomacromolecules. 17 (10), 3338-3346 (2016).
- Das, S., Stivison, E., Folta-Stogniew, E., Oliver, D. Reexamination of the role of the amino terminus of SecA in promoting its dimerization and functional state. Journal of Bacteriology. 190 (21), 7302-7307 (2008).
- Reshetnyak, A. V., et al. The strength and cooperativity of KIT ectodomain contacts determine normal ligand-dependent stimulation or oncogenic activation in cancer. Molecular Cell. 57 (1), 191-201 (2015).
- Zambelli, B., et al. a Chaperone in the Urease Assembly Process, Is an Intrinsically Unstructured GTPase That Specifically Binds Zn2+. Journal of Biological Chemistry. 280 (6), 4684-4695 (2005).
- Ren, X., et al. Hybrid Structural Model of the Complete Human ESCRT-0 Complex. Structure. 17 (3), 406-416 (2009).
- Moriarty, D. F., Fiorillo, C., Miller, C., Colón, W. A truncated peptide model of the mutant P61A FIS forms a stable dimer. Biochimica et Biophysica Acta (BBA) – Proteins and Proteomics. 1774 (1), 78-85 (2007).
- la Garza, C. E., Miranda-Hernández, M. P., Acosta-Flores, L., Pérez, N. O., Flores-Ortiz, L. F., Medina-Rivero, E. Analysis of therapeutic proteins and peptides using multiangle light scattering coupled to ultra high performance liquid chromatography. Journal of Separation Science. 38 (9), 1537-1543 (2015).
- Beirne, J., Truchan, H., Rao, L. Development and qualification of a size exclusion chromatography coupled with multiangle light scattering method for molecular weight determination of unfractionated heparin. Analytical and Bioanalytical Chemistry. 399 (2), 717-725 (2011).
- Wang, W., et al. A new green technology for direct production of low molecular weight chitosan. Carbohydrate Polymers. 74 (1), 127-132 (2018).
- Kaderli, S., et al. A novel biocompatible hyaluronic acid-chitosan hybrid hydrogel for osteoarthrosis therapy. International Journal of Pharmaceutics. 483 (1-2), 158-168 (2015).
- Porterfield, J. Z., Zlotnick, A. A Simple and General Method for Determining the Protein and Nucleic Acid Content of Viruses by UV Absorbance. Virology. 407 (2), 281-288 (2010).
- Podzimek, S. The use of GPC coupled with a multiangle laser light scattering photometer for the characterization of polymers. On the determination of molecular weight, size and branching. Journal of Applied Polymer Science. 54 (1), 91-103 (1994).
- Podzimek, S., Vlcek, T., Johann, C. Characterization of branched polymers by size exclusion chromatography coupled with multiangle light scattering detector. I. Size exclusion chromatography elution behavior of branched polymers. Journal of Applied Polymer Science. 81 (7), 1588-1594 (2001).
- Podzimek, S. Importance of Multi-Angle Light Scattering in Polyolefin Characterization. Macromolecular Symposia. 330 (1), 81-91 (2013).
- Tarazona, M. P., Saiz, E. Combination of SEC/MALS experimental procedures and theoretical analysis for studying the solution properties of macromolecules. Journal of Biochemical and Biophysical Methods. 56 (1), 95-116 (2003).
- Minton, A. P. Recent applications of light scattering measurement in the biological and biopharmaceutical sciences. Analytical Biochemistry. 501, 4-22 (2016).
- Hirayama, K., Akashi, S., Furuya, M., Fukuhara, K. Rapid confirmation and revision of the primary structure of bovine serum albumin by ESIMS and frit-FAB LC/MS. Biochemical and Biophysical Research Communications. 173 (2), 639-646 (1990).
- Zhu, H., Ownby, D. W., Riggs, C. K., Nolasco, N. J., Stoops, J. K., Riggs, A. F. Assembly of the gigantic hemoglobin of the earthworm Lumbricus terrestris. Roles of subunit equilibria, non-globin linker chains, and valence of the heme iron. The Journal of biological chemistry. 271 (47), 30007-30021 (1996).
- Peters, T., Feldhoff, R. C. Fragments of bovine serum albumin produced by limited proteolysis. Isolation and characterization of tryptic fragments. Biochemistry. 14 (15), 3384-3391 (1975).
- Lebendiker, M., Danieli, T. Production of prone-to-aggregate proteins. FEBS Letters. 588 (2), 236-246 (2014).
- Amartely, H., Avraham, O., Friedler, A., Livnah, O., Lebendiker, M. Coupling Multi Angle Light Scattering to Ion Exchange chromatography (IEX-MALS) for protein characterization. Scientific Reports. 8 (1), 6907 (2018).
- Astafieva, I. V., Eberlein, G. A., John Wang, Y. Absolute on-line molecular mass analysis of basic fibroblast growth factor and its multimers by reversed-phase liquid chromatography with multi-angle laser light scattering detection. Journal of Chromatography A. 740 (2), 215-229 (1996).
- Onsberg, M., Øgendal, L. H., Jensen, M. L., Howells, L. B., Andersen, B., Bjerrum, M. J. Light scattering coupled with reversed phase chromatography to study protein self-association under separating conditions. Journal of Chromatography B. 938, 60-64 (2013).
- Kim, Y., et al. High-throughput protein purification and quality assessment for crystallization. Methods. 55 (1), 12-28 (2011).

