Caracterización de proteínas por cromatografía de exclusión de tamaño acoplada a dispersión de luz multiángulo (SEC-MALS)
Summary
Este protocolo describe la combinación de cromatografía de exclusión de tamaño con dispersión de luz multiángulo (SEC-MALS) para la caracterización absoluta de proteínas y complejos en solución. SEC-MALS determina el peso molecular y el tamaño de proteínas puras, oligómeros nativos, heterocomplejos y proteínas modificadas como glicoproteínas.
Abstract
La cromatografía analítica de exclusión de tamaño (SEC), comúnmente utilizada para la determinación del peso molecular de proteínas y complejos proteínas-proteínas en solución, es una técnica relativa que se basa en el volumen de elución del analito para estimar el Peso. Cuando la proteína no es globular o sufre interacciones de columna no ideales, la curva de calibración basada en estándares de proteína no es válida, y el peso molecular determinado a partir del volumen de elución es incorrecto. La dispersión de luz multiángulo (MALS) es una técnica absoluta que determina el peso molecular de un analito en solución a partir de ecuaciones físicas básicas. La combinación de SEC para la separación con MALS para el análisis constituye un medio versátil y confiable para caracterizar las soluciones de una o más especies de proteínas, incluidos monómeros, oligómeros o agregados nativos, y heterocomplejos. Dado que la medición se realiza en cada volumen de elución, SEC-MALS puede determinar si un pico de elución es homogéneo o heterogéneo y distinguir entre una distribución de peso molecular fija frente al equilibrio dinámico. También es posible el análisis de proteínas modificadas como glicoproteínas o lipoproteínas, o conjugados como las proteínas de membrana sofilizadas con detergente. Por lo tanto, SEC-MALS es una herramienta crítica para el químico proteico que debe confirmar las propiedades biofísicas y el comportamiento de la solución de las moléculas producidas para la investigación biológica o biotecnológica. Este protocolo para SEC-MALS analiza el peso molecular y el tamaño de los monómeros y agregados de proteínas puras. Los datos adquiridos sirven como base para nuevos análisis SEC-MALS, incluidos los de complejos, glicoproteínas y proteínas de membrana unidas a surfactantes.
Introduction
El análisis fiable del peso molecular (MW) de las proteínas en solución es esencial para la investigación biomolecular1,2,3,4. El análisis MW informa al científico si se ha producido la proteínacorrecta y si es adecuada para su uso en la experimentación posterior 5,6. Como se describe en los sitios web de las redes de investigación proteica P4EU7 y ARBRE-Mobieu8, el control de la calidad de las proteínas debe caracterizar no sólo la pureza del producto final, sino también su estado oligomérico, homogeneidad, identidad, conformación, estructura, modificaciones posteriores a la traducción y otras propiedades.
La medición de MW en solución no desnaturalizante identifica la forma de la proteína que está presente en un entorno acuoso, ya sea monomérico u oligomérico. Mientras que para muchas proteínas el objetivo es producir la forma monomérica, para otros un oligómero nativo específico es clave para la actividad biológica9,10,11,12. Otros oligómeros y agregados no nativos son indeseables y darán lugar a defectos en la determinación estructural por cristalografía, resonancia magnética nuclear (RMN) o dispersión de rayos X de ángulo pequeño, así como artefactos o imprecisiones en ensayos funcionales cuantificar la unión por calorimetría de valoración isotérmica o resonancia de plasmón superficial2,13.
En el caso de bioterapias como los anticuerpos monoclonales (mAbs), el análisis de MW basado en soluciones tiene un propósito similar de control de calidad y caracterización del producto. Los agregados y fragmentos excesivos son indicativos de un producto inestable que no es adecuado para uso humano. Los organismos reguladores requieren una caracterización cuidadosa, no sólo de la molécula terapéutica, sino también de los posibles degradantes que pueden estar presentes en el producto final14,15,16,17.
Algunos de los métodos más extendidos para analizar la proteína MW son la electroforesis de gel de poliacrilamida de dodecil sulfato sódico (SDS-PAGE), la electroforesis capilar (CE), la PAGE nativa, la espectrometría de masas (MS), la cromatografía de exclusión de tamaño (SEC) y la analítica ultracentrifugación (AUC). De estos, SDS-PAGE, CE y MS no se realizan en el estado nativo y normalmente conducen a la disociación de oligómeros y agregados, por lo tanto no son adecuados para determinar el oligómero nativo o cuantificar agregados. Aunque PAGE nativo, teóricamente, conserva el estado nativo, en nuestra experiencia es difícil optimizar para muchas proteínas, y los resultados no son muy confiables. AUC, ya sea por velocidad de sedimentación o equilibrio de sedimentación, es cuantitativo y puede determinar MW a partir de los primeros principios, pero es bastante engorroso, que requiere mucha mano de obra manual y experiencia significativa en la interpretación de datos, un largo tiempo de experimento y un instrumento muy caro.
La SEC analítica es un método cuantitativo y relativamente robusto y sencillo que separa las macromoléculas durante el flujo a través de una columna empaquetada. Los principios y aplicaciones de la SEC están bien presentados en varios exámenes18,19,20 y en el manual “Cromatografía de Exclusión de Tamaño: Principios y Métodos”21. Las diferencias en la retención se deben a diferentes cantidades de tiempo dedicado a la difusión dentro y fuera de los poros en la fase estacionaria antes de eludar desde el final de la columna. Las diferencias surgen (nominalmente) de los tamaños relativos y coeficientes de difusión de las moléculas22. Una curva de calibración se construye utilizando una serie de moléculas de referencia, relacionando el MW de la molécula con el volumen de elución. En el caso de las proteínas, las moléculas de referencia son generalmente bien comportadas, proteínas globulares que no interactúan con la columna a través de la carga o residuos de superficie hidrófoba. El volumen de elución se mide con un detector de absorbancia ultravioleta (UV). Si el coeficiente de extinción UV se conoce a menudo calculado a partir de la secuencia, la masa total máxima de la proteína también puede ser cuantificada.
En particular, el análisis de MW por SEC se basa en dos supuestos clave con respecto a las proteínas a caracterizar: 1) comparten con las normas de referencia la misma conformación y volumen específico (en otras palabras, la misma relación entre las propiedades de difusión y MW) y 2) al igual que las normas de referencia, no interactúan con la columna excepto por propiedades legales,no se adhieren al embalaje de la columna por carga o interacciones hidrofóbicas. Las desviaciones de estos supuestos invalidan la curva de calibración y conducen a determinaciones erróneas de MW. Este es el caso de las proteínas intrínsecamente desordenadas que tienen grandes radios Stokes debido a sus extensas regiones no estructuradas23,24 o conjuntos oligoméricos no esféricos/lineales10. Las proteínas glicosiladas generalmente tendrán un radio Stokes más grande que la forma no glucosilada, incluso cuando se tenga en cuenta la masa de carbohidratos añadida19. Las proteínas de membrana solubibilizadas detergente eluyen de manera diferente que las proteínas de calibración porque su elución de SEC depende del tamaño total del complejo de polipéptidos-detergentes-lípidos en lugar del estado oligomérico y la masa molar de la proteína25 ,26. La química de las columnas, el pH y las condiciones de sal afectan todos los volúmenes de elución de proteínas con residuos de superficie cargados o hidrofóbicos27,28.
SEC se vuelve mucho más versátil y fiable para la determinación de MW cuando se combina con detectores de dispersión de luz multiángulo (MALS) e índice diferencial de refracción (dRI)3,4,11,29, 30,31,32. Un detector dRI determina la concentración en función del cambio en el índice de refracción de la solución debido a la presencia del analito. Un detector MALS mide la proporción de luz dispersa por un analito en múltiples ángulos en relación con el rayo láser incidente. Conocida colectivamente como SEC-MALS, esta instrumentación determina el MW independientemente del tiempo de elución, ya que el MW se puede calcular directamente a partir de los primeros principios utilizando la Ecuación 1,
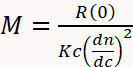 ①
①
donde M es el peso molecular del analito, R(0) la relación reducida con Rayleigh (es decir, la cantidad de luz dispersada por el analito en relación con la intensidad del láser) determinada por el detector MALS y extrapolada al ángulo cero, c el concentración de peso determinada por el detector UV o dRI, dn/dc el incremento del índice de refracción del analito (esencialmente la diferencia entre el índice de refracción del analito y el búfer), y K una constante óptica que depende de la propiedades del sistema, como la longitud de onda y el índice de refracción de disolvente29.
En SEC-MALS, la columna SEC se utiliza únicamente para separar las diversas especies en solución para que entren en las células del detector de concentración y MALS individualmente. El tiempo de retención real no tiene importancia para el análisis, excepto en cuanto a lo bien que resuelve las especies de proteínas. Los instrumentos se calibran independientemente de la columna y no se basan en normas de referencia. Por lo tanto, SEC-MALS se considera un método “absoluto” para la determinación de MW a partir de ecuaciones físicas básicas. Si la muestra es heterogénea y no está completamente separada por la columna, entonces el valor proporcionado en cada volumen de elución será un promedio de peso de las moléculas en cada volumen de elución que fluye a través de la celda de flujo por segmento de tiempo, aproximadamente 75 L.
Mediante el análisis de la variación angular de la intensidad de dispersión, MALS también puede determinar el tamaño (radio medio-cuadrado raíz, Rg) de macromoléculas y nanopartículas con un radio geométrico mayor que aproximadamente 12,5 nm29. Para especies más pequeñas como proteínas monoméricas y oligómeros, se puede añadir un módulo de dispersión de luz dinámica (DLS) al instrumento MALS para medir los radios hidrodinámicos a partir de 0,5 nm y hasta33.
Mientras que el análisis de concentración UV o dRI puede proporcionar el valor de c en Eq. 1, se prefiere el uso de dRI por dos razones: 1) dRI es un detector de concentración universal, adecuado para analizar moléculas como azúcares o polisacáridos que no contienen un UV cromóforo34; y 2) la respuesta de concentración dn/dc de casi todas las proteínas puras en tampón acuoso es la misma dentro de uno o dos por ciento (0.185 mL/g)35,por lo que no hay necesidad de conocer el coeficiente de extinción UV.
El uso de SEC-MALS en la investigación proteica es bastante extenso. Con mucho, las aplicaciones más comunes son establecer si una proteína purificada es monomérica u oligomérica y el grado de oligomerización, y evaluar los agregados3,10,11,17, 31,36,37,38. La capacidad de hacerlo para las proteínas de membrana sóloubicadas con detergente que no pueden caracterizarse por medios tradicionales es especialmente apreciada, y se han publicado protocolos detallados para ello31,39,40 , 41 , 42 , 43. Otras aplicaciones comunes incluyen el establecimiento del grado de modificación post-traduccional y polidispersidad de glicoproteína, lipoproteínas y conjugados similares4,31,44 , 45 , 46 , 47; la formación (o falta de ella) y la estequiometría absoluta (a diferencia de la relación estequiométrica) de heterocomplejos, incluidos los complejos proteína-proteína, ácido proteico-nucleico y proteína-polisacárido24,46, 48,49,50,51,52; determinar la constante de disociación monomero-dímero49,53,54; y evaluar la conformación proteica55,56. Más allá de las proteínas, SEC-MALS es invaluable para la caracterización de péptidos57,58, polímeros naturales ampliamente heterogéneos como heparinas59 y quitosanses60,61, pequeños virus62 y la mayoría de los tipos de polímeros sintéticos o procesados63,64,65,66. Una extensa bibliografía se puede encontrar en la literatura67 y en línea (a http://www.wyatt.com/bibliography).
Aquí, presentamos un protocolo estándar para ejecutar y analizar un experimento SEC-MALS. La albúmina sérica bovina (BSA) se presenta como un ejemplo para la separación y caracterización de monómeros y oligómeros proteicos. El protocolo BSA determina ciertas constantes del sistema que sirven como base para nuevos análisis SEC-MALS, incluidos los de complejos, glicoproteínas y proteínas de membrana unidas a surfactantes.
Observamos que se puede realizar SEC-MALS utilizando un equipo estándar de cromatografía líquida de alto rendimiento (HPLC) o cromatografía líquida de proteína rápida (FPLC) de muchos proveedores. Este protocolo describe el uso de un sistema FPLC que se encuentra comúnmente en laboratorios que producen proteínas para investigación y desarrollo (ver Tabla de Materiales). Antes de ejecutar el protocolo, el sistema FPLC, los detectores MALS y dRI deberían haber sido instalados, junto con sus respectivos paquetes de software para el control, la adquisición y el análisis de datos según las instrucciones del fabricante y cualquier constante de calibración requerida u otros ajustes introducidos en el software. Se debe colocar un filtro en línea entre la bomba y el inyector con una membrana hidrofílica de poros de 0,1 m instalada.
Protocol
Representative Results
Discussion
El experimento SEC-MALS ha proporcionado una buena separación de monómero, dimero y recortador, y resultados cuantitativos para las masas molares y los tamaños hidrodinámicos de cada pico. Esto a su vez identifica y caracteriza claramente cada especie presente, así como la cuantificación de la pureza. Por lo general, los resultados obtenidos son precisos dentro del 5%, y precisos y repetibles dentro de 1-2%3,69. Este nivel de precisión y repetibilidad permite distinguir con confianza entre las especies que pueden estar cerca en MW, siempre y cuando estén separadas por sec (pueden superponerse parcialmente dentro del mismo pico). Los beneficios para el control de la calidad de las proteínas y la caracterización biofísica fundamental son evidentes.
La verificación de la ausencia de partículas es muy importante para las mediciones sensibles y repetibles de SEC-MALS. Los picos de partículas generalmente aparecen como señales MALS grandes no acompañadas de señales UV o RI comparables. La fase móvil y la muestra deben prepararse cuidadosamente para eliminar dichas partículas. Uso de reactivos de grado HPLC o mejor, filtración de fase móvil y tampón de dilución a 0,1 – 0,2 m (prelavado del filtro para eliminar partículas que siempre están presentes en membranas secas), mantenimiento de botellas de fase móvil extralimpia dasluz y otros cristalería para Se recomienda el EQUILIBRIO de columnas SEC-MALS y de columnas extendidas bajo flujo (para eliminar partículas y agregados que pueden haberse acumulado cuando se detuvo el flujo o una columna que no está en uso). La muestra debe filtrarse al tamaño de poro más pequeño que no elimine el material de interés, por lo general no más de 0,1 m y, si es posible, 0,02 m. Si el filtro se obstruye rápidamente, la muestra puede ser centrifugada, filtrada en etapas de tamaño de poro descendente y/o re-purificada. Cuando los sistemas se mantienen consistentemente con fase móvil de alta calidad, fresca y filtrada, se evitan los choques de columna, las muestras están limpias y no se adhieren a la columna, las mediciones no mostrarán el ruido de partículas antes mencionado y proporcionarán datos de alta calidad.
MALS es agnóstico de los tampones TÍPICOs de SEC para proteínas; muchos otros sistemas de amortiguación además de PBS se pueden utilizar para optimizar la separación y la estabilidad, incluyendo una variedad de excipientes71. MALS también es independiente de la columna específica, que debe seleccionarse para una separación y recuperación óptimas. Las principales preocupaciones de los excipientes con respecto al análisis son cambios significativos en el índice de refracción, lo que lleva a la modificación del incremento específico del índice de refracción dn/dc,y los excipientes que absorben 280 nm cuando se necesita análisis UV. Por ejemplo, la arginina es un excipiente reductor de agregación común que puede afectar dramáticamente el dn/dc de una proteína típica, incluso llevándola al régimen negativo (una proteína con dn/dc negativo todavía puede ser analizada por SEC-MALS si dn/ DC se determina empíricamente, pero si dn/dc a 0 la intensidad de la luz dispersada por la proteína será nula y el análisis de MW será imposible). El tema de los valores dn/DC para proteínas es discutido extensamente por Zhao et al.35 donde se muestra que para los tampones acuosos estándar, la gran mayoría de las proteínas no modificadas caen dentro del 2-3% del valor estándar (0.185 o 0.186 mL/g a 660 nm) , aunque las proteínas por debajo de 10 kDa son más variables, y hay algunas especies que pueden llegar hasta 0,21 ml/g.
Los perfiles de MW en los picos de monómero y dimer de BSA en los datos presentados fueron bastante homogéneos en un 2% o menos, lo que indica que las especies monodispersas. Los valores de MW no uniformes a lo largo de un pico pueden surgir de heterogeneidad o análisis incorrectos. En particular, un perfil de MW de BSA cóncavo («sonrisa») o convexo (en lo que se trata de muecas) podría resultar de no aplicar correctamente la corrección de ampliación de banda. Para otras proteínas, un perfil convexo también podría surgir del equilibrio dinámico entre monómeros y oligómeros, donde la relación entre monómero y oligomero-y por lo tanto el valor mw aparente-depende de la concentración máxima (BSA no exhibe este comportamiento y se utiliza a menudo como una muestra de control para verificar los parámetros de ensanchamiento de banda correctos). Un perfil de MW que varía desde el que conduce al borde final y no cambia con la concentración de la muestra es típico de una distribución de especies de peso molecular que se resuelven parcialmente por la SEC. El equilibrio dinámico se distingue fácilmente de la otra las fuentes de distribuciones aparentemente inhomogéneas mediante la inyección de diferentes cantidades totales de proteínas- la distribución variará con el equilibrio dinámico, pero no con una distribución fija o una corrección incorrecta de ampliación de la banda.
Dadas las restricciones descritas anteriormente, SEC-MALS no es adecuado para varias tareas. No es adecuado para el análisis de muestras brutas; las muestras deben estar bien purificadas por métodos estándar de afinidad y pulido. No tiene suficiente precisión o poder de resolución para identificar mutantes y variantes de una proteína o mAb con la misma o muy cerca de masa, y no se puede utilizar con analitos que no eluden o se separan en una columna SEC, aunque recientemente se ha demostrado que el ion-exchang e o cromatografía de fase inversa se puede combinar con MALS para separar y caracterizar especies que no se resuelven con SEC72,73,74. Cuando las cantidades de proteínas están severamente restringidas, la SEC-MALS puede no ser factible, ya que normalmente requiere 10 – 200 g3 y incluso más puede ser requerida por un sistema FPLC con diámetro interior de tubo superior a 0,25 mm; sin embargo, UHPLC-SEC-MALS puede analizar cantidades más pequeñas. Las proteínas altamente inestables que se agregan al introducción a la fase móvil no son adecuadas para el análisis SEC-MALS, aunque la optimización del búfer mediante dispersión de luz dinámica fuera de línea puede superar este problema75.
A pesar del esfuerzo adicional que implica SEC-MALS, es invaluable para la investigación proteica y es ampliamente utilizado por las comunidades académicas y biofarmacéuticas. Además de la caracterización de monómeros, oligómeros y agregados como se describe en el protocolo anterior, SEC-MALS puede caracterizar proteínas modificadas como glicoproteínas (determinando el MW de los componentes proteicos y glicanos individualmente), tensioactivos o proteínas de membrana sofibilizadas con lípidos (determinando el MW de los componentes de proteínas y solubilizadores individualmente), conjuntos de proteínas como partículas similares a virus, complejos de proteínas-proteínas y ácidos proteicos-nucleicos, polisacáridos, conjugados proteicos-polisacáridos, péptidos y muchas otras biomacromoléculas.
Disclosures
The authors have nothing to disclose.
Acknowledgements
Agradecemos al Dr. Tsafi Danieli (Centro Wolfson de Biología Estructural Aplicada, Universidad Hebrea) por su asesoramiento y colaboraciones. También agradecemos a Daniel Biotech Ltd. (Rehovot, Israel) la asistencia y el establecimiento del sistema analítico FPLC-MALS utilizado en este estudio.
Materials
| AKTA pure | GE Healthcare | 29-0182-26 | fast protein liquid chromatograph (FPLC) |
| AKTA UNICORN | GE Healthcare | FPLC control software | |
| ASTRA | Wyatt Technology | MALS data acquisition and analysis software | |
| Bovine serum albumine (purity >97%) | Sigma | A1900 | analyte |
| DAWN or miniDAWN | Wyatt Technology | WH2 or WTREOS | multi-angle light scattering (MALS) detector |
| Increase 200 10/300 | GE Healthcare | size exclusion column, 200 Å pores, 10 mm i.d., 300 mm length | |
| Optilab | Wyatt Technology | WTREX | differential refractive index (RI) detector |
| Sodium chloride NaCl | Sigma | 71382 | HPLC grade NaCl |
| Stericup bottle top filter polyether sulfone 0.1 µm 1000 mL | Millipore | SCVPU11RE | mobile phase filter |
| Whatman Anotop 10 syringe filter 0.02 µm | GE Healthcare | 6809-1002 | sample filter |
| Whatman Anotop 10 syringe-tip filter, 0.1 µm pore, 10 mm diameter | GE Healthcare | 6809-1012 | sample filter |
| Whatman Anotop 25 syringe filter 0.1 µm | GE Healthcare | 6809-2012 | mobile phase filter |
| WyattQELS | Wyatt Technology | WIQ | dynamic light scattering detector |
References
- Acton, T. B., et al. Robotic cloning and Protein Production Platform of the Northeast Structural Genomics Consortium. Methods in Enzymology. 394, 210-243 (2005).
- Acton, T. B., et al. Preparation of protein samples for NMR structure, function, and small-molecule screening studies. Methods in Enzymology. 493, 21-60 (2011).
- Folta-Stogniew, E., Williams, K. R. Determination of molecular masses of proteins in solution: Implementation of an HPLC size exclusion chromatography and laser light scattering service in a core laboratory. Journal of Biomolecular Technology. 10 (2), 51-63 (1999).
- Kendrick, B. S., Kerwin, B. A., Chang, B. S., Philo, J. S. Online size-exclusion high-performance liquid chromatography light scattering and differential refractometry methods to determine degree of polymer conjugation to proteins and protein-protein or protein-ligand association states. Analytical Biochemistry. 299 (2), 136-146 (2001).
- Hughes, C. S., Longo, E., Phillips-Jones, M. K., Hussain, R. Quality control and biophysical characterisation data of. Data Brief. 14, 41-47 (2017).
- Muthurajan, U., et al. In Vitro Chromatin Assembly: Strategies and Quality Control. Methods in Enzymology. 573, 3-41 (2016).
- P4EU. . Protein Quality Standard PQS. , (2019).
- Arbre Mobieu. . Guidelines on Protein Quality Control. , (2019).
- Bowman, A., et al. The histone chaperones Vps75 and Nap1 form ring-like, tetrameric structures in solution. Nucleic Acids Research. 42 (9), 6038-6051 (2014).
- Bowman, G. R., et al. Oligomerization and higher-order assembly contribute to sub-cellular localization of a bacterial scaffold. Molecular Microbiology. 90 (4), 776-795 (2013).
- Folta-Stogniew, E. Oligomeric states of proteins determined by size-exclusion chromatography coupled with light scattering, absorbance, and refractive index detectors. Methods in Molecular Biology. 328, 97-112 (2006).
- Vieux, E. F., Wohlever, M. L., Chen, J. Z., Sauer, R. T., Baker, T. A. Distinct quaternary structures of the AAA+ Lon protease control substrate degradation. Proceedings of the National Academy of Sciences of the United States of America. 110 (22), E2002-E2008 (2013).
- Group, N.S.. . use of SEC-MALS in crystallization QC. , (2018).
- Ahrer, K., Buchacher, A., Iberer, G., Josic, D., Jungbauer, A. Analysis of aggregates of human immunoglobulin G using size-exclusion chromatography, static and dynamic light scattering. Journal of Chromatography A. 1009 (1-2), 89-96 (2003).
- Narhi, L. O., Schmit, J., Bechtold-Peters, K., Sharma, D. Classification of protein aggregates. Journal of Pharmaceutical Sciences. 101 (2), 493-498 (2012).
- Spiess, C., et al. Bispecific antibodies with natural architecture produced by co-culture of bacteria expressing two distinct half-antibodies. Nature Biotechnology. 31 (8), 753-758 (2013).
- Philo, J. S. A critical review of methods for size characterization of non-particulate protein aggregates. Current Pharmaceutical Biotechnology. 10 (4), 359-372 (2009).
- Stellwagen, E. Gel filtration. Methods in Enzymology. 463, 373-385 (2009).
- Burgess, R. R. A brief practical review of size exclusion chromatography: Rules of thumb, limitations, and troubleshooting. Protein Expression and Purification. 150, 81-85 (2018).
- Striegel, A. M., Yau, W. W., Kirkland, J. J., Bly, D. D. . Modern Size-Exclusion Liquid Chromatography. , (2009).
- GE Healthcare. . Size Exclusion Chromatography: Principles and Methods. , (2014).
- Uliyanchenko, E. Size-exclusion chromatography-from high-performance to ultra-performance. Analytical and Bioanalytical Chemistry. 406 (25), 6087-6094 (2014).
- Dunker, A. K., Silman, I., Uversky, V. N., Sussman, J. L. Function and structure of inherently disordered proteins. Current Opinion in Structural Biology. 18 (6), 756-764 (2008).
- Hsiao, H. H., Nath, A., Lin, C. Y., Folta-Stogniew, E. J., Rhoades, E., Braddock, D. T. Quantitative characterization of the interactions among c-myc transcriptional regulators FUSE, FBP, and FIR. Biochemistry. 49 (22), 4620-4634 (2010).
- Hayashi, Y., Takagi, T., Maezawa, S., Matsui, H. Molecular weights of alpha beta-protomeric and oligomeric units of soluble (Na+, K+)-ATPase determined by low-angle laser light scattering after high-performance gel chromatography. Biochimica et Biophysica Acta. 748 (2), 153-167 (1983).
- Folta-Stogniew, E. J. Macromolecular Interactions: Light Scattering. Encyclopedia of Life Sciences. , (2009).
- Hong, P., Koza, S., Bouvier, E. S. P. Size-Exclusion Chromatography for the Analysis of Protein Biotherapeutics and their Aggregates. Journal of Liquid Chromatography and Related Technology. 35 (20), 2923-2950 (2012).
- Chakrabarti, A. Separation of Monoclonal Antibodies by Analytical Size Exclusion Chromatography. Antibody Engineering. , (2018).
- Wyatt, P. J. Light scattering and the absolute characterization of macromolecules. Analytica Chimica Acta. 272, (1993).
- Takagi, T. Application of low-angle laser light scattering detection in the field of biochemistry: review of recent progress. Journal of Chromatography A. 506, 51-63 (1990).
- Wen, J., Arakawa, T., Philo, J. S. Size-exclusion chromatography with on-line light-scattering, absorbance, and refractive index detectors for studying proteins and their interactions. Analytical Biochemistry. 240 (2), 155-166 (1996).
- Mogridge, J. Using light scattering to determine the stoichiometry of protein complexes. Methods in Molecular Biology. 1278, 233-238 (2015).
- Larkin, M., Wyatt, P. J. Light-Scattering Techniques and their Application to Formulation and Aggregation Concerns. Formulation and Process Development Strategies for Manufacturing Biopharmaceuticals. , (2010).
- Wolfender, J. L. HPLC in Natural Product Analysis: The Detection Issue. Planta Medica. 75 (07), 719-734 (2009).
- Zhao, H., Brown, P. H., Schuck, P. On the distribution of protein refractive index increments. Biophysical Journal. 100, (2011).
- Serebryany, E., Folta-Stogniew, E., Liu, J., Yan, E. C. Homodimerization enhances both sensitivity and dynamic range of the ligand-binding domain of type 1 metabotropic glutamate receptor. FEBS Letters. 590 (23), 4308-4317 (2016).
- Arakawa, T., Wen, J. Size-exclusion chromatography with on-line light scattering. Current Protocols in Protein Science. Chapter 20, Unit 20.6 (2001).
- Müller, R., et al. High-resolution structures of the IgM Fc domains reveal principles of its hexamer formation. Proceedings of the National Academy of Sciences. 110 (25), 10183-10188 (2013).
- Slotboom, D. J., Duurkens, R. H., Olieman, K., Erkens, G. B. Static light scattering to characterize membrane proteins in detergent solution. Methods. 46 (2), 73-82 (2008).
- Miercke, L. J., Robbins, R. A., Stroud, R. M. Tetra detector analysis of membrane proteins. Current Protocols in Protein Science. 77, 1-30 (2014).
- Gimpl, K., Klement, J., Keller, S. Characterising protein/detergent complexes by triple-detection size-exclusion chromatography. Biological Procedures Online. 18 (1), 4 (2016).
- Korepanova, A., Matayoshi, E. D. HPLC-SEC characterization of membrane protein-detergent complexes. Current Protocols in Protein Science. Chapter 29, 1-12 (2012).
- Roy, A., Breyton, C., Ebel, C. Analytical Ultracentrifugation and Size-Exclusion Chromatography Coupled with Light Scattering for Characterization of Membrane Proteins in Solution. Membrane Proteins Production for Structural Analysis. , (2014).
- Hastie, K. M., et al. Crystal structure of the prefusion surface glycoprotein of the prototypic arenavirus LCMV. Nature Structural & Molecular Biology. 23 (6), 513-521 (2016).
- Pallesen, J., et al. Structures of Ebola virus GP and sGP in complex with therapeutic antibodies. Nature Microbiology. 1 (9), 16128 (2016).
- Micoli, F., Adamo, R., Costantino, P. Protein Carriers for Glycoconjugate Vaccines: History, Selection Criteria, Characterization and New Trends. Molecules. 23 (6), (2018).
- Li, J., et al. Characterizing the Size and Composition of Saposin A Lipoprotein Picodiscs. Analytical Chemistry. 88 (19), 9524-9531 (2016).
- Crichlow, G. V., et al. Dimerization of FIR upon FUSE DNA binding suggests a mechanism of c-myc inhibition. EMBO Journal. 27 (1), 277-289 (2008).
- Kapoor, N., Gupta, R., Menon, S. T., Folta-Stogniew, E., Raleigh, D. P., Sakmar, T. P. Nucleobindin 1 is a calcium-regulated guanine nucleotide dissociation inhibitor of G{alpha}i1. Journal of Biological Chemistry. 285 (41), 31647-31660 (2010).
- Pirruccello, M., Swan, L. E., Folta-Stogniew, E., De Camilli, P. Recognition of the F&H motif by the Lowe syndrome protein OCRL. Nature Structural & Molecular Biology. 18 (7), 789-795 (2011).
- Lockyer, K., Gao, F., Derrick, J. P., Bolgiano, B. Structural correlates of carrier protein recognition in tetanus toxoid-conjugated bacterial polysaccharide vaccines. Vaccine. 33 (11), 1345-1352 (2015).
- Steinbach, T., Wurm, F. R. Degradable Polyphosphoester-Protein Conjugates: "PPEylation" of Proteins. Biomacromolecules. 17 (10), 3338-3346 (2016).
- Das, S., Stivison, E., Folta-Stogniew, E., Oliver, D. Reexamination of the role of the amino terminus of SecA in promoting its dimerization and functional state. Journal of Bacteriology. 190 (21), 7302-7307 (2008).
- Reshetnyak, A. V., et al. The strength and cooperativity of KIT ectodomain contacts determine normal ligand-dependent stimulation or oncogenic activation in cancer. Molecular Cell. 57 (1), 191-201 (2015).
- Zambelli, B., et al. a Chaperone in the Urease Assembly Process, Is an Intrinsically Unstructured GTPase That Specifically Binds Zn2+. Journal of Biological Chemistry. 280 (6), 4684-4695 (2005).
- Ren, X., et al. Hybrid Structural Model of the Complete Human ESCRT-0 Complex. Structure. 17 (3), 406-416 (2009).
- Moriarty, D. F., Fiorillo, C., Miller, C., Colón, W. A truncated peptide model of the mutant P61A FIS forms a stable dimer. Biochimica et Biophysica Acta (BBA) – Proteins and Proteomics. 1774 (1), 78-85 (2007).
- la Garza, C. E., Miranda-Hernández, M. P., Acosta-Flores, L., Pérez, N. O., Flores-Ortiz, L. F., Medina-Rivero, E. Analysis of therapeutic proteins and peptides using multiangle light scattering coupled to ultra high performance liquid chromatography. Journal of Separation Science. 38 (9), 1537-1543 (2015).
- Beirne, J., Truchan, H., Rao, L. Development and qualification of a size exclusion chromatography coupled with multiangle light scattering method for molecular weight determination of unfractionated heparin. Analytical and Bioanalytical Chemistry. 399 (2), 717-725 (2011).
- Wang, W., et al. A new green technology for direct production of low molecular weight chitosan. Carbohydrate Polymers. 74 (1), 127-132 (2018).
- Kaderli, S., et al. A novel biocompatible hyaluronic acid-chitosan hybrid hydrogel for osteoarthrosis therapy. International Journal of Pharmaceutics. 483 (1-2), 158-168 (2015).
- Porterfield, J. Z., Zlotnick, A. A Simple and General Method for Determining the Protein and Nucleic Acid Content of Viruses by UV Absorbance. Virology. 407 (2), 281-288 (2010).
- Podzimek, S. The use of GPC coupled with a multiangle laser light scattering photometer for the characterization of polymers. On the determination of molecular weight, size and branching. Journal of Applied Polymer Science. 54 (1), 91-103 (1994).
- Podzimek, S., Vlcek, T., Johann, C. Characterization of branched polymers by size exclusion chromatography coupled with multiangle light scattering detector. I. Size exclusion chromatography elution behavior of branched polymers. Journal of Applied Polymer Science. 81 (7), 1588-1594 (2001).
- Podzimek, S. Importance of Multi-Angle Light Scattering in Polyolefin Characterization. Macromolecular Symposia. 330 (1), 81-91 (2013).
- Tarazona, M. P., Saiz, E. Combination of SEC/MALS experimental procedures and theoretical analysis for studying the solution properties of macromolecules. Journal of Biochemical and Biophysical Methods. 56 (1), 95-116 (2003).
- Minton, A. P. Recent applications of light scattering measurement in the biological and biopharmaceutical sciences. Analytical Biochemistry. 501, 4-22 (2016).
- Hirayama, K., Akashi, S., Furuya, M., Fukuhara, K. Rapid confirmation and revision of the primary structure of bovine serum albumin by ESIMS and frit-FAB LC/MS. Biochemical and Biophysical Research Communications. 173 (2), 639-646 (1990).
- Zhu, H., Ownby, D. W., Riggs, C. K., Nolasco, N. J., Stoops, J. K., Riggs, A. F. Assembly of the gigantic hemoglobin of the earthworm Lumbricus terrestris. Roles of subunit equilibria, non-globin linker chains, and valence of the heme iron. The Journal of biological chemistry. 271 (47), 30007-30021 (1996).
- Peters, T., Feldhoff, R. C. Fragments of bovine serum albumin produced by limited proteolysis. Isolation and characterization of tryptic fragments. Biochemistry. 14 (15), 3384-3391 (1975).
- Lebendiker, M., Danieli, T. Production of prone-to-aggregate proteins. FEBS Letters. 588 (2), 236-246 (2014).
- Amartely, H., Avraham, O., Friedler, A., Livnah, O., Lebendiker, M. Coupling Multi Angle Light Scattering to Ion Exchange chromatography (IEX-MALS) for protein characterization. Scientific Reports. 8 (1), 6907 (2018).
- Astafieva, I. V., Eberlein, G. A., John Wang, Y. Absolute on-line molecular mass analysis of basic fibroblast growth factor and its multimers by reversed-phase liquid chromatography with multi-angle laser light scattering detection. Journal of Chromatography A. 740 (2), 215-229 (1996).
- Onsberg, M., Øgendal, L. H., Jensen, M. L., Howells, L. B., Andersen, B., Bjerrum, M. J. Light scattering coupled with reversed phase chromatography to study protein self-association under separating conditions. Journal of Chromatography B. 938, 60-64 (2013).
- Kim, Y., et al. High-throughput protein purification and quality assessment for crystallization. Methods. 55 (1), 12-28 (2011).

