Motilidade de Moléculas Únicas e Aglomerados de Kinesin-5 Cin8 Bidiso
Summary
A cinesinismo mitostática bi-direcional-5 Cin8 acumula-se em aglomerados que se dividem e se fundem durante sua motilidade. O acúmulo em clusters também altera a velocidade e a direcionalidade do Cin8. Aqui, é descrito um protocolo de ensaios de motilidade com Cin8-GFP purificado e análise de propriedades motile de moléculas únicas e aglomerados de Cin8.
Abstract
Os motores de cinesina bipolar mitotítica-5 desempenham funções essenciais na dinâmica do fuso. Estes motores exibem uma estrutura homo-tetramérica com dois pares de domínios motores catalíticos, localizados em extremidades opostas do complexo ativo. Esta arquitetura única permite que os motores kinesin-5 cruzem e deslizem para além de microtúbulos de eixo antiparaol (MTs), fornecendo assim a força direcionada externamente que separa os polos do eixo. Anteriormente, acreditava-se que os motores kinesin-5 eram exclusivamente mais-end dirigidos. No entanto, estudos recentes revelaram que vários motores fúngicos de cinesina-5 são menos-finais direcionados ao nível de molécula única e podem mudar a direcionalidade em várias condições experimentais. O Saccharomyces cerevisiae kinesin-5 Cin8 é um exemplo de tal proteína motora bidirecional: em alta resistência iônica, moléculas únicas de Cin8 se movem na direção menos final dos MTs. Também foi demonstrado que o Cin8 forma clusters motile, predominantemente na extremidade inferior dos MTs, e tal agrupamento permite que Cin8 mude de direcionalidade e se submeta a motilidade direcionada lenta e plus-end. Este artigo fornece um protocolo detalhado para todas as etapas de trabalho com kinesin-5 Cin8 marcado por GFP, desde a superexpressão de proteínas em células S. cerevisiae e sua purificação até o ensaio de motilidade de molécula única in vitro . Um método recém-desenvolvido descrito aqui ajuda a diferenciar entre moléculas únicas e aglomerados de Cin8, com base em sua intensidade de fluorescência. Este método permite a análise separada da motilidade de moléculas únicas e aglomerados de Cin8, proporcionando assim a caracterização da dependência da motilidade Cin8 em seu tamanho de cluster.
Introduction
Um grande número de eventos de motilidade dentro das células eucarióticas são mediados pela função das proteínas motoras moleculares. Esses motores se movem ao longo dos filamentos citoesqueléticos, filamentos de actin e microtúbulos (MTs), e convertem a energia química da hidrólise ATP em forças cinéticas e mecânicas necessárias para conduzir a motilidade biológica dentro das células. O S. cerevisiae Cin8, baseado em MT, é uma proteína motora bipolar, homotetrameric quinase-5 que cruza e desliza MTs de eixo1. O Cin8 executa funções essenciais durante a mitose, na montagem do eixo 2,3,4 e no alongamento do eixo durante a anafase 5,6,7. Anteriormente, havia sido demonstrado que o Cin8 é um motor bidirecional, que muda a direcionalidade em diferentes condições experimentais. Por exemplo, sob altas condições de resistência iônica, motores Cin8 únicos movem-se em direção à extremidade inferior dos MTs, enquanto em clusters, em ensaios de deslizamento MT multimotores, e entre MTs antiparalhais, os motores Cin8 movem-se principalmente em direção às extremidades mais altas dos MTs 8,9,10,11,12 . Esses achados foram altamente inesperados por várias razões. Primeiro, o Cin8 carrega seu domínio motor catalítico no amino-terminus e tais motores foram previamente acreditados como exclusivamente mais-end dirigidos, enquanto Cin8 foi mostrado ser menos-fim direcionado ao nível de molécula única. Em segundo lugar, acreditava-se que os motores de cinesina eram unidirecionais, seja de ponta ou de ponta, enquanto o Cin8 mostrou-se bi-direcional, dependendo das condições experimentais. Finalmente, por causa da orientação em MT no fuso mitótico, o papel clássico dos motores cinesina-5 na separação de postes de fuso durante a montagem do fuso e da anfífase B só poderia ser explicado por sua motilidade direcionada plus-end nos MTs que eles cruzam 1,13. Após os primeiros relatos sobre a bidirecionalidade do Cin8, alguns outros motores de cinesina foram demonstrados como bi-direcionais 14,15,16, indicando que a motilidade bidirecional dos motores de cinesina pode ser mais comum do que se acreditava anteriormente.
Foi relatado anteriormente que nas células, o Cin8 também se move de forma bidirecional8, apoiando a noção de que a motilidade bidirecional de alguns motores cinesin-5 é importante para suas funções intracelulares. Além disso, uma vez que os três motores cinesin-5 que foram relatados como bidirecionais são de células fúngicas, um possível papel para a bidalidade bi-direcionalidade dos motores cinesin-5 foi recentemente proposto nessas células10. De acordo com este modelo, em mitose fechada de células fúngicas, onde o envelope nuclear não quebra durante a mitose, os motores de quinase-5 fornecem a força inicial que separa os polos do eixo antes da montagem do eixo. Para realizar esta tarefa, antes da separação do polo de fuso, os motores kinesin-5 localizam-se perto dos polos de fuso, por sua motilidade direcionada sem fim em MTs nucleares únicos. Uma vez nesta posição, os motores kinesin-5 agrupam, alternam direcionalidade, capturam e cruzam MTs de postes de fuso vizinhos. Posteriormente, os motores kinesin-5 fornecem a separação inicial dos polos por motilidade dirigida plus-end nos MTs que eles cruzam. Por este modelo, tanto a motilidade dirigida por fim em MTs único quanto a motilidade direcionada plus-end em MTs transversais durante o deslizamento antiparal são necessárias para que os motores fúngicos kinesin-5 realizem seus papéis na montagemdo eixo 1,13.
O objetivo geral do método descrito é obter kinesina-5 Cin8 marcada por FFP de alta pureza e realizar ensaios de motilidade de molécula única (Figura 1) enquanto analisa separadamente a motilidade de moléculas únicas e aglomerados de Cin8. A separação entre moléculas únicas e aglomerados é importante, uma vez que um dos fatores que foram demonstrados para afetar a direcionalidade do Cin8 é o seu acúmulo em clusters nos MTs10,12. Ensaios alternativos de motilidade, como o deslizamento de superfície mt e ensaios deslizantes de MT não fornecem informações sobre a atividade de proteínas motoras únicas 17,18. Os robustos métodos de ensaio e análise de motilidade de molécula única descritos aqui foram aplicados com sucesso para caracterizar diferentes aspectos dos motores kinesin-5, Cin8 e Kip1 10,11,12,14,19,20.
Aqui, um protocolo detalhado é apresentado para a superexpressão e purificação do Cin8, polimerização de MTs e o ensaio de motilidade de molécula única. Além disso, também são descritas as análises para diferenciar moléculas e aglomerados únicos de Cin8 e determinar velocidades motoras e cluster únicas por deslocamento médio (MD) e análise média de deslocamento quadrado (MSD). Este protocolo tem como objetivo ajudar os pesquisadores a visualizar todas as etapas dos procedimentos e auxiliar na solução de problemas desse tipo de ensaio.
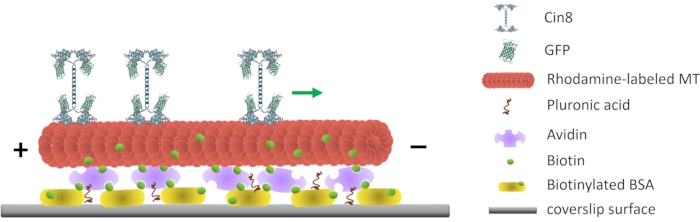
Figura 1: Representação esquemática do ensaio de motilidade de molécula única. Os MTs fluorescentes biotinilados são anexados à superfície de vidro, revestidos com o Avidin que interage com o biotinilado-BSA conectado à superfície. O arqueiro verde representa a direção de movimento de moléculas cin8 únicas sob altas condições de resistência iônica. +/- representar a polaridade do MT. Clique aqui para ver uma versão maior desta figura.
Protocol
Representative Results
Discussion
Neste trabalho, é apresentado um protocolo para ensaio de motilidade de molécula única com a cinesina bidirecional-5 Cin8 e a análise de motilidade. O Cin818 de comprimento completo, incluindo o sinal de localização nuclear nativa (NLS) no terminal C foi purificado do hospedeiro nativo S. cerevisiae. Como o Cin8 é uma proteína motora nuclear, moer as células S. cerevisiae sob nitrogênio líquido é considerado o método mais eficiente para a lise celular. Após a lise, …
Disclosures
The authors have nothing to disclose.
Acknowledgements
Esta pesquisa foi apoiada em parte pela bolsa da Israel Science Foundation (ISF-386/18) e pela bolsa da Israel Binational Science Foundation (BSF-2019008), concedida à L.G.
Materials
| Adenine | FORMEDIUM | DOC0230 | |
| ATP | Sigma | A7699 | |
| Biotinylated-BSA | Sigma | A8549 | |
| Casein | Sigma | C7078 | |
| Catalase (C40) | Sigma | C40 | |
| Creatine-Kinase | Sigma | C3755 | |
| Dithiothreitol (DTT) | Sigma | D0632 | |
| EDTA | Sigma | E5134 | |
| EGTA | Sigma | E4378 | |
| Fluorescence filter set for GFP | Chroma | 49002: ET-EGFP (FITC/Cy2) | |
| Fluorescence filter set for Rhodamine | Chroma | 49004: ET-CY3/TRITC | |
| Fluorescence inverted microscope | Zeiss | Axiovert 200M | |
| Galactose | Tivan Biotech | GAL02 | |
| Glucose | Sigma | G8270 | |
| Glucose Oxidase | Sigma | G7141 | |
| Glycerol | Sigma | G5516 | |
| GlycylGlycine | Merck | G0674 | |
| GMPCPP | Jana Bioscience | Nu-405L | |
| GTB | Cytoskeleton | BST01-010 | |
| GTP | Sigma | G8877 | |
| Histidine | Duchefa Biochemie | H0710.0100 | |
| ImageJ-FIJI software | https://imagej.net/plugins/trackmate/ | version 2.1.0/1.53c; Java 1.8.0_172 [64-bit] for Windows 10 | |
| Imidazole | Sigma | I0125 | |
| InstantBlue Coomassie Protein Stain | Abcam | ab119211 | |
| Lens | Zeiss | 100x/1.4 oil DIC objective | |
| Lysine | FORMEDIUM | DOC0161 | |
| Magnesium Chloride | Sigma | M8266 | |
| Methionine | Duchefa Biochemie | M0715.0100 | |
| Neo | Andor Technologies | sCMOS camera | |
| NeutraAvidin | Life | A2666 | |
| Ni-NTA Agarose | Invitrogen | R901-15 | |
| Phospho-Creatine | Sigma | P1937 | |
| Pipes | Sigma | P1851 | |
| Pluronic acid F-127 (poloxamer) | Sigma | P2443 | |
| Potassium Chloride | Sigma | P9541 | |
| Raffinose | Tivan Biotech | RAF01 | |
| Size Exclusion chromatography instument | GE Healthcare | AKTA Pure | |
| Spectrophotometer | ThermoFisher Scientific | NanoDrop | |
| Superose-6 10/300 GL | GE Healthcare | 17-5172-01 | |
| Tris | Roshe | 10708976001 | |
| Triton X-100 | Sigma | T8787 | |
| Tryptophan | Duchefa Biochemie | T0720.0100 | |
| Tubulin protein | Cytoskeleton | T240 | |
| Tubulin, biotinylated | Cytoskeleton | T333P | |
| Tubulin, TRITC Rhodamine | Cytoskeleton | TL530M | |
| Uracil | Sigma | U0750-100G | |
| Yeast nitrogen base | FORMEDIUM | CYN0401S | |
| α-GFP antibody | Santa Cruz Biotechnology | SC8036 | |
| β-mercaptoethanol | Sigma | M3148 |
References
- Singh, S. K., Pandey, H., Al-Bassam, J., Gheber, L. Bidirectional motility of kinesin-5 motor proteins: structural determinants, cumulative functions and physiological roles. Cellular and Molecular Life Sciences. 75 (10), 1757-1771 (2018).
- Hoyt, M. A., He, L., Totis, L., Saunders, W. S. Loss of function of Saccharomyces cerevisiae kinesin-related CIN8 and KIP1 is suppressed by KAR3 motor domain mutations. Genetics. 135 (1), 35-44 (1993).
- Saunders, W. S., Hoyt, M. A. Kinesin-related proteins required for structural integrity of the mitotic spindle. Cell. 70 (3), 451-458 (1992).
- Hoyt, M. A., He, L., Loo, K. K., Saunders, W. S. Two Saccharomyces cerevisiae kinesin-related gene products required for mitotic spindle assembly. Journal of Cell Biology. 118 (1), 109-120 (1992).
- Gerson-Gurwitz, A., et al. Mid-anaphase arrest in S. cerevisiae cells eliminated for the function of Cin8 and dynein. Cellular and Molecular Life Sciences. 66 (2), 301-313 (2009).
- Fridman, V., Gerson-Gurwitz, A., Movshovich, N., Kupiec, M., Gheber, L. Midzone organization restricts interpolar microtubule plus-end dynamics during spindle elongation. EMBO Reports. 10 (4), 387-393 (2009).
- Movshovich, N., et al. Slk19-dependent mid-anaphase pause in kinesin-5-mutated cells. Journal of Cell Science. 121 (15), 2529-2539 (2008).
- Gerson-Gurwitz, A., et al. Directionality of individual kinesin-5 Cin8 motors is modulated by loop 8, ionic strength and microtubule geometry. Embo Journal. 30 (24), 4942-4954 (2011).
- Roostalu, J., et al. Directional switching of the kinesin Cin8 through motor coupling. Science. 332 (6025), 94-99 (2011).
- Shapira, O., Goldstein, A., Al-Bassam, J., Gheber, L. A potential physiological role for bi-directional motility and motor clustering of mitotic kinesin-5 Cin8 in yeast mitosis. Journal of Cell Science. 130 (4), 725-734 (2017).
- Goldstein-Levitin, A., Pandey, H., Allhuzaeel, K., Kass, I., Gheber, L. Intracellular functions and motile properties of bi-directional kinesin-5 Cin8 are regulated by neck linker docking. eLife. 10, 71036 (2021).
- Pandey, H., et al. Drag-induced directionality switching of kinesin-5 Cin8 revealed by cluster-motility analysis. Science Advances. 7 (6), 1687 (2021).
- Pandey, H., Popov, M., Goldstein-Levitin, A., Gheber, L. Mechanisms by which kinesin-5 motors perform their multiple intracellular functions. International Journal of Molecular Sciences. 22 (12), 6420 (2021).
- Fridman, V., et al. Kinesin-5 Kip1 is a bi-directional motor that stabilizes microtubules and tracks their plus-ends in vivo. Journal of Cell Science. 126, 4147-4159 (2013).
- Edamatsu, M. Bidirectional motility of the fission yeast kinesin-5, Cut7. Biochemical and Biophysical Research Communications. 446 (1), 231-234 (2014).
- Popchock, A. R., et al. The mitotic kinesin-14 KlpA contains a context-dependent directionality switch. Nature Communications. 8, 13999 (2017).
- Bodrug, T., et al. The kinesin-5 tail domain directly modulates the mechanochemical cycle of the motor domain for anti-parallel microtubule sliding. eLife. 9 (9), 51131 (2020).
- Gheber, L., Kuo, S. C., Hoyt, M. A. Motile properties of the kinesin-related Cin8p spindle motor extracted from Saccharomyces cerevisiae cells. Journal of Biological Chemistry. 274 (14), 9564-9572 (1999).
- Pandey, H., et al. Flexible microtubule anchoring modulates the bi-directional motility of the kinesin-5 Cin8. Cellular and Molecular Life Sciences. 78 (16), 6051-6068 (2021).
- Shapira, O., Gheber, L. Motile properties of the bi-directional kinesin-5 Cin8 are affected by phosphorylation in its motor domain. Scientific Reports. 6, 25597 (2016).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Britto, M., et al. Schizosaccharomyces pombe kinesin-5 switches direction using a steric blocking mechanism. Proceedings of the National Academy of Sciences of the United States of America. 113 (47), 7483-7489 (2016).
- Kapitein, L. C., et al. Microtubule cross-linking triggers the directional motility of kinesin-5. Journal of Cell Biology. 182 (3), 421-428 (2008).
- Furuta, K., Edamatsu, M., Maeda, Y., Toyoshima, Y. Y. Diffusion and directed movement in vitro motile properties of fission yeast kinesin-14 Pkl1. Journal of Biological Chemistry. 283 (52), 36465-36473 (2008).
- Katrukha, E. A., et al. Probing cytoskeletal modulation of passive and active intracellular dynamics using nanobody-functionalized quantum dots. Nature Communications. 8, 14772 (2017).
- Tinevez, J. Y., et al. TrackMate: An open and extensible platform for single-particle tracking. Methods. 115, 80-90 (2017).
- Jakobs, M. A. H., Dimitracopoulos, A., Franze, K. KymoBulter, a deep learning software for automated kymograph analysis. eLife. 8, 42288 (2019).

