Motilità di singole molecole e cluster di Kinesin-5 Cin8 bidirezionale purificata da cellule S. cerevisiae
Summary
La cinesina mitotica bidirezionale-5 Cin8 si accumula in ammassi che si dividono e si fondono durante la loro motilità. L’accumulo negli ammassi cambia anche la velocità e la direzionalità di Cin8. Qui viene descritto un protocollo per saggi di motilità con Cin8-GFP purificato e analisi delle proprietà mobili di singole molecole e cluster di Cin8.
Abstract
I motori mitotici bipolari kinesin-5 svolgono funzioni essenziali nella dinamica del mandrino. Questi motori presentano una struttura omo-tetramerica con due coppie di domini motori catalitici, situati alle estremità opposte del complesso attivo. Questa architettura unica consente ai motori kinesin-5 di reticolare e far scorrere i microtubuli antiparallelo del mandrino (MT), fornendo così la forza diretta verso l’esterno che separa i poli del mandrino. In precedenza, si riteneva che i motori kinesin-5 fossero esclusivamente diretti verso il plus-end. Tuttavia, studi recenti hanno rivelato che diversi motori fungini kinesin-5 sono meno diretti a livello di singola molecola e possono cambiare direzionalità in varie condizioni sperimentali. Il Saccharomyces cerevisiae kinesin-5 Cin8 è un esempio di tale proteina motoria bidirezionale: in condizioni di elevata forza ionica singole molecole di Cin8 si muovono nella direzione meno-estremità delle MT. È stato anche dimostrato che Cin8 forma ammassi mobili, prevalentemente all’estremità negativa delle MT, e tale clustering consente a Cin8 di cambiare direzionalità e subire una motilità lenta e diretta plus-end. Questo articolo fornisce un protocollo dettagliato per tutte le fasi di lavoro con kinesin-5 Cin8 con tag GFP, dalla sovraespressione proteica nelle cellule di S. cerevisiae e la sua purificazione al saggio di motilità a singola molecola in vitro . Un metodo di nuova concezione qui descritto aiuta a distinguere tra singole molecole e cluster di Cin8, in base alla loro intensità di fluorescenza. Questo metodo consente un’analisi separata della motilità di singole molecole e cluster di Cin8, fornendo così la caratterizzazione della dipendenza della motilità di Cin8 dalla sua dimensione del cluster.
Introduction
Un gran numero di eventi di motilità all’interno delle cellule eucariotiche sono mediati dalla funzione delle proteine motorie molecolari. Questi motori si muovono lungo i filamenti citoscheletrici, i filamenti di actina e i microtubuli (MT) e convertono l’energia chimica dell’idrolisi dell’ATP in forze cinetiche e meccaniche necessarie per guidare la motilità biologica all’interno delle cellule. La S. cerevisiae Cin8 basata su MT è una proteina motoria bipolare omotetramerica kinesin-5 che reticola e fa scorrere le MT a parte1. Cin8 svolge funzioni essenziali durante la mitosi, nell’assemblaggio del mandrino 2,3,4 e nell’allungamento del mandrino durante l’anafase 5,6,7. In precedenza, era stato dimostrato che Cin8 è un motore bidirezionale, che commuta la direzionalità in diverse condizioni sperimentali. Ad esempio, in condizioni di elevata resistenza ionica, i singoli motori Cin8 si muovono verso l’estremità negativa degli MT, mentre nei cluster, nei saggi di scorrimento MT multimotore e tra GLI MT antiparallelo, i motori Cin8 si muovono principalmente verso le estremità più degli MT 8,9,10,11,12 . Questi risultati sono stati altamente inaspettati a causa di diversi motivi. In primo luogo, Cin8 porta il suo dominio motorio catalitico all’ammino-terminale e tali motori erano precedentemente ritenuti esclusivamente diretti verso il plus-end, mentre Cin8 ha dimostrato di essere minus-end diretto a livello di singola molecola. In secondo luogo, si credeva che i motori kinesin fossero unidirezionali, diretti meno-end o plus-end, mentre Cin8 si è dimostrato bidirezionale, a seconda delle condizioni sperimentali. Infine, a causa dell’orientamento MT al mandrino mitotico, il ruolo classico dei motori kinesin-5 nella separazione dei poli del mandrino durante l’assemblaggio del mandrino e l’anafase B potrebbe essere spiegato solo dalla loro motilità diretta plus-end sugli MT che reticolano 1,13. A seguito dei primi rapporti sulla bidirezionalità di Cin8, alcuni altri motori kinesin sono stati dimostrati bidirezionali 14,15,16, indicando che la motilità bidirezionale dei motori kinesin potrebbe essere più comune di quanto si credesse in precedenza.
È stato precedentemente riportato che nelle cellule, Cin8 si muove anche in modo bidirezionale8, sostenendo l’idea che la motilità bidirezionale di alcuni motori kinesin-5 sia importante per le loro funzioni intracellulari. Inoltre, poiché i tre motori kinesin-5 che sono stati segnalati come bidirezionali provengono da cellule fungine, un possibile ruolo per la bidirezionalità dei motori kinesin-5 è stato recentemente proposto in tali cellule10. Secondo questo modello, nella mitosi chiusa delle cellule fungine, dove l’involucro nucleare non si rompe durante la mitosi, i motori kinesin-5 forniscono la forza iniziale che separa i poli del mandrino prima dell’assemblaggio del mandrino. Per eseguire questo compito, prima della separazione dei poli del mandrino, i motori kinesin-5 si localizzano vicino ai poli del mandrino, per la loro motilità diretta all’estremità inferiore su singoli MT nucleari. Una volta in questa posizione, i motori kinesin-5 si raggruppano, commutano la direzionalità, catturano e collegano incrociato i NET dai poli del mandrino vicini. Successivamente, i motori kinesin-5 forniscono la separazione iniziale dei poli mediante motilità diretta plus-end sugli MT che reticolano. Con questo modello, sia la motilità diretta minus-end su singoli MT che la motilità diretta plus-end su MT reticolati durante lo scorrimento antiparallelo sono necessarie affinché i motori fungini kinesin-5 svolgano i loro ruoli nell’assemblaggio mandrino 1,13.
L’obiettivo generale del metodo descritto è quello di ottenere kinesin-5 Cin8 fungina di elevata purezza marcata con GFP e di eseguire saggi di motilità a singola molecola (Figura 1) analizzando separatamente la motilità di singole molecole e cluster di Cin8. La separazione tra singole molecole e cluster è importante poiché uno dei fattori che ha dimostrato di influenzare la direzionalità di Cin8 è il suo accumulo in cluster sulle MT10,12. Saggi di motilità alternativi, come i saggi di scorrimento superficiale MT e MT sliding non forniscono informazioni sull’attività delle proteine monomotore17,18. I robusti metodi di analisi e analisi della motilità a singola molecola qui descritti sono stati applicati con successo per caratterizzare diversi aspetti dei motori kinesin-5, Cin8 e Kip1 10,11,12,14,19,20.
Qui viene presentato un protocollo dettagliato per la sovraespressione e la purificazione di Cin8, la polimerizzazione delle MT e il test di motilità a singola molecola. Inoltre, vengono descritte anche le analisi per differenziare tra singole molecole e cluster di Cin8 e per determinare le velocità di singolo motore e cluster mediante l’analisi dello spostamento medio (MD) e dello spostamento quadrato medio (MSD). Questo protocollo ha lo scopo di aiutare i ricercatori a visualizzare tutte le fasi delle procedure e assistere nella risoluzione dei problemi di questo tipo di test.
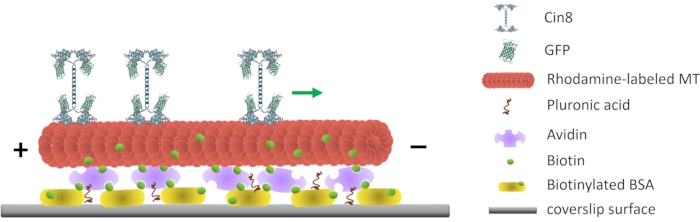
Figura 1: Rappresentazione schematica del saggio di motilità a singola molecola. Le MT fluorescenti biotinilate sono attaccate alla superficie del vetro, rivestite con Avidin che interagisce con il BSA biotinilato attaccato alla superficie. La freccia verde rappresenta la direzione del movimento di singole molecole Cin8 in condizioni di elevata forza ionica. +/- rappresenta la polarità della MT. Fare clic qui per visualizzare una versione più grande di questa figura.
Protocol
Representative Results
Discussion
In questo lavoro, viene presentato un protocollo per il saggio di motilità a singola molecola con la kinesin-5 bidirezionale Cin8 e l’analisi della motilità. Il Cin818 a lunghezza intera, incluso il segnale di localizzazione nucleare nativo (NLS) al C-terminale, è stato purificato dall’ospite nativo S. cerevisiae. Poiché il Cin8 è una proteina del motore nucleare, la macinazione delle cellule S. cerevisiae sotto azoto liquido è il metodo più efficiente per la lisi cellular…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Questa ricerca è stata sostenuta in parte dalla sovvenzione della Israel Science Foundation (ISF-386/18) e dalla sovvenzione della Israel Binational Science Foundation (BSF-2019008), assegnata a L.G.
Materials
| Adenine | FORMEDIUM | DOC0230 | |
| ATP | Sigma | A7699 | |
| Biotinylated-BSA | Sigma | A8549 | |
| Casein | Sigma | C7078 | |
| Catalase (C40) | Sigma | C40 | |
| Creatine-Kinase | Sigma | C3755 | |
| Dithiothreitol (DTT) | Sigma | D0632 | |
| EDTA | Sigma | E5134 | |
| EGTA | Sigma | E4378 | |
| Fluorescence filter set for GFP | Chroma | 49002: ET-EGFP (FITC/Cy2) | |
| Fluorescence filter set for Rhodamine | Chroma | 49004: ET-CY3/TRITC | |
| Fluorescence inverted microscope | Zeiss | Axiovert 200M | |
| Galactose | Tivan Biotech | GAL02 | |
| Glucose | Sigma | G8270 | |
| Glucose Oxidase | Sigma | G7141 | |
| Glycerol | Sigma | G5516 | |
| GlycylGlycine | Merck | G0674 | |
| GMPCPP | Jana Bioscience | Nu-405L | |
| GTB | Cytoskeleton | BST01-010 | |
| GTP | Sigma | G8877 | |
| Histidine | Duchefa Biochemie | H0710.0100 | |
| ImageJ-FIJI software | https://imagej.net/plugins/trackmate/ | version 2.1.0/1.53c; Java 1.8.0_172 [64-bit] for Windows 10 | |
| Imidazole | Sigma | I0125 | |
| InstantBlue Coomassie Protein Stain | Abcam | ab119211 | |
| Lens | Zeiss | 100x/1.4 oil DIC objective | |
| Lysine | FORMEDIUM | DOC0161 | |
| Magnesium Chloride | Sigma | M8266 | |
| Methionine | Duchefa Biochemie | M0715.0100 | |
| Neo | Andor Technologies | sCMOS camera | |
| NeutraAvidin | Life | A2666 | |
| Ni-NTA Agarose | Invitrogen | R901-15 | |
| Phospho-Creatine | Sigma | P1937 | |
| Pipes | Sigma | P1851 | |
| Pluronic acid F-127 (poloxamer) | Sigma | P2443 | |
| Potassium Chloride | Sigma | P9541 | |
| Raffinose | Tivan Biotech | RAF01 | |
| Size Exclusion chromatography instument | GE Healthcare | AKTA Pure | |
| Spectrophotometer | ThermoFisher Scientific | NanoDrop | |
| Superose-6 10/300 GL | GE Healthcare | 17-5172-01 | |
| Tris | Roshe | 10708976001 | |
| Triton X-100 | Sigma | T8787 | |
| Tryptophan | Duchefa Biochemie | T0720.0100 | |
| Tubulin protein | Cytoskeleton | T240 | |
| Tubulin, biotinylated | Cytoskeleton | T333P | |
| Tubulin, TRITC Rhodamine | Cytoskeleton | TL530M | |
| Uracil | Sigma | U0750-100G | |
| Yeast nitrogen base | FORMEDIUM | CYN0401S | |
| α-GFP antibody | Santa Cruz Biotechnology | SC8036 | |
| β-mercaptoethanol | Sigma | M3148 |
References
- Singh, S. K., Pandey, H., Al-Bassam, J., Gheber, L. Bidirectional motility of kinesin-5 motor proteins: structural determinants, cumulative functions and physiological roles. Cellular and Molecular Life Sciences. 75 (10), 1757-1771 (2018).
- Hoyt, M. A., He, L., Totis, L., Saunders, W. S. Loss of function of Saccharomyces cerevisiae kinesin-related CIN8 and KIP1 is suppressed by KAR3 motor domain mutations. Genetics. 135 (1), 35-44 (1993).
- Saunders, W. S., Hoyt, M. A. Kinesin-related proteins required for structural integrity of the mitotic spindle. Cell. 70 (3), 451-458 (1992).
- Hoyt, M. A., He, L., Loo, K. K., Saunders, W. S. Two Saccharomyces cerevisiae kinesin-related gene products required for mitotic spindle assembly. Journal of Cell Biology. 118 (1), 109-120 (1992).
- Gerson-Gurwitz, A., et al. Mid-anaphase arrest in S. cerevisiae cells eliminated for the function of Cin8 and dynein. Cellular and Molecular Life Sciences. 66 (2), 301-313 (2009).
- Fridman, V., Gerson-Gurwitz, A., Movshovich, N., Kupiec, M., Gheber, L. Midzone organization restricts interpolar microtubule plus-end dynamics during spindle elongation. EMBO Reports. 10 (4), 387-393 (2009).
- Movshovich, N., et al. Slk19-dependent mid-anaphase pause in kinesin-5-mutated cells. Journal of Cell Science. 121 (15), 2529-2539 (2008).
- Gerson-Gurwitz, A., et al. Directionality of individual kinesin-5 Cin8 motors is modulated by loop 8, ionic strength and microtubule geometry. Embo Journal. 30 (24), 4942-4954 (2011).
- Roostalu, J., et al. Directional switching of the kinesin Cin8 through motor coupling. Science. 332 (6025), 94-99 (2011).
- Shapira, O., Goldstein, A., Al-Bassam, J., Gheber, L. A potential physiological role for bi-directional motility and motor clustering of mitotic kinesin-5 Cin8 in yeast mitosis. Journal of Cell Science. 130 (4), 725-734 (2017).
- Goldstein-Levitin, A., Pandey, H., Allhuzaeel, K., Kass, I., Gheber, L. Intracellular functions and motile properties of bi-directional kinesin-5 Cin8 are regulated by neck linker docking. eLife. 10, 71036 (2021).
- Pandey, H., et al. Drag-induced directionality switching of kinesin-5 Cin8 revealed by cluster-motility analysis. Science Advances. 7 (6), 1687 (2021).
- Pandey, H., Popov, M., Goldstein-Levitin, A., Gheber, L. Mechanisms by which kinesin-5 motors perform their multiple intracellular functions. International Journal of Molecular Sciences. 22 (12), 6420 (2021).
- Fridman, V., et al. Kinesin-5 Kip1 is a bi-directional motor that stabilizes microtubules and tracks their plus-ends in vivo. Journal of Cell Science. 126, 4147-4159 (2013).
- Edamatsu, M. Bidirectional motility of the fission yeast kinesin-5, Cut7. Biochemical and Biophysical Research Communications. 446 (1), 231-234 (2014).
- Popchock, A. R., et al. The mitotic kinesin-14 KlpA contains a context-dependent directionality switch. Nature Communications. 8, 13999 (2017).
- Bodrug, T., et al. The kinesin-5 tail domain directly modulates the mechanochemical cycle of the motor domain for anti-parallel microtubule sliding. eLife. 9 (9), 51131 (2020).
- Gheber, L., Kuo, S. C., Hoyt, M. A. Motile properties of the kinesin-related Cin8p spindle motor extracted from Saccharomyces cerevisiae cells. Journal of Biological Chemistry. 274 (14), 9564-9572 (1999).
- Pandey, H., et al. Flexible microtubule anchoring modulates the bi-directional motility of the kinesin-5 Cin8. Cellular and Molecular Life Sciences. 78 (16), 6051-6068 (2021).
- Shapira, O., Gheber, L. Motile properties of the bi-directional kinesin-5 Cin8 are affected by phosphorylation in its motor domain. Scientific Reports. 6, 25597 (2016).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Britto, M., et al. Schizosaccharomyces pombe kinesin-5 switches direction using a steric blocking mechanism. Proceedings of the National Academy of Sciences of the United States of America. 113 (47), 7483-7489 (2016).
- Kapitein, L. C., et al. Microtubule cross-linking triggers the directional motility of kinesin-5. Journal of Cell Biology. 182 (3), 421-428 (2008).
- Furuta, K., Edamatsu, M., Maeda, Y., Toyoshima, Y. Y. Diffusion and directed movement in vitro motile properties of fission yeast kinesin-14 Pkl1. Journal of Biological Chemistry. 283 (52), 36465-36473 (2008).
- Katrukha, E. A., et al. Probing cytoskeletal modulation of passive and active intracellular dynamics using nanobody-functionalized quantum dots. Nature Communications. 8, 14772 (2017).
- Tinevez, J. Y., et al. TrackMate: An open and extensible platform for single-particle tracking. Methods. 115, 80-90 (2017).
- Jakobs, M. A. H., Dimitracopoulos, A., Franze, K. KymoBulter, a deep learning software for automated kymograph analysis. eLife. 8, 42288 (2019).

