Split-Luciferase Reassembly Assay to Measure Endoplasmic Reticulum-Mitochondria Contacts in Live Cells
Summary
We have established a split-luciferase reassembly assay to monitor the endoplasmic reticulum-mitochondria contacts in live cells. Using this assay, we describe a protocol to quantitatively measure the level of these inter-organelle couplings in HEK293T cells, under the condition of chemical treatment.
Abstract
Endoplasmic reticulum (ER)-mitochondria contact sites play a critical role in cell health and homeostasis, such as the regulation of Ca2+ and lipid homeostasis, mitochondrial dynamics, autophagosome and mitophagosome biogenesis, and apoptosis. Failure to maintain normal ER-mitochondrial coupling is implicated in many neurodegenerative diseases such as Alzheimer's disease, Parkinson's disease, amyotrophic lateral sclerosis, and hereditary spastic paraplegia. It is of considerable significance to explore how the dysregulation of ER-mitochondrial contacts could lead to cell death and whether repairing these contacts to the normal level could ameliorate neurodegenerative conditions. Thus, improved assays that measure the level of these contacts could help to illuminate the pathogenic mechanisms of these diseases. Ultimately, establishing simple and reliable assays will facilitate the development of new therapeutic strategies. Here we describe a split-luciferase assay to quantitatively measure the level of ER-mitochondria contacts in live cells. This assay can be used to study the pathophysiological role of these contacts as well as to identify their modulators in high-throughput screening.
Introduction
The interactions between the ER and the mitochondria are vital for cellular homeostasis and survival1,2,3,4. Previous evidence indicates that any type of disruption or dysregulation in ER-mitochondria contact sites can contribute to several neurodegenerative, metabolic, and cardiovascular diseases, as well as cancer5,6,7,8,9,10. For example, an abnormal increase of Ca2+ uptake into the mitochondria can lead to cell death by opening mitochondria permeability transition pores, which are commonly seen in some models of Alzheimer's disease5,11. Similarly, reduced ER-mitochondria contacts can result in a decrease in ATP production and impairment of Ca2+ intake, as seen in models of amyotrophic lateral sclerosis5,11,12. As more studies are being conducted in the realm of ER-mitochondria contacts, additional disease-related proteins and genes that could affect these contacts are being discovered. Despite current knowledge and evidence showcasing the role of ER-mitochondria contact sites, much work is still needed to elucidate how these contacts could lead to loss of cellular function and ultimately cell death.
There have been various methods developed to evaluate the proximity of the two membranes, structural morphology, and the distance between the two organelle contact sites3,4,13. The approaches to monitor ER-mitochondria coupling include fluorescence marker-based imaging14,15, FRET-reporter-based imaging16, and split-fluorescence-probe-based imaging17,18, which use epifluorescence and confocal microscopy. Super-resolution and atomic resolution microscopy are also powerful tools for accurately visualizing inter-organelle contacts, although their utilization in contact site analysis is still limited since they require highly dedicated microscopes and technical expertise19. In addition, transmission electron microscopy (TEM), scanning electron microscopy (SEM), and other EM techniques such as electron tomography (ET) and cryo-electron microscopy, are commonly used as they provide high-resolution ultrastructural imaging of the contact sites, which are often impossible to explore using other experimental approaches20,21,22. However, these EM-based methods are a very low-throughput technique that can also be affected by chemical fixation procedures. More recently, proximity labeling-based methods have been used to detect contact sites as well as to identify new contact-site proteins. For example, proximity ligation assay (PLA) has been used to quantify organelle proximities23,24, while a revised version of the ascorbate peroxidase (APEX) assay has been utilized in identifying new contact-site proteins25,26. It is important to recognize that all these methods described above have strengths and intrinsic limitations in detecting the contacts between the organelles. Thus, the pairing of different techniques is required to obtain a thorough interpretation of the organelle contact sites.
Previously we have established the split-Renilla luciferase 8 reassembly assay (split-Rluc assay) to monitor the level of ER-mitochondria membrane contacts (Figure 1A)24,26,27. Briefly, each split half of Renilla luciferase is conjugated with an ER- or mitochondria- targeting sequence. When transfected together, each split half of the enzyme is expressed either in the ER or mitochondrial membrane. When the ER and mitochondria are positioned in close proximity to each other, the split halves come together and reconstitute the whole enzyme with luciferase activity. For the split-Rluc construct, we used Renilla luciferase 8 (Rluc8) in pBAD/Myc-His27 for the initial template. The split site (between amino acids 91 and 92) was determined based on previous reports27. For the N-terminal half of the Rluc8, DNA sequences for amino acids 1-91 of the Rluc8 were fused to the 3' end of the FLAG tag and the mouse AKAP1 mitochondrial-targeting sequence in pcDNA3.1 TOPO vector by PCR27. For the C-terminal half targeted to the ER, DNA sequences that encode amino acids 92-311 were fused to the 5' end of the myc tag and the yeast UBC6 ER localization sequence. Here, we have upgraded the split-Rluc plasmid construct such that split halves of the Renilla luciferase are expressed in a single vector (pCAG) under the same promoter and subsequently cleaved into two fragments as T2A, a self-cleaving peptide 2A sequence from Thosea asigna virus, is inserted in between the two split halves (Figure 1B). The plasmid DNA map and sequences are provided in Supplemental File 1 and Supplemental Figure S1. Using this system, we have measured the effects of three chemicals (inhibiting GTPases involved in actin polymerization) on ER-mitochondria contacts. This split-Rluc assay is a simple but robust assay system for high-throughput screening for inter-organelle contact modulators24.
Protocol
1. Cell maintenance and seeding (Day 1)
- Maintain HEK293T cells in cell culture media containing Dulbecco's Modified Eagle Medium (DMEM) with 10% fetal bovine serum (FBS) (in 100 mm culture dishes) in a humidified incubator at 37 °C with 5% CO2.
- Before starting, check the confluency of the plate by viewing under the microscope. When the cells reach approximately 80-90% confluency, prepare to seed the cells in a 6-well culture plate by removing the media and washing with 10 mL of Dulbecco's phosphate-buffered saline (DPBS).
- Remove the DPBS from the culture dish and treat the cells with 1 mL of 0.05% Trypsin-EDTA phenol red. Incubate for 3 min, then take out the plate from the incubator and add 9 mL of culture media (DMEM with 10% FBS) to stop the trypsin reaction. Slowly pipette up and down to ensure any cells still stuck to the plate are dislodged. Transfer the cell suspension to a 50 mL tube.
- Centrifuge at 300 × g for 5 min at room temperature. Remove the media and resuspend the cell pellets with 1 mL of fresh media (DMEM + 10% FBS).
- Count the number of cells using a hemocytometer.
- Ensure even distribution of cells by gently swirling the tube. Immediately take out 10 µL of the cell suspension and place into a 1.7 mL microcentrifuge tube. Add 90 µL of media to the same tube.
- After mixing the cells and media gently, take the cell suspension and apply 10 µL to each of the chambers of the hemocytometer by gently pipetting underneath the coverslip.
- Using a 10x objective, focus the grid lines of the hemocytometer under a microscope. Once focused, count the number of cells in one set of 16 squares (4 x 4 squares) using a hand tally counter. Repeat until all four sets of 16 squares are counted.
- Calculate the total number of cells/mL by taking the average cell count (from each of 16 corner squares) and multiplying that by 105.
NOTE: Instead of using a hemocytometer to count cells, an automated cell counter can be used according to the manufacturer's instructions.
- Once cell counting is completed, prepare to plate cells in a 6-well plate at 7.5 × 105 cells/well density with 2 mL of media/well. In a 50 mL tube, add the necessary volume of cells along with 13 mL of media. Dispense 2 mL of the diluted cell suspension into each of the 6 wells.
- Before plating, calculate the number of cells necessary for 7.5 × 105 cells/well using C1V1 = C2V2 where C1 is the initial cell count (cell number/mL), V1 is the volume needed from the initial cell suspension, C2 is the desired target cell density (cell number/mL), and V2 is the final volume needed for cell seeding.
- After dispensing, tap the plate gently on all sides to spread the cells and then place it in a humidified incubator at 37 °C with 5% CO2 overnight.
2. Polyethyleneimine (PEI)-mediated cell transfection and post transfection cell seeding (Day 2)
- Remove the 6-well plate from the incubator and aspirate out the existing culture media from each well. Add 2 mL of fresh media per well.
- Transfect the cells with split-Rluc plasmid DNA (pCAG-MitoRlucN-T2A-RlucCER-IRES-mCherry) dissolved in TE buffer (pH 8.0).
- For each well, mix the DNA (750 ng) and PEI (ratio of DNA/PEI = 1:3) in 200 µL of DMEM in a 1.7 mL microcentrifuge tube. Quickly vortex the DNA/PEI at level 8 for approximately 2 s to ensure they mix well together to form complex. After that, spin it down in a mini centrifuge briefly (<3 s).
- Let the mixture of DNA/PEI incubate at room temperature for 15 min and then add the mixture (by dropping it) to the surface of the culture media in the 6-well plate containing cells. Tap the plate gently (to evenly distribute the mixture) and incubate it in a humidified incubator at 37 °C with 5% CO2 for 6 h.
- While incubating, prepare a Poly-D-Lysine (PDL)-coated 96-well plate by first adding 70 µL of PDL (50 µg/mL in DPBS) to each well and incubating in a humidified incubator at 37 °C with 5% CO2 for at least 1 h. Remove the PDL solution and then wash each well 2x with 100 µL of DPBS. At the end of the second wash, ensure that all the remaining DPBS is removed.
- At 6 h post transfection, prepare to seed the transfected cells in a PDL-coated 96-well plate.
- Take out the 6-well plate containing transfected cells from the incubator, aspirate out the media, and wash each well with 2 mL of DPBS. After removing the DPBS, add 350 µL of 0.05% Trypsin-EDTA phenol red and incubate for 1-2 min. To stop the trypsin reaction, add 1.7 mL of media to each well.
- After transferring the cells along with media to a 50 mL tube, spin the tube at 300 × g for 5 min in a tabletop centrifuge. Aspirate the media and resuspend the cell pellet with 1 mL of fresh media. Count the number of cells using a hemocytometer (as described in step 1.5).
- Seed the cells in a PDL-coated 96-well plate at a density of 4 × 104 cells/well in 100 µL of culture media. For this, in a 50 mL tube, add the necessary volume of cells (see below for calculation) along with appropriate volume of media (12 mL total volume). Using a multichannel pipette, dispense 100 µL of the diluted cell suspension into each of the 96 wells. Incubate the cells for 18 h in a humidified incubator at 37 °C with 5% CO2.
- Calculate the number of cells necessary for 4 × 104 cells/well using C1V1 = C2V2 where C1 is the initial cell count (cell number/mL), V1 is the volume needed from the initial cell suspension, C2 is the desired target cell density (cell number/mL), and V2 is the final volume needed for cell seeding.
3. Chemical treatment and luciferase assay in live cells (Day 3)
- In a 1.7 mL microfuge tube, prepare the three fresh solutions of 50 µM Rhosin (50 mM stock in DMSO), 25 µM Ehop-016 (25 mM stock in DMSO), and 50 µM ZCL278 (50 mM stock in DMSO) by diluting each stock solution (50 mM Rhosin, 25 mM Ehop-016, and 50 mL ZCL278; all in DMSO) 1:1,000 in the required amount (50 µL/well × number of wells) of culture media. To each solution, add live cell substrate (50 mM stock in DMSO) for Renilla luciferase diluted 1:2,000 to a final concentration of 25 µM. To make 0.5, 5, and 50 µM ZCL278 solutions, perform serial dilutions using the initial 50 µM ZCL278 in culture media.
NOTE: If the chemical incubation time is desired for less than 1-1.5 h, the cells can be preincubated with live cell substrate (for Renilla luciferase) for 1-1.5 h before they are treated with each chemical. We add chemicals and live cell substrate simultaneously to optimize the protocol for high-throughput drug screening. - Remove the media from each well of the 96-well plate containing transfected cells and add 50 µL of chemical and substrate media mixture to each well.
- After incubating the cells for 1 h, 2 h, or 5 h, take out the culture plate from the incubator, load it onto the luminescence plate reader, and measure luminescence.
- Before starting, ensure that the plate reader is turned on. Access the microplate reader software and create a new file by clicking New Session. Set the temperature of the plate reader to 37 °C by clicking Incubator, checking the temperature option, and typing in 37 °C. Under Plate Layout, choose the option Unknown and specify the wells to measure by clicking and dragging.
- Under Protocol, click Luminescence and keep to the default settings. Once finished with the setup, load the plate with the lid off into the plate reader and choose Run Plate In. Click Start and save file window will appear. Save the file to your desktop by renaming it and clicking Save.
NOTE: The luminescence will continue to be produced for >24 h after the addition of the live cell substrate.
- Once the reading is complete, remove the plate from the plate reader and then click Run Plate In to put the reader back in. Once completed, place the plate back in the humidified incubator (37 °C, 5% CO2) until the next reading.
- When finished, transfer the luminescence data to a graphing program to plot relative luminescence unit (RLU) (y-axis) for each variable (e.g., chemicals or concentrations) (x-axis) and analyze the data.
4. Validation of split-Rluc assay with other methods.
- Perform other assays such as proximity ligation assay (PLA), transmission electron microscopy, and mitochondrial calcium uptake monitoring to validate the results obtained from split-Rluc assay as described previously24.
Representative Results
We have used the protocol described above to measure the level of ER-mitochondria contacts upon the addition of three compounds known to inhibit specific GTPases. CDC42, RHO, and RAC are GTPases that promote actin polymerization28 when activated and are inhibited by ZCL278, Rhosin, and Ehop-016, respectively24. HEK293T cells transfected with split-Rluc were treated with DMSO (control), ZCL278 (50 µM), Rhosin (50 µM), or Ehop-016 (25 µM) and incubated for 1 h, 2 h, and 5 h. Using a plate reader, we measured split-Rluc activity at defined time points (Figure 2).
Rhosin (RHO GTPase inhibitor)-treated cells showed no significant changes in luciferase activity compared to the control DMSO-treated cells at 1 h, 2 h, and 5 h of the treatment (p > 0.9999, p= 0.6956, p > 0.9999) (Figure 2). These results demonstrate that Rhosin does not affect ER-mitochondria contacts, suggesting no involvement of RHO GTPase activity in regulating the contacts between the two organelles.
In contrast, the RAC GTPase inhibitor Ehop-016 showed significantly lower luciferase activity than DMSO at 1 h, 2 h, and 5 h of the treatment (p = 0.0106, p = 0.0009, p = 0.0024) (Figure 2). These data indicate that RAC GTPase activity is required for maintaining normal ER-mitochondria interaction in the cell.
Finally, CDC42 inhibitor ZCL278 showed the most drastic changes in luciferase activity (Figure 2). ZCL278-treated cells showed a significant decrease in luciferase activity compared to the control DMSO and had the lowest p-values through all three time points, maintaining a p < 0.0001 throughout. Our data demonstrate a strong inhibition of ER-mitochondria coupling when CDC42 GTPase activity is blocked by ZCL278, consistent with our previous report using the original split-Rluc assay performed in cells co-transfected with two vectors encoding each half of the split-luciferase separately24.
Given the striking results with ZCL278, especially when compared to the other GTPase inhibitors, ZCL278 activity was further tested at various concentrations [0.5 µM, 5 µM, 50 µM] against a DMSO control. Luciferase activities measured at 1 h, 2 h, and 5 h post treatment showed a dose-dependent response; 0.5 µM of ZCL278 resulted in the weakest effect on luciferase activity, while 5 µM showed stronger, and 50 µM showed the strongest reduction in reconstituted enzyme activity (Figure 3). Together, our results show that ZCL278 downregulates split-Rluc activity in a dose-dependent manner and, as time progresses, higher concentrations of ZCL278 (CDC42 inhibitor) still show a significant change relative to the control DMSO as compared to lower concentrations.
As a way to validate our split-Rluc assay, well-established independent assays, such as proximity ligation assay (PLA), transmission electron microscopy (TEM), and mitochondrial calcium uptake monitoring, can be performed (Figure 4 and data not shown)24. Isoproterenol, a β2 adrenergic receptor agonist, which is one of the compounds identified in our screen for ER-mitochondria contact promoters (Figure 4A), was used to confirm that the luciferase activity of the split-Rluc upon isoproterenol treatment was increased due to enhanced ER-mitochondria contacts as demonstrated by changes in PLA signal intensity as well as mitochondrial calcium uptake (Figure 4B-E).
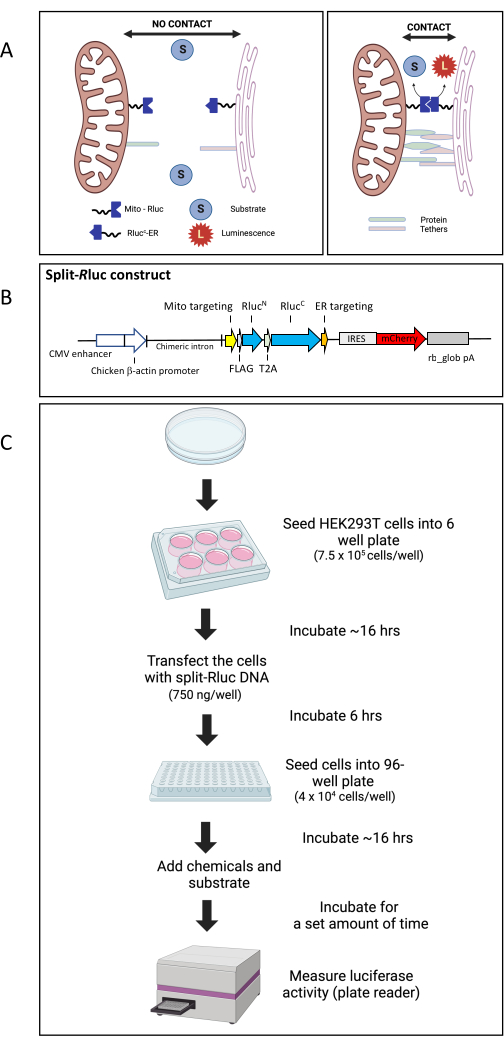
Figure 1: The split-Rluc reassembly assay. (A) Schematic representation of the Split-RLuc assay. Where there are no membrane contacts between the mitochondria and the endoplasmic reticulum, the Mito-RlucN and the RlucC-ER fail to interact and thus there is no Rluc8 activity. However, interaction between the Mito-RlucN and the RlucC-ER, where the ER and mitochondria membranes come into contact, results in full enzymatic activity, which is detected by the conversion of the substrate to luminescence. (B) Schematic map of the split-Rluc DNA construct (pCAG-MitoRlucN-T2A-RlucCER-IRES-mCherry). (C) Diagram of workflow for split-Rluc assay in HEK293T cells. Abbreviations: Mito = mitochondria; ER = endoplasmic reticulum; IRES, internal ribosomal entry site; rb_glob pA, rabbit beta-globin poly adenylation signal; T2A, a self-cleaving peptide 2A sequence from Thosea asigna virus. Please click here to view a larger version of this figure.
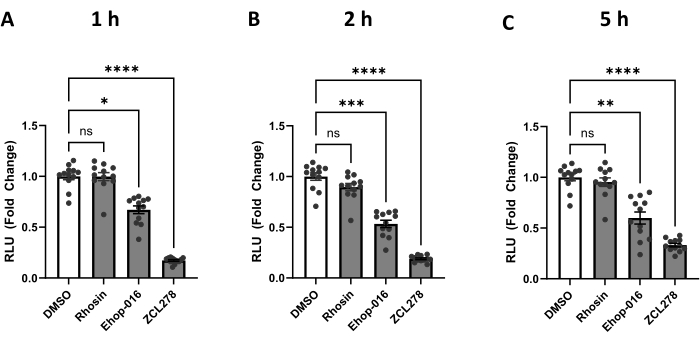
Figure 2: The split-Rluc activities corresponding to the levels of ER-Mito contacts in the cells treated with GTPase inhibitors. (A-C) Reconstituted luciferase activities at (A) 1 h, (B) 2 h, or (C) 5 h after DMSO (control), Rhosin [50 µM], Ehop-016 [25 µM], or ZCL278 [50 µM] treatment (n = 12 for each group). All data were analyzed using one-way ANOVA with multiple comparisons. P values are reported as p > 0.05 (ns), p ≤ 0.05 (*), p ≤ 0.01 (**), p ≤ 0.001 (***), p ≤ 0.0001 (****). Error bars indicate mean ± standard error of measurement. Abbreviations: Mito = mitochondria; ER = endoplasmic reticulum; RLU = relative light unit. Please click here to view a larger version of this figure.
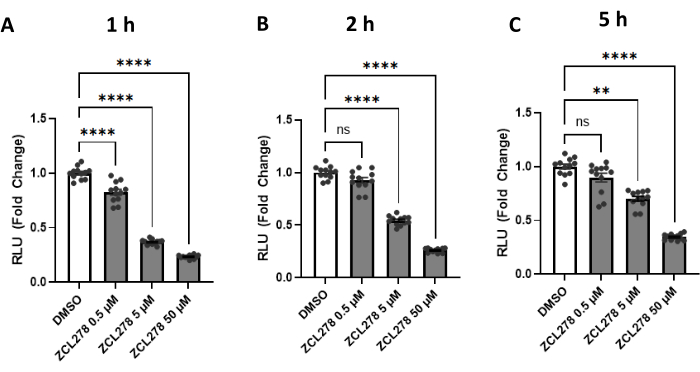
Figure 3: The split-Rluc activities measuring the levels of ER-Mito contacts in the cells treated with ZCL278. (A-C) Reconstituted luciferase activities at (A) 1 h, (B) 2 h, or (C) 5 h after DMSO (control) or ZCL278 at [0.5 µM], [5 µM], or [50 µM] concentration (n = 12 for each group). All data were analyzed using one-way ANOVA with multiple comparisons. P values are reported as p > 0.05 (ns), p ≤ 0.05 (*), p ≤ 0.01 (**), p ≤ 0.001 (***), p ≤ 0.0001 (****). Error bars indicate mean ± standard error of measurement. Abbreviations: Mito = mitochondria; ER = endoplasmic reticulum; RLU = relative light unit. Please click here to view a larger version of this figure.
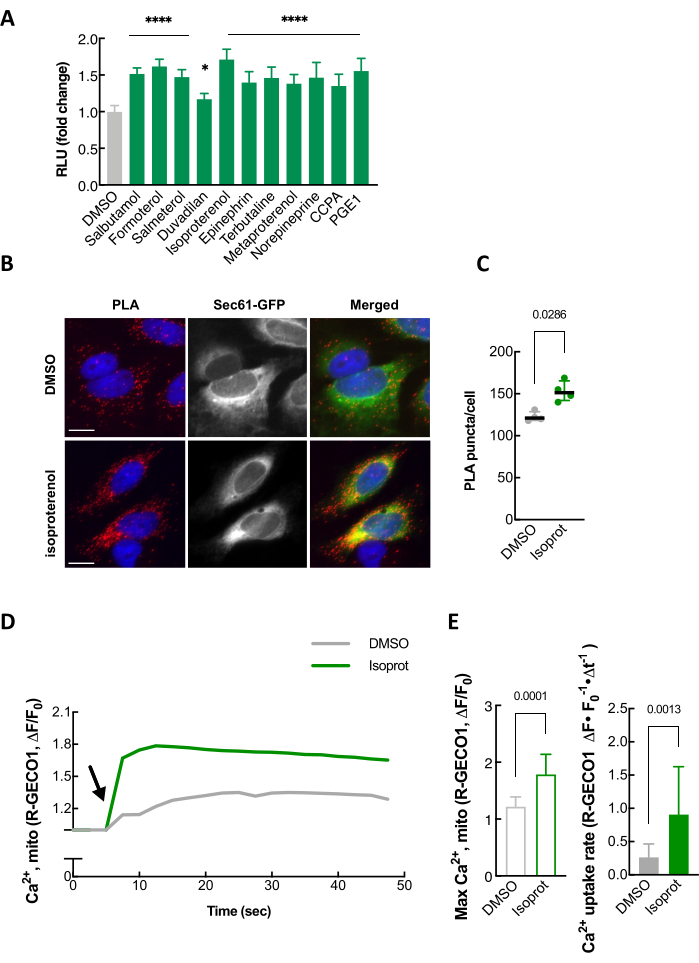
Figure 4: Validation of split-Rluc assay. (A) An example of quantification of split-Rluc activity after treating HEK293T cells with 11 drugs, including isoproterenol, identified in a compound screen for ER-mitochondria contact promoters. (B) An example of proximity ligation assay in HeLa cells that were treated with DMSO or isoproterenol (1 µM). PLA signal (red) indicates a close contact between ER and Mito. Sec61-GFP (green): to label ER; DAPI (blue): nucleus. (C) Quantification of PLA signal in A (n = 4 experiments with 77-97 cells each; Mann-Whitney test). (D) Mitochondrial Calcium uptake in response to histamine (100 µM). Mito-R-GECO1 fluorescent intensity change (average was taken over multiple HeLa cells) plotted every 2.5 s in DMSO- or isoproterenol-treated cells. Arrow indicates histamine addition. (E) Left: Maximum peak of mitochondrial calcium uptake in A. Unpaired two-tailed t-test (n = 10, DMOS; 14, Isoproterenol). Right: Mitochondrial calcium uptake rate (as in A; 7.5 to 12.5 s). Mann-Whitney test (n = 12, DMSO; 11, Isoproterenol). This figure is modified from Lim et al.24. Abbreviations: RLU = relative light unit; PLA = proximity ligation assay. Please click here to view a larger version of this figure.
Table 1: Summary of current techniques for ER-mitochondria contacts. List of advantages and limitations for each method. Please click here to download this Table.
Supplemental File 1: Zip file containing DNA sequence (PDF and GBK formats) of pCAG-MitoRlucN-T2A-RlucCER-IRES-mCherry. Please click here to download this File.
Supplemental Figure S1: Map of pCAG-MitoRlucN-T2A-RlucCER-IRES-mCherry. Please click here to download this File.
Discussion
We have used a split-Renilla luciferase 8 reassembly assay (split-Rluc assay) to quantify the level of ER-mitochondria couplings. In this study, we have modified the original split-Rluc construct24 by generating a single vector, pCAG-MitoRlucN-T2A-RlucCER-IRES-mCherry, encoding each split-Rluc component (MitoRlucN and RlucCER) and a self-cleaving peptide 2A sequence from Thosea asigna virus to ensure the expression of the same amount of each split half of luciferase. As a substrate for the Renilla luciferase, instead of coelenterazine, a modified live cell substrate (protected derivative of coelenterazine; see the Table of Materials) was used for the following reasons. First, unlike coelenterazine which generates autoluminescence in an aqueous environment (especially worse in serum), this protected derivative generates very low autoluminescence (often not detectable) in medium containing 10% serum, thus its signal-to-background ratio is higher than that generated by coelenterazine. Second, differently from coelenterazine, where luminescence is the strongest shortly after adding the substrate but begins to diminish very quickly, luminescence produced by the modified live substrate arrives at its highest point attainable approximately 1.5 h after substrate addition and stays continuous for more than 24 h. These changes in reaction kinetics make it easier to measure luminescence in a multi-well plate, which is very useful for high-throughput screening. Furthermore, the luminescence produced by the protected substrate remaining constant in the long run allows for the measuring of a single set of samples at multiple time points, as opposed to measuring multiple sets of samples used at various time points (single point per sample).
Using this updated version of the split-Rluc construct, we tested if RHO, RAC, and CDC42 GTPases participate in ER-Mito contact formation. We added GTPase inhibitors, Rhosin (RHO inhibitor), Ehop-016 (RAC inhibitor), and ZCL278 (CDC42 inhibitor), to HEK293T cells expressing split-Rluc components. Our assay results indicate that ZCL278 is the most potent inhibitor of ER-mitochondria contact formation and Ehop-016 is the next significant inhibitor, while Rhosin did not affect their contacts. These data suggest that CDC42-mediated actin polymerization (as well as RAC-mediated one to a lesser extent) is required to maintain ER-mitochondria contacts in the cell, while RHO-mediated actin polymerization is probably not important.
To ensure maximum efficiency, there are many critical steps that must be considered for this protocol. First and foremost, the amount of construct transfected must be tested as too much DNA can have a toxic effect on the cells. Second, as the number of cells can have a significant impact on the luciferase levels measured, keeping the density of cells as close as what was stated in the protocol is crucial in ensuring the ideal and consistent 80% confluency of cells by the time of the assay. There may be a reduction in luminescence if the cells reach confluency during the chemical exposure time, as confluent cells make less protein in general. Third, to ensure the cells do not slough off during the addition of chemicals and substrate, we use a 96-well plate pre-coated with Poly-D-Lysine. Fourth, for the compounds (including the substrate) used in this protocol, it is important to keep them at -20 °C or lower (-70 °C) in aliquots and add them to the cells immediately thawed. Lastly, due to possible temperature fluctuations that can affect luciferase activity, it may be best to choose to use the innermost wells of the plate and ignore the wells lining the outside of the plate.
The split-Rluc8 assay offers an easy yet robust method to determine ER-mitochondria contacts, ideal for high-throughput screening; however, there are some limitations that need to be noted. For cell physiology, there is the issue of changes in cell behavior due to the constant overexpression of the construct DNA. Currently, we are working on a construct with an inducible promoter to combat the potential changes in cell physiology due to constant overexpression. Further, when this assay is used for a screening purpose, it should be noted that false positive results can arise from any indirect effect of the drugs on i) expression levels, ii) organelle-targeting efficiencies of the said protein fragments, or iii) enzymatic activities of the reconstituted luciferases. Due to these limitations, this assay must be tested in conjunction with other methods to further validate results. Table 1 refers to various methods of testing these contact sites along with their advantages and disadvantages. We have taken advantage of other methods, including but not limited to proximity ligation assays (PLA), which detect proximity between proteins, and transmission electron microscopy (TEM), which allows for clear visualization of organelle contacts, in conjunction with the split-Rluc assay to validate that the resulting changes in RLU are indeed due to changes in ER-mitochondria contact sites (Figure 4 and Lim et al.24). We have also shown a correlation between alteration in ER-mitochondria contacts and functional parameters such as calcium transfer and mitochondrial fission (Figure 4 and Lim et al.24). To ensure the effect of the drugs on split-Rluc activity is not due to increased expression of split-Rluc gene, a transcription or translation inhibitor can be added together with the drugs as we have done previously24.
For the application of the split-Rluc system in differentiated neurons in culture where the transfection rate is low, viral infection (with lentivirus expressing split-Rluc components) would be an alternative approach. Its use can be extended to create a screening system where the cells (e.g., human induced pluripotent stem cells) are engineered to stably express each component of the split-Rluc, MitoRlucN and RlucCER, either by viral infection or by CRSPR-mediated gene editing. However, for a stable expression system, one must be aware that there could be a selection pressure for cells tolerating the split-Rluc system better, which might not be representative of the normal physiological state. This adaptation or selection could introduce bias in the experimental outcomes.
In summary, despite some of the limitations discussed above, split-Rluc is a fast, straightforward, and powerful assay for high-throughput screening for small molecules or genes regulating the inter-organelle contacts in the cell, as it has been shown in our previous drug screening study24.
Divulgations
The authors have nothing to disclose.
Acknowledgements
The authors are thankful to Dr. Jeffrey Golden (Cedars-Sinai Medical Center) for the critical review of the manuscript. This work was funded in part by the National Institute of Neurological Disease and Stroke (NINDS, R01NS113516).
Materials
| 1.7 mL SafeSeal Microcentrifuge Tube | Sorenson | 16070 | |
| 6-well plate TC Treated | USA Scientific | CC7682-7506 | |
| 10 mL Pipette Tips OneTip | USA Scientific | 1110-3700 | |
| 10 μL pipette tips OneTip | USA Scientific | 1110-3700 | |
| 20-200 μL Beveled tips OneTip | USA Scientific | 1111-1210 | |
| 50 mL Polypropylene Conical Tube | Falcon | 352070 | |
| 96-Well Flipper Microtube Racks | ThermoFisher Scientific | 8770-11 | |
| 96-well plate TC Treated | USA Scientific | CC7682-7596 | |
| 100 mm x 20 mm TC Treated Dish | USA Scientific | CC7682-3394 | |
| 1250 μL Tips OneTip | USA Scientific | 1112-1720 | |
| Centrifuge 5910 Ri – Refrigerated Centrifuge | Eppendorf | 5943000131 | |
| Dimethyl sulfoxide, anhydrous, ≥99.9% | Sigma-Aldrich | 276855-100ML | |
| DMEM, high glucose | ThermoFisher Scientific | 11965092 | |
| DPBS, no calcium, no magnesium | ThermoFisher Scientific | 14190144 | |
| EHop 016 | Bio-Techne Tocris | 6248 | Dissolve in DMSO; store at -70 °C |
| EnduRen Live Cell Substrate | Promega | E6481 | Store aliquots at -70 °C |
| Eppendorf 2-20 μL pipette | Eppendorf | 3123000039 | |
| Eppendorf Research plus 100-1000 μL pipette | Eppendorf | 3123000063 | |
| Eppendorf Research Plus 1-10 µL pipette | Eppendorf | 3123000020 | |
| Eppendorf Research plus 12-channel | Eppendorf | 3125000028 | |
| Eppendorf Research plus 200 μL pipette | Eppendorf | 3123000055 | |
| Fetal Bovine Serum, qualified, USDA-approved regions | ThermoFisher Scientific | 10437028 | |
| Forma Steri-Cycle CO2 Incubator, 184 L, Polished Stainless Steel | ThermoFisher Scientific | 381 | |
| Hand tally counter | Sigma-Aldrich | HS6594 | |
| HEK 293T Cells | ATCC | CRL-3216 | |
| Hemacytometer – Neubauer Bright Line, Double-Counting Chamber | LW Scientific | CTL-HEMM-GLDR | |
| Invitrogen TE Buffer | ThermoFisher Scientific | 8019005 | |
| Microscope | Zeiss | Axiovert 25 CFL | |
| Mini centrifuge | Benchmark Scientific | C1012 | |
| Multi Tube Rack For 50ml Conical, 15ml Conical, And Microcentrifuge Tubes | Boekel Scientific | 120008 | |
| PEI MAX – Transfection Grade Linear Polyethylenimine Hydrochloride (MW 40,000) | Polysciences | 24765-100MG | |
| Pipet-Aid XP | USA Scientific | 4440-0101 | |
| Poly-D-lysine hydrobromide | Sigma-Aldrich | P6407-5MG | |
| Rhosin hydrochloride | Bio-Techne Tocris | 5003 | Dissolve in DMSO; store at -70 °C |
| Trypsin-EDTA (0.05%), phenol red | ThermoFisher Scientific | 25300054 | |
| Varioskan LUX multimode microplate reader | ThermoFisher Scientific | VL0000D0 | |
| Vortex | ThermoFisher Scientific | 2215365 | level 8 |
| VWR Vacuum Aspiration System | VWR | 75870-734 | |
| ZCL 278 | Bio-Techne Tocris | 4794 | Dissolve in DMSO; store at -70 °C |
References
- Aoyama-Ishiwatari, S., Hirabayashi, Y. Endoplasmic reticulum-mitochondria contact sites-emerging intracellular signaling hubs. Front Cell Dev Biol. 9, 653828 (2021).
- Sassano, M. L., Felipe-Abrio, B., Agostinis, P. ER-mitochondria contact sites; a multifaceted factory for Ca2+ signaling and lipid transport. Front Cell Dev Biol. 10, 988014 (2022).
- Scorrano, L., et al. Coming together to define membrane contact sites. Nat Commun. 10 (1), 1287 (2019).
- Voeltz, G. K., Sawyer, E. M., Hajnóczky, G., Prinz, W. A. Making the connection: How membrane contact sites have changed our view of organelle biology. Cell. 187 (2), 257-270 (2024).
- Paillusson, S., et al. There’s something wrong with my MAM; the ER-mitochondria axis and neurodegenerative diseases. Trends Neurosci. 39 (3), 146-157 (2016).
- Sasi, U. S. S., Sindhu, G., Raj, P. S., Raghu, K. G. Mitochondria associated membranes (MAMs): emerging drug targets for diabetes. Curr Med Chem. 27 (20), 3362-3385 (2019).
- Joshi, A. U., Kornfeld, O. S., Mochly-Rosen, D. The entangled ER-mitochondrial axis as a potential therapeutic strategy in neurodegeneration: A tangled duo unchained. Cell Calcium. 60 (3), 218-234 (2016).
- Moltedo, O., Remondelli, P., Amodio, G. The mitochondria-endoplasmic reticulum contacts and their critical role in aging and age-associated diseases. Front Cell Dev Biol. 7, 172 (2019).
- Rieusset, J. The role of endoplasmic reticulum-mitochondria contact sites in the control of glucose homeostasis: an update. Cell Death Dis. 9 (3), 388 (2018).
- Bouguerra, M. D., Lalli, E. ER-mitochondria interactions: Both strength and weakness within cancer cells. Biochim Biophys Acta Mol Cell Res. 1866 (4), 650-662 (2019).
- Tepikin, A. V. Mitochondrial junctions with cellular organelles: Ca2+ signalling perspective. Pflügers Arch. 470 (8), 1181-1192 (2018).
- Masson, G. L., Przedborski, S., Abbott, L. F. A computational model of motor neuron degeneration. Neuron. 83 (4), 975-988 (2014).
- Giamogante, F., Barazzuol, L., Brini, M., Calì, T. ER-mitochondria contact sites reporters: strengths and weaknesses of the available approaches. Int J Mol Sci. 21 (21), 8157 (2020).
- Rizzuto, R., et al. Close contacts with the endoplasmic reticulum as determinants of mitochondrial Ca2+ responses. Science. 280 (5370), 1763-1766 (1998).
- Valm, A. M., et al. Applying systems-level spectral imaging and analysis to reveal the organelle interactome. Nature. 546 (7656), 162-167 (2017).
- Csordás, G., et al. Imaging interorganelle contacts and local calcium dynamics at the ER-mitochondrial interface. Mol Cell. 39 (1), 121-132 (2010).
- Cieri, D., et al. SPLICS: a split green fluorescent protein-based contact site sensor for narrow and wide heterotypic organelle juxtaposition. Cell Death Differ. 25 (6), 1131-1145 (2018).
- Kakimoto, Y., et al. Visualizing multiple inter-organelle contact sites using the organelle- targeted split-GFP system. Sci Rep. 8 (1), 6175 (2018).
- Wu, M. M., Covington, E. D., Lewis, R. S. Single-molecule analysis of diffusion and trapping of STIM1 and Orai1 at ER-plasma membrane junctions. Mol Biol Cell. 25 (22), (2014).
- Csordas, G., et al. Structural and functional features and significance of the physical linkage between ER and mitochondria. J Cell Biol. 174 (7), 915-921 (2006).
- de Brito, O. M., Scorrano, L. Mitofusin 2 tethers endoplasmic reticulum to mitochondria. Nature. 456 (7222), 605-610 (2008).
- Kremer, A., et al. Developing 3D SEM in a broad biological context. J Microsc. 259 (2), 80-96 (2015).
- Söderberg, O., et al. Direct observation of individual endogenous protein complexes in situ by proximity ligation. Nat Methods. 3 (12), 995-1000 (2006).
- Lim, Y., Cho, I. -. T., Rennke, H. G., Cho, G. β2-adrenergic receptor regulates ER-mitochondria contacts. Sci Rep. 11 (1), 21477 (2021).
- Lam, S. S., et al. Directed evolution of APEX2 for electron microscopy and proximity labeling. Nat Methods. 12 (1), 51-54 (2014).
- Cho, I. -. T., et al. Ascorbate peroxidase proximity labeling coupled with biochemical fractionation identifies promoters of endoplasmic reticulum-mitochondrial contacts. J Biol Chem. 292 (39), 16382-16392 (2017).
- Lim, Y., Cho, I. -. T., Schoel, L. J., Cho, G., Golden, J. A. Hereditary spastic paraplegia-linked REEP1 modulates endoplasmic reticulum/mitochondria contacts. Ann Neurol. 78 (5), 679-696 (2015).
- Arnold, T. R., Stephenson, R. E., Miller, A. L. Rho GTPases and actomyosin: Partners in regulating epithelial cell-cell junction structure and function. Exp Cell Res. 358 (1), 20-30 (2017).

.
