단일 분자의 구조 정보는 고속 나노 측위 시스템을 사용하여 실험을 FRET
Summary
We present the setup and experimental procedure to obtain smFRET data from large donor-acceptor networks with a TIRF microscope. The step-by-step analysis of these measurements with the Bayesian inference software Fast-NPS yields high-resolved structural information via the application of adapted dye models.
Abstract
Single-molecule Förster Resonance Energy Transfer (smFRET) can be used to obtain structural information on biomolecular complexes in real-time. Thereby, multiple smFRET measurements are used to localize an unknown dye position inside a protein complex by means of trilateration. In order to obtain quantitative information, the Nano-Positioning System (NPS) uses probabilistic data analysis to combine structural information from X-ray crystallography with single-molecule fluorescence data to calculate not only the most probable position but the complete three-dimensional probability distribution, termed posterior, which indicates the experimental uncertainty. The concept was generalized for the analysis of smFRET networks containing numerous dye molecules. The latest version of NPS, Fast-NPS, features a new algorithm using Bayesian parameter estimation based on Markov Chain Monte Carlo sampling and parallel tempering that allows for the analysis of large smFRET networks in a comparably short time. Moreover, Fast-NPS allows the calculation of the posterior by choosing one of five different models for each dye, that account for the different spatial and orientational behavior exhibited by the dye molecules due to their local environment.
Here we present a detailed protocol for obtaining smFRET data and applying the Fast-NPS. We provide detailed instructions for the acquisition of the three input parameters of Fast-NPS: the smFRET values, as well as the quantum yield and anisotropy of the dye molecules. Recently, the NPS has been used to elucidate the architecture of an archaeal open promotor complex. This data is used to demonstrate the influence of the five different dye models on the posterior distribution.
Introduction
생체 분자의 구조를 결정하는 것은 그 기능을 이해하기위한 중요한 전제 조건이다. 구조 결정을위한 두 개의 잘 확립 된 방법은 극저온 전자 현미경 및 X 선 결정학 (1, 2)이다. 오늘, 두 가지 방법은 옹스트롬 수준 아래로 해상도를 가진 고해상도 구조 정보를 제공합니다. 이러한 두 가지 방법은 단백질 복합체 같은 큰 생 분자의 구조를 규명하기 위해 광범위하게 사용되고있다. 기존의 방법은 지속적 지난 수십 년에 걸쳐 개선되어 왔지만, 큰 동적 과도 복합체 3을 조사하는 경우, 생물학적 구조의 복잡성은 여전히, 특히 구조 생물학에 중요한 도전을 제기.
고분자 복합체의 역학, 특히 구조 – 기능 관계를 연구하기 위해서, 단일 분자 방법론 잠을ided 유용한 정보 4. 여러 새로운 전략 구조 및 동적 정보를 획득하는 방법에 직교 제공 개발되었다. 예 고속 AFM (5) 기계적 조작 6 지역화 형광 현미경 (7)뿐만 아니라, 단일 분자 포스터 공명 에너지 전이 (smFRET) (8, 9)이다. FRET 매우 이른 이후에 의한 생체 거대 분자 (10)의 길이 규모의 거리에 의존하는 분자자를 지칭되었다.
smFRET의 한 특히 흥미로운 애플리케이션은 추론 smFRET 측정으로부터 얻어진 거리 정보를 사용하는 정보는 11, 12, 13, 14, 15의 구조 </sup> 16, 17, 18, 19, 20, 21, 22, 23. smFRET 인해 높은 시간 분해능으로, 단백질 구조의 이동 부분의 위치는 국소 수있다. 그러나, 염료 분자에 대한 smFRET 데이터 중요한 보정 파라미터로부터 정량적 정보를 추출하기 위해 측정을 24 동안 결정되어야한다. 이들 보정 계수로, FRET 효율 E FRET은 공식을 사용하여 계산 될 수있다
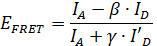 ,
,
어디 A와 I D </sUB는> 각각 공여체 및 수용체 분자의 형광 강도 (도 2 참조)이다. β-인자 수용체 채널에 크로스 – 토크에 대한 도너 발광 누설 계정에 의해 계산된다

나는 'A와 나'D 경우 기증자의 형광 강도와 수용체 분자의 사진 표백 후 수용체 분자이다.
γ 인자는 두 채널의 검출 상대 효율의 차이뿐만 아니라 도너의 형광 양자 수율 셉터 염료의 차이를 보정한다. 그것은으로 모든 개인 시간 추적 계산한다
<p class="jove_content" fo:keep-together.within-page="1" fo:text-align="center" style = "텍스트 정렬 : 센터;">
이 설명은 종종 중요뿐만 아니라 보정 될 필요 셉터 분자의 직접적인 자극을 무시한다는 참고. 이러한 보정 인자를 결정하는 것은 광 신체적 변화 및 구조 역학 구별하기 위해 교류 방식 (25)에 도너뿐만 아니라 수용체 모두를 여기 시키는데 유용하다.
정량적 smFRET 효율뿐만 아니라 양적 구조 정보를 얻을뿐만 아니라 위해, 나노 포지셔닝 시스템 (NPS)는 2008 (26)에 도입 하였다. 이름은 위성 기반의 위성 위치 확인 시스템 (GPS)과의 유사성을 기준으로 선택되었다. 국민 연금은 biomacromolecular 단지에서 알 수없는 염료 위치의 현지화를 위해 smFRET 및 X 선 결정학 데이터를 결합하는 하이브리드 기술이다. 는 Crystal 구조는 기준 프레임으로서 작용하고 smFRET 결과를 알 수없는 위치 형광 (안테나)과 결정 구조 (위성)로부터 공지 된 위치 사이의 거리 정보를 획득하기 위해 사용된다. 연속적인 실험에서 안테나와 여러 개의 위성들 사이의 거리가 측정되고, 상기 안테나의 위치는 베이지안 파라미터 추정에 기초하여 통계적으로 엄격한 분석 기법에 의해 결정된다. 결과적으로, 상기 안테나의 위치를 가장 가능성뿐만 계산되지만 완전한 3D 불확실성 분포 소위 후방은 신뢰할 볼륨 시각화. 더욱이, NPS smFRET 완전한 네트워크 (27)의 분석을 허용하도록 확장되었다.
NPS는 진핵 전사 중요한 질문 상류 DNA, 비 – 주형 DNA 및 RNA 중합 효소 II 신장 공동 내의 초기의 mRNA 즉 과정들을 해결하기 위하여 사용되어왔다또한 전사 개시의 효과를 보여주는 mplex 12, 28, 26 요인 29 오픈 프로모터의 동적 구조는 복잡. 더욱이, NPS는 전사 연장 인자 Spt4 / 5 (31)와 동일한 위치에 경쟁적으로 결합하는 전사 개시 인자 TFE의 위치는 고세균 RNA 폴리머 라제 오픈 착체 (30)의 특히 구조를 규명 하였다.
이후 smFRET 기반 구조 방식의 수는 15, 18, 21, 23을 발표 하였다. 다른 smFRET 기반 구조 방법을 비교하면,이 방법의 정밀도 명백한 색소 모델의 특정 선택에 크게 의존하는 것이 분명해진다. 하나는주의해야염료 분자는 지역 환경에 따라 서로 다른 공간과 배 양성 문제가 발생할 수 있습니다.
이를 위해, 빠른 NPS는 32 도입되었다. 고속 NPS 크게 계산 시간을 감소시키는 향상된 샘플링 알고리즘을 사용한다. 또한, 고속 NPS는 하나의 구조 분석을 수행하고 각 염료 분자의 사용자는 다음에 설명한다 다섯 가지 염료 모델 세트를 선택할 수 있습니다. 고전 불리는 가장 보수적 모델 염료는 하나이지만 알려지지 않은 위치를 차지하는 것으로 가정한다. 이 위치에서, 형광 물질은 크기가 각각의 (시간에 따른) 형광 이방성 결정하는 콘에서 자유롭게 회전 할 수있다. 원뿔의 방향으로 측정 된 거리 smFRET 효율 변환시 큰 불확실성에 이르게하는 공지되지 않는다. 그것은 다른 염료 모드에 비해 작은 정밀도로 이끌 것이기 때문에 이러한 점에서, 모델은, 보수적LS. 매우 짧은 거리를해야 띄게 잘못된 위치 결정에 클래식 모델 리드에 의해 가정. 전형적인 smFRET 값의 경우, 정확한 위치는 항상 비교적 큰 신뢰할 수있는 볼륨에 묶여 있습니다.
보다 정밀 바람직하므로 그러나, 개발 및 정밀도를 향상시키기 위해 도움이 될 다른 염료 모델을 시험하는 것이 중요하다. 염료 고유의 형광 수명보다 빠르게 회전하면, 소위 ISO 모델을 적용 할 수있다. 여기서, 2 κ 배향 계수 (특성 등방성 포스터 반경을 계산하기 위해 필요한  ) 2/3로 설정됩니다. 전형적인 모델 (32)에서와 비교하여 그 결과 계산 신뢰할 볼륨은 작은 크기의 약 2 명령이다. 빠른 reori뿐만 있도록 형광이있는 환경에서 발견되는 경우에모든 액세스 볼륨 이상이 정해져있는,하지만 또한 빠른 동작은 meanpos 이소 모델이 사용되어야한다. 이 모델에서, 상기 염료는 효과적으로 공간적 평균화 다항식 거리 변환 (15)에 의해 차지되는 평균 하나의 위치를 차지한다. 예를 들어, (일반적으로 소수성) 염료는 친수성 영역, 예를 들면, DNA에 연결되어있는 경우,이 모델이 적용됩니다. meanpos 이소 모델의 적용은 약 2의 팩터에 의해 신뢰할 볼륨의 크기를 더욱 감소 리드. 그러나, 단백질에 결합 염료는 입체적 액세스 볼륨 (AV)의 몇몇 소수성 패치에 가역적으로 결합 할 수 있습니다. 순간적으로이 지역 사이에서 전환하지만, 한 지역이 자유 회전을 겪게하고 내 빠른 현지화 움직임이 가장 VAR-meanpos 이소 모델에 의해 설명되는 형광. 비슷한 상황에있는 염료는 모델이 적용되는 VAR-meanpos을 자유롭게 회전 할 수 없습니다. 더 라 이 모델에 대한 etails는 우리의 최근 출판물 (32)에서 찾을 수 있습니다.
) 2/3로 설정됩니다. 전형적인 모델 (32)에서와 비교하여 그 결과 계산 신뢰할 볼륨은 작은 크기의 약 2 명령이다. 빠른 reori뿐만 있도록 형광이있는 환경에서 발견되는 경우에모든 액세스 볼륨 이상이 정해져있는,하지만 또한 빠른 동작은 meanpos 이소 모델이 사용되어야한다. 이 모델에서, 상기 염료는 효과적으로 공간적 평균화 다항식 거리 변환 (15)에 의해 차지되는 평균 하나의 위치를 차지한다. 예를 들어, (일반적으로 소수성) 염료는 친수성 영역, 예를 들면, DNA에 연결되어있는 경우,이 모델이 적용됩니다. meanpos 이소 모델의 적용은 약 2의 팩터에 의해 신뢰할 볼륨의 크기를 더욱 감소 리드. 그러나, 단백질에 결합 염료는 입체적 액세스 볼륨 (AV)의 몇몇 소수성 패치에 가역적으로 결합 할 수 있습니다. 순간적으로이 지역 사이에서 전환하지만, 한 지역이 자유 회전을 겪게하고 내 빠른 현지화 움직임이 가장 VAR-meanpos 이소 모델에 의해 설명되는 형광. 비슷한 상황에있는 염료는 모델이 적용되는 VAR-meanpos을 자유롭게 회전 할 수 없습니다. 더 라 이 모델에 대한 etails는 우리의 최근 출판물 (32)에서 찾을 수 있습니다.
이 모델은 특히 염료 발생할 수있는 다양한 환경을 설명하고이를 적용하는 것은 현명 현지화 정밀도를 최적화에 광범위한 레퍼토리를 제공합니다. 고속 NPS의 특정 위치에 부착 된 각 염료 분자는 FRET-파트너가 서로 다른 모델이 허용되도록, 각각의 모델에 할당 될 수있다. 이 무한한 및 확대에 자연을 모델링 할 수 있습니다. 그러나, 하나의 최종 모델의 조합에 의해 얻어진 결과는 실험 데이터와 일치 여전히되도록 엄격한 통계 학적 테스트를 수행하는 것이 중요하다. 이 시험은 빨리 NPS 소프트웨어에 포함되어 있습니다.
실험 데이터에 고속 NPS를 적용하기 위해 (단지) 세 개의 입력 변수의 측정이 필요하다. 우선, 색소 쌍 특정 등방성 포스터 반경 (/54782/54782eq5.jpg "/>)를 정의 할 필요가 도너 염료. 따라서, 양자 수율 (QY) 상기 도너 형광 발광 스펙트럼과 수용자의 흡수 스펙트럼을 측정 할 필요가있다. 이러한 측정은 수행 될 수있다 표준 분광기 및 표준 형광 분광기를 사용하여 대량. 각각의 쌍의 경우, R 0은 프리웨어 PhotochemCAD을 사용하여 계산되고, NPS 분석에 사용될 수있다. 또한, 염료 분자 (시간 – 분해) 형광 이방성 필요 편광 (시간)에 민감한 형광 분광기를 사용하여 얻을 수있다. 그러나, 고속 NPS 가장 중요한 입력 파라미터는 전반사 형광 현미경 (TIRFM)와 같은 단일 분자 형광 현미경 설정에서 측정 smFRET 효율이다 .
여기서는 smFRET 데이터를 획득하고 고속 NPS (도 1)를 적용하는 단계별 프로토콜을 제시한다.
Protocol
Representative Results
Discussion
우리는 정확하게 즉 생체 거대 분자, 핵산 및 / 또는가요 성 단백질 링커를 통해 부착 염료 간의 FRET 효율성을 결정하기 위해 설정 및 실험 절차를 제시한다.
smFRET 정확한 측정 (제 3)을 보장하기 위해, 측정하는 동안 언제든지 유동 챔버로부터 공기를 배제하는 것이 중요하다. 또한, 형광체와 유량 용기를 너무 많이 넣지해야합니다. 형광 명확하게 정확한 분석을 위해 구분해야합니다. 도너의 표백 보이지 않는다 smFRET 쌍은 분석에서 제외되어야 할 바와 같이, 시야에서 분자의> 80 %가 영화의 끝에 표백 있는지 확인. 도너 및 억 셉터 채널의 크로스 토크 (cross-talk)와 상대 검출 효율 보정 샘플 β 인자 및 γ 이중의 불균일성을 고려하여 각각마다 개별적으로 계산 페어링 FRET.
<p class= "jove_content는"> 카메라 설정 (통합 시간, 전자 증 이득 전치 증폭기 이득 및 3.9 절에 설명 된 판독 레이트) 잡음비, 동적 범위 및 시간 해상도로 신호의 최적 균형을 제공하는 값으로 설정되어야한다. 서로 다른 실험하거나 상이한 하드웨어를 사용하는 경우 재조정 될 필요가있다. 프레임 번호는 도너 대부분 관측 시간 내에 표백 분자되도록 충분히 높게 할 필요가있다.형광 분광기 (7 항 내지 9)의 측정에 대한 신호 강도 및 기록 된 데이터의 스펙트럼 해상도 간의 절충이 발견되어야한다. 이를 위해 형광 분광계의 여기 및 방출 통로의 슬릿은 사용되는 장비 및 시료 농도에 따라 적응되어야한다.
또한, 우리는 과도 또는 동적 macrom의 구조 정보를 얻기 위해 고속 NPS 분석 방법을 제시olecular 단지. NPS는 비 템플릿 DNA 가닥의 경로 및 고세균 RNA 폴리머 라제 오픈 복잡 전사 개시 인자의 위치를 나타 내기 위해 적용되었다. 60 개 이상의 서로 다른 거리 측정의 네트워크를 사용하여, 우리는 새로 구현 샘플링 엔진 (준비 EILERT, T., 베 커스, M., DRECHSLER, F., 미카엘, J.)가 장착 빨리 NPS, 보여 주었다 원래 글로벌 NPS 방법 (27)에 비해 크기 ≈2 수주 복잡한 smFRET 네트워크 분석에 필요한 시간을 감소시킨다. 이 알고리즘의 견고성은 병렬 템퍼링 방식과 결합 된 대도시 – 내 – 깁스 샘플러에 뿌리를두고있다. 빠른 NPS 네트워크 결과의 정확한 재현성을 보여줍니다 이전 30 게시 된 결과와 일치한다.
몇 가지 다른 방법이 smFRET 측정 (11)로부터 구조 정보를 추론하는 것을 목표로 발표되었다 </s업> 12, 13, 14, 15, 16, 17, 18. 이들 접근법 모두는 하나의 특정 색소 모델을 제공한다. 따라서, 각각의 모델에서 가정을 만족하지 않는 염료가 사용될 거짓 구조 정보 발생할 수 없다. 빠른 NPS는 반대로, 각 염료 분자 다른 모델을 선택할 수 있습니다. 이 두 가지의 구조적인 동작을 설명하는 데 도움이 염료 분자 자체뿐만 아니라 그것의 부착을 위해 사용되는 링커. 상기 염료 분자의 분자량 로컬 환경뿐 아니라 물리적 특성에 가장 적합한 어느 모델을 결정한다.
고세균 개시 복합체의 분석 smFRET 네트워크의 모든 염료 분자 등방성 가정이 급격히 감소 나 리드n은 신뢰할 볼륨의 크기는 고전 모델에 비해. 모든 염료에 대한 평균 동적 위치와 함께 (95 %에서) 모든 신뢰할 볼륨 크기의 중앙값을 분자보다 0.5 내지 3으로 감소시킨다. 그러나 이러한 염료 분자의 포스 테리어는 가정이 잘못된 구조 정보에 리드를 한 것으로 표시, 그들의 smFRET 측정과 더 이상 일치하지 않습니다. 대조적으로, 전형적인 모델에서 결정된 포스 테리어 결정된 smFRET 효율과 일치한다.
모든 염료에 대한 평균 등방성 및 / 또는 동적 위치의 가정으로 빨리 NPS 각 염료 다섯 가지 모델 중 하나를 할당 할 수있는 염료 분자 전과을 가능하게 불일치로 이어집니다. 각 모델은 동일한 액세스 볼륨을 사용한다. 염료 AVS의 계산 알고리즘은 몇 가지 가정한다. 우선, 형광체의 공간적 형상은 구형으로 근사된다. 따라서, 직경은 계정 형광의 WID 고려일, 높이와 두께 (제 12 조)을 사용해야합니다. 또한, 상기 링커의 형태는가요 성로드에 의해 근사된다. 제 12 항에 제시된 값은 12 C 링커를 통해 부착 염료 알렉사 647에 대해 계산 하였다. 지금까지, 이는 실험 형상 주어진 정확하게 모델이 가장 적합 선험적으로 결정하는 것은 불가능하고, 따라서 모든 모델 시험해야한다. 여전히 데이터와 일치하면서 일반적으로, 하나는 가장 작은 가능한 후방 크기를 제공하는 모델을 선택한다. 모델 선택은 smFRET 데이터와 일치하는지 여부를 테스트하기 위해 사후 우도를 모두 계산한다. 일관성 후방에서 수집 한 샘플의 90 % 이상이 우도의 95 % 신뢰 구간 내에있는 것을 의미한다.
그것이 사실이지만 염료 분자 smFRET 네트워크 거리 불확실성이 작을 이방성 하부 기하학적 배열도 고려 될 필요가있다. 따라서, 잠시 연구전형적인 첫번째 선택은 ISO 모델 낮은 형광 이방성 염료 분자이다 epresenting, 일관성 검사는 올바른 염료 모델을 선택하기위한 직접적인 수단을 제공한다. 염료 모델의 최적 선택은 위치 파악 정확성의 급격한 증가를 야기 함과 동시에 그 FRET 데이터 네트워크의 일관성을 유지할 수있다.
요약하면, 빨리 NPS는 큰 거대 분자 복합체의 구조 및 동적 정보를 얻을 수 있습니다. 이러한 X 선 결정학 또는이 따라서 매우 복잡한 생물학적 과정의 기전을 이해 우리 넓어 높은가요 또는 과도 복합체를 모니터링 할 수 극저온 전자 현미경 등의 일반적인 구조 방법 대조적.
Declarações
The authors have nothing to disclose.
Acknowledgements
The authors thank B. Gruchmann for the mechanical drawings of the flow chamber. Further, we want to express our gratitude to Max Beckers and Florian Drechsler for insightful comments and discussions regarding NPS and the underlying sampling engine.
Materials
| Flowchamber preparation | |||
| Customized metall sample holder | self-built | n/a | |
| quartz-glass slides, 76 x 26 mm | Technical Glass Products | 26007 | |
| coverslips, 60 x 24 mm | Marienfeld | 101242 | |
| detergent, Hellmanex II | Hellma | 320.001 | |
| ultra-pure water from Synergy UV | Millipore | 2512600 | |
| Zepto plasma cleaner | Diener | n/a | |
| (3-aminopropyl)-triethoxysilane, p.a. | Sigma-Aldrich | A3648 | |
| methoxy PEG-succinimidyl valerate, 5 kDa | Laysan Bio Inc. | MPEG-SVA-5000-1g | |
| biotinylated PEG-succinimidyl valerate, 5 kDa | Laysan Bio Inc. | BIOTIN-PEG-SVA-5000 | |
| Sodium biocarbonate | Sigma-Aldrich | S5761 Sigma | |
| Sodium carbonate | Sigma-Aldrich | S2127 Sigma-Aldrich | |
| sealing film (Nescofilm) | Fisher Scientific | 12981805 | |
| Tygon Flexible Silicone Tubing, 0.8 mm ID, 2.4 mm OD | Saint-Gobain Performance Plastics | 720958 | |
| Fine-Bore Polyethylene Tubing, 0.58 mm ID, 0.96 mm OD (Smiths Medical) | Fisher Scientific | 12665497 | |
| Neutravidin | Life Technologies | A2666 | |
| Name | Company | Catalog Number | Comments |
| Total internal reflection fluorescence microscope | |||
| Nd:YAG Laser, 532 nm | Newport Spectra-Physics | EXLSR-532-100-CDRH | |
| diode-pumped solid-state laser, 491 nm, Calypso | Cobolt | 904010050 | |
| diode laser 643 nm, iBeam smart | Toptica | iBEAM-SMART-640-S | |
| dichroic mirror, 532 RDC | Chroma | F33-540 | |
| dichroic mirror, 476 RDC | Chroma | F33-476z | |
| acousto-optic tunable filter | AA Opto-Electronic | AOTFnC-VIS | |
| plano-convex cylindrical lens, f = 75 mm | Thorlabs | LJ1703L1-A | |
| plano-concave cylindrical, f = -300 mm | Thorlabs | ||
| prism, PS 991 | Thorlabs | PS991 | |
| focussing lens, f = 75 mm | Thorlabs | LA1608-B | |
| syringe pump, PHD 2000 | Harvard Apparatus | 70-2002 | |
| 2 stepper motors, Z812B | Thorlabs | Z812B | |
| piezoelectric actuator, PE4 | Thorlabs | PE4 | |
| IR diode laser | Edmund Optics | CPS808 | part of the autofocus system |
| dichroic mirror, 775 DCXR | Chroma | 775 DCXR | |
| position-sensing detector (PSD), PDP90A | Thorlabs | PDP90A | part of the autofocus system |
| water-immersion objective, Plan Apo 60X WI, NA 1.2 | Nikon | MRD07601 | |
| dichroic mirror, 645 DCXR | Chroma | 645 DCXR | part of the emission pathway |
| emission filter, 3RD550-510 | Omega Optical | 3RD550-510 | green channel in the emission pathway |
| emission filter, 3RD660-760 | Omega Optical | 3RD660-760 | red channel in the emission pathway |
| EMCCD camera, iXon+ DU897EBV | Andor | AND-20-00032 | |
| EMCCD camera, iXon3 DU897D-BV | Andor | AND-20-000141 | |
| Name | Company | Catalog Number | Comments |
| Miscellaneous | |||
| Varian 50 | Cary | UV-VIS spectrometer | |
| Fluorolog2 | SPEX | fluorescence spectrometer | |
| Solis (V4.15) | Andor | control software for the EM-CCD camera | |
| Apt user utility (V1.022) | Thorlabs | control software for the piezo-motors | |
| Norland Optical Adhesive 68 | Thorlabs | adhesive | |
| PC-AFN-0.8 Nile red | Kisker Biotech | avidin-coated fluorescent multispec beads | |
| Matlab | Mathworks | technical computing language for custon written software | |
| Origin (V9.0) | Originlab | scientific graphing and data analysis software | |
| Hellma 105-202-15-40 | Hellma | 105-202-15-40 | absorption cuvette of 1 cm path length |
| Hellma 105-251-15-40 | Hellma | 105-251-15-40 | fluorescence cuvette with 3 mm path length |
Referências
- Cheng, Y. Single-Particle Cryo-EM at Crystallographic Resolution. Cell. 161, 450-457 (2015).
- Garman, E. F. Developments in X-ray Crystallographic Structure Determination of Biological Macromolecules. Science. 343 (6175), 1102-1108 (2014).
- Sali, A. Outcome of the First wwPDB Hybrid/Integrative Methods Task Force Workshop. Structure. 23, 1156-1167 (2015).
- Hopfner, K. P., Michaelis, J. Mechanisms of nucleic acid translocases: lessons from structural biology and single-molecule biophysics. Curr Opin Struct Biol. 17, 87-95 (2007).
- Ando, T., Uchihashi, T., Kodera, N. High-speed AFM and applications to biomolecular systems. Annu Rev Biophys. 42, 393-414 (2013).
- Neuman, K. K. C., Nagy, A. Single-molecule force spectroscopy: optical tweezers, magnetic tweezers and atomic force microscopy. Nat Methods. 5, 491-505 (2008).
- Yildiz, A. Myosin V walks hand-over-hand: single fluorophore imaging with 1.5-nm localization. Science. 300 (5628), 2061-2065 (2003).
- Joo, C., Balci, H., Ishitsuka, Y., Buranachai, C., Ha, T. Advances in Single-Molecule Fluorescence Methods for Molecular Biology. Annu Rev Biochem. 77 (1), 51-76 (2008).
- Hohlbein, J., Craggs, T. D., Cordes, T. Alternating-laser excitation: single-molecule FRET and beyond. Chem Soc Rev. 43 (4), 1156-1171 (2014).
- Stryer, L., Haugland, R. P. Energy transfer: a spectroscopic ruler. Proc Natl Acad Sci U S A. 58 (2), 719-726 (1967).
- Rasnik, I., Myong, S., Cheng, W., Lohman, T. M., Ha, T. DNA-binding Orientation and Domain Conformation of the E. coli Rep Helicase Monomer Bound to a Partial Duplex Junction: Single-molecule Studies of Fluorescently Labeled Enzymes. J Mol Biol. 336 (2), 395-408 (2004).
- Andrecka, J. Single-molecule tracking of mRNA exiting from RNA polymerase II. Proc Natl Acad Sci U S A. 105 (1), 135-140 (2008).
- Schröder, G. F., Grubmüller, H. FRETsg: Biomolecular structure model building from multiple FRET experiments. Comput Phys Commun. 158 (3), 150-157 (2004).
- Margittai, M. Single-molecule fluorescence resonance energy transfer reveals a dynamic equilibrium between closed and open conformations of syntaxin 1. Proc Natl Acad Sci U S A. 100 (26), 15516-15521 (2003).
- Kalinin, S. A toolkit and benchmark study for FRET-restrained high-precision structural modeling. Nat Methods. 9 (12), 1218-1227 (2012).
- Choi, J. N6-methyladenosine in mRNA disrupts tRNA selection and translation-elongation dynamics. Nat Struct Mol Biol. 23 (August 2015), 110-115 (2015).
- Svensson, B. FRET-based trilateration of probes bound within functional ryanodine receptors. Biophys J. 107 (9), 2037-2048 (2014).
- Stephenson, J. D., Kenyon, J. C., Symmons, M. F., Lever, A. M. L. Characterizing 3D RNA structure by single molecule FRET. Methods. (2016), 1-11 (2016).
- Lee, N. K. Accurate FRET measurements within single diffusing biomolecules using alternating-laser excitation. Biophys J. 88 (4), 2939-2953 (2005).
- McCann, J. J., Choi, U. B., Zheng, L., Weninger, K., Bowen, M. E. Optimizing methods to recover absolute FRET efficiency from immobilized single molecules. Biophys J. 99 (3), 961-970 (2010).
- Brunger, A. T., Strop, P., Vrljic, M., Chu, S., Weninger, K. R. Three-dimensional molecular modeling with single molecule FRET. J Struct Biol. 173, 497-505 (2011).
- Schuler, B. Single-molecule FRET of protein structure and dynamics – a primer. J nanoboitechnology. 11, 1-17 (2013).
- Choi, U. B. Single-molecule FRET-derived model of the synaptotagmin 1-SNARE fusion complex. Nat Struct Mol Biol. 17 (3), 318-324 (2010).
- Dale, R. E., Eisinger, J., Blumberg, W. E. The orientational freedom of molecular probes. The orientation factor in intramolecular energy transfer. Biophys J. 26 (2), 161-193 (1979).
- Kapanidis, A. N. Alternating-laser excitation of single molecules. Acc Chem Res. 38 (7), 523-533 (2005).
- Muschielok, A. A nano-positioning system for macromolecular structural analysis. Nat Methods. 5 (11), 965-971 (2008).
- Muschielok, A., Michaelis, J. Application of the nano-positioning system to the analysis of fluorescence resonance energy transfer networks. J Phys Chem B. 115 (41), 11927-11937 (2011).
- Andrecka, J. Nano positioning system reveals the course of upstream and nontemplate DNA within the RNA polymerase ii elongation complex. Nucleic Acids Res. 37 (17), 5803-5809 (2009).
- Treutlein, B. Dynamic Architecture of a Minimal RNA Polymerase II Open Promoter Complex. Mol Cell. 46 (2), 136-146 (2012).
- Nagy, J. Complete architecture of the archaeal RNA polymerase open complex from single-molecule. FRET and NPS. Nat Commun. 6, 6161 (2015).
- Grohmann, D., et al. The Initiation Factor TFE and the Elongation Factor Spt4/5 Compete for the RNAP Clamp during Transcription Initiation and Elongation. Mol Cell. 43 (2), 263-274 (2011).
- Beckers, M., Drechsler, F., Eilert, T., Nagy, J., Michaelis, J. Quantitative structural information from single-molecule FRET. Faraday Discuss. 184, 117-129 (2015).
- Bennink, M. L. Unfolding individual nucleosomes by stretching single chromatin fibers with optical tweezers. Nat Struct Biol. 8 (7), 606-610 (2001).
- Chandradoss, S. D. Surface passivation for single-molecule protein studies. J Vis Exp. (86), e50549 (2014).
- Würth, C., Grabolle, M., Pauli, J., Spieles, M., Resch-Genger, U. Relative and absolute determination of fluorescence quantum yields of transparent samples. Nat Protoc. 8 (8), 1535-1550 (2013).
- Lakowicz, J. R. . Principles of Fluorescence Spectroscopy. , (2006).
- Korkhin, Y. Evolution of complex RNA polymerases: The complete archaeal RNA polymerase structure. PLoS Biol. 7 (5), (2009).

