Перевод Рибосома сродство очищения (TRAP) для расследования Arabidopsis thaliana Корневая развития в масштабе типа клеток
Summary
Перевод рибосомной очистки сродства (TRAP) дает возможность вскрыть программы развития с минимальной обработкой органов и тканей. Протокол дает высококачественную РНК из клеток, ориентированных на зеленый флуоресцентный белок (GFP) помечены рибосомной субъединицы. Инструменты анализа вниз по течению, такие как qRT-PCR или RNA-seq, раскрывают профили экспрессии тканей и клеток.
Abstract
В этой статье мы даем практические инструкции для получения транслатомных данных от различных типов корневых клеток Arabidopsis thaliana с помощью метода очистки сродства рибосомы (TRAP) и последовательной оптимизированной подготовки библиотеки с низким уровнем ввода.
В качестве исходного материала, мы используем растительные линии, которые выражают GFP-тегами рибосомного белка RPL18 в клеточном типе-специфического образом с помощью адекватных промоутеров. Перед иммуноочищением и экстракцией РНК ткань замораживается, что сохраняет целостность тканей и одновременно позволяет проводить исследования временных рядов с высоким временным разрешением. Примечательно, что структуры клеточной стенки остаются нетронутыми, что является основным недостатком в альтернативных процедурах, таких как флуоресценция активированных клеток сортировки на основе подходов, которые полагаются на ткани протоптертодляя изолировать различные популяции клеток. Кроме того, не требуется фиксация тканей, как в лазерных захвата микродиссекции на основе методов, что позволяет высококачественной РНК быть получены.
Однако отбор проб из субпопуляций клеток и только изоляция полисососообразной РНК серьезно ограничивает урожайность РНК. Поэтому необходимо применять достаточно чувствительные методы подготовки библиотекдля для успешного получения данных РНК-сек.
TRAP предлагает идеальный инструмент для исследования растений, так как многие процессы развития включают клеточные стены, связанные с механическими и механическими сигнальными путями. Использование промоутеров для целевых конкретных популяций клеток преодолевает разрыв между органом и одноклеточным уровнем, которые, в свою очередь, страдают от небольшого разрешения или очень высоких затрат. Здесь мы применяем TRAP для изучения клеточной связи в боковом корневом образовании.
Introduction
Благодаря все более широкому применению методов секвенирования следующего поколения, пространственное разрешение в биологии развития может быть увеличено. Современные исследования направлены на вскрытие тканей вплоть до специализированных типов клеток, если не одноклеточного уровня1,,2,,3,,4. С этой целью за последние пятьдесят лет было разработано множество различных методов (см. рисунок 1A)5,6,6,7,,88,9,,10,,11,,12,,13,,14,,15.
Многие инструменты в науке о растениях были адаптацией методов, которые были впервые в исследованиях на животных. Это не относится к методу, который мы вводим в деталях здесь. В 2005 году, оснащенный сильным фоном в переводе белка, Лаборатория Бейли-Серрес приступила к разработке рибосомных белков для последующего очищения сродства16. Таким образом, они могли бы избежать трудоемких и трудоемких полисомного профилирования, которое основано на ультрацентрифугации с градиентом сахарозы и было использовано для оценки перевода рибосом с 1960-хгодов 17,18. Метод с тех пор был назван переводной рибосомы очистки сродства (TRAP)16. После успешных исследований транслатома в растениях, Heiman et al. адаптированы TRAP для животных19 и другие расширили свое применение дрожжей20, Drosophila21, Xenopus22 и зебрафиш23,24.
Хотя генетическая модификация модельной системы является необходимым условием для TRAP, которая ограничивает ее применение видов, поддавшихся генетической трансформации, можно одновременно использовать это возражение в целевых подмножествах клеток, которые представляют особый интерес и в противном случае чрезвычайно трудно изолировать от нетронутой ткани / органа25 (например, высоко разветвленные дендритные клетки в мозге мыши или грибковых hyphae инфицированных растительных тканей). В растениях все клетки удерживаются на месте через клеточные стенки, которые составляют основу гидростатического скелета26. Чтобы освободить клетку растений из этой матрицы, ученые либо физически вырезать клетку из окружающей ткани с помощью лазерного захвата микродиссекции (LCM)27 или выполняется ферментативное пищеварение клеточныхстенок 28. Среди последних клеток, так называемых протопластов, популяция, интересующая сяртово,констачированная и может быть отделена с помощью флуоресцентной сортировки клеток (FACS)7. LCM обычно требует, чтобы образец был исправлен и встроен в воск, что в конечном итоге ухудшает качество его РНК29. Методы на основе FACS дают высококачественную РНК, но сам процесс протоптертора вводит различия в экспрессии генов30, а ткани с модифицированными и толстыми вторичными клеточными стенками, как известно, трудно поддаются лечению. Кроме того, многие процессы развития в растениях, как предполагается, полагают, полагаются на механически передаваемые сигналы и, следовательно, целостность клеточной стенки имеет первостепенное значение31. Два метода, которые используют ярлык, чтобы обойти изоляцию клеток, работая на уровне ядер, являются флуоресценция активированной ядерной сортировки (FANS) и изоляции ядер помечены в конкретных типах клеток (INTACT). Как и в TRAP, они используют клеточные тип-специфические промоутеры для того чтобы маркировать ядра, которые затем получают обогащенными через сортировать или вытягивать вниз, соответственно8,,15. Основная проблема для всех этих подходов заключается в том, чтобы получить достаточное количество РНК-материала из подмножества клеток в ткани. Поскольку TRAP захватывает лишь часть клеточных РНК, сбор образцов является значительным узким местом. Поэтому для получения высококачественных данных из низких объемов ввода необходимы особенно чувствительные протоколы подготовки библиотек.
С момента своего создания, TRAP был либо использован в сочетании с ДНК microarrays или, как секвенирование расходы значительно снизились в последние годы, РНК-сек10,32,33. Множество вопросов исследования уже были выяснены, как рассмотрено в Sablok и др.34. Мы убеждены, что больше докладов будет следовать в ближайшие годы, как техника является очень универсальным при объединении различных промоутеров для целевых конкретных типов клеток. В конце концов, это будет сделано даже в индуцируемой образом, и может быть объединена с зондирования реакции растения на многие биотических и абиотических факторов стресса. Кроме того, там, где стабильные трансгенные линии отсутствуют, волосатые системы экспрессии корней также успешно используются для выполнения TRAP в томате и medicago35,36.
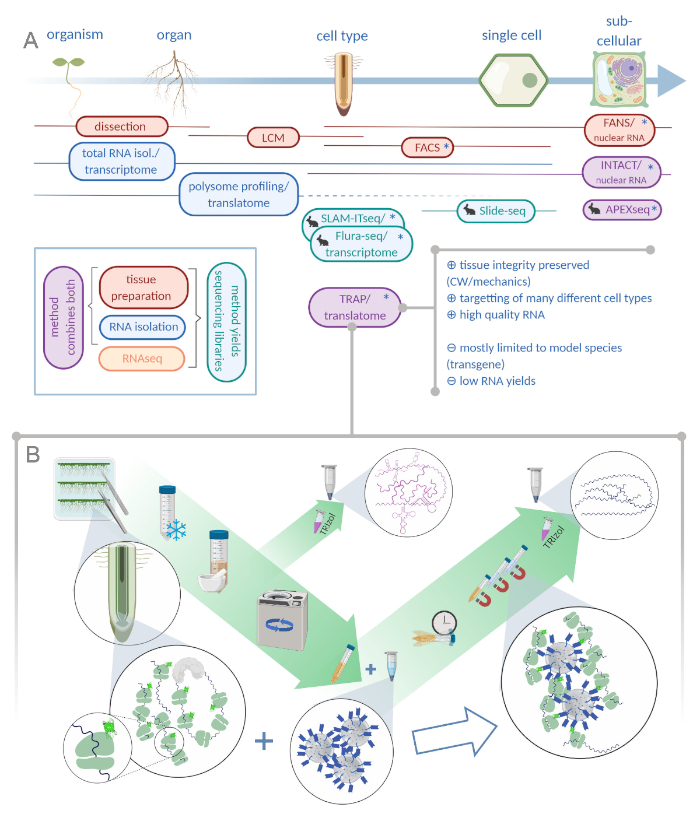
Рисунок 1: Перевод циризующей сродства рибосомы (TRAP) дополняет портфель анализа “омики”. О. Повышение уровня аналитической точности, вплоть до одноклеточного или даже субклеточного разрешения может быть достигнуто с помощью множества методов или их комбинаций. Схема дает обзор имеющихся в настоящее время инструментов в области растений и животных. Сбор тканей при клеточном разрешении может быть достигнут с помощью таких протоколов, как LCM или FACS, которые затем соединяются со стандартным транскриптомом или полисомным профилированием/анализом транслатома. TRAP и INTACT интегрируют захват тканей и изоляцию РНК, поскольку они основаны на эпитоп-пометке. Тем не менее, образцы INTACT только ядра клеток и представляет собой, следовательно, особый случай анализа транскриптома. Небольшой значок кролика отмечает недавно разработанные методы в области животных: В то время как SLAM-ITseq и Flura-seq полагаются на метаболическое тарзание зарождающихся РНК с модифицированными базами урацила в клетках, выражающих разрешительный фермент, Slide-seq использует стеклянный слайд с покрытием со штрих-кодами ДНК, которые предоставляют позиционную информацию в клеточном диапазоне. В APEX-seq используется подход к маркировке непосредственной маркировки для пробы РНК в конкретных субклеточных отсеках. Примечательно, что повышенное разрешение часто требует генерации трансгенного материала (звездочек), и эти методы, таким образом, в основном используются для типовых видов. TRAP особенно подходит для исследований растительной науки с участием клеточной стенки (CW) или механической сигнализации, а также клеточных видов, которые трудно выпустить из их матрицы CW. B. Детальные шаги влажной лаборатории процедуры TRAP: Рассада, выражающая GFP-тегами рибосомный белок в различных типах клеток (например, корневые эндодермисы), выращиваются на чашках Петри в течение семи дней, а корневой материал собирают путем замораживания оснастки. Общий образец контроля РНК собирается из гомогенизированного сырого экстракта, прежде чем гранулировать мусор с помощью центрифугации. Магнитные бусы анти-GFP добавляются в очищенный экстракт для выполнения иммунопротеберации. После инкубации и трех этапов стирки, полисомо-ассоциированной РНК (TRAP /полисома РНК) непосредственно получается через экстракцию фенола-хлороформа. LCM: лазерный захват микродиссекции, FACS/ FANS: флуоресценция активированной ячейки / ядерной сортировки, APEX-seq: метод, основанный на инженерии аскорбат перекистой, INTACT: изоляция ядер помечены в конкретных типах клеток, SLAM-ITseq: тиол (SH)-связанных алкилирования для метаболического секвенирования РНК в тканях, Flura-seq: фторурацил помечены РНК секвенирования (Создано с Biorender.com) Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
Целью данной статьи является предоставление подробного описания метода TRAP, выделение критических шагов и руководство по возможному методу подготовки библиотеки.
Общий эксперимент TRAP будет по существу состоять из следующих шагов (см. также Рисунок 1B):(1) Подготовка растительного материала, включая клонирование рибосом-теговая конструкция, трансгенное производство линии и отбор, выращивание и ссыпая семян, стерилизации и покрытия, а также стрессовое применение/обработка (необязательно) и сбор тканей; (2) иммуноочищение, включая гомогенизацию тканей и очистку сырого экстракта, мытье биса и иммуноочищение, и шаги мытья; (3) добыча РНК и оценка качества; и (4) подготовка библиотеки.
Корень Arabidopsis была модельсистемы для изучения развития растений с момента его введения в качестве модели завода37,38. Здесь применение TRAP демонстрируется в контексте развития бокового корня растений. В растениях, накопление всей корневой системы опирается на выполнение этой программы и поэтому очень важно для выживания организма39. В Arabidopsis, боковые корни происходят из ткани перицикл, который находится рядом с ксилем судов и, следовательно, называется ксилем полюс перицикл (XPP; см. Рисунок 2C)40. Некоторые клетки XPP, которые расположены глубоко внутри корня, приобретают идентичность ячейки основателя и, на местном гормональном триггере, начинают размножаться путем отеков и деления антиклинально41. Однако, из-за наличия жесткой матрицы стенки клетки, этот процесс оказывает механическое давление на окружающие ткани. В частности, поражена надлежащая эндодермис, так как она находится на пути боковой оси роста корня42,,43,,44. Действительно, вновь формирующихся primordium придется расти через надлежащую клетку эндодермис(Рисунок 2C2) в то время как коры и эпидермиз клетки просто отодвинуты в сторону для приморского,наконец,выйти45,46. Недавняя работа в нашей лаборатории показала, что эндодермис активно способствует распространению в перицикле. Целенаправленной блокировки эндодермальной гормональной сигнализации достаточно, чтобы ингибировать даже самое первое деление в клетках XPP47. Таким образом, перицикл эндодермис связи представляет собой очень ранний контрольно-пропускной пункт для бокового развития корней в Arabidopsis. Это, однако, не известно, как этот перекрестный разговор выполняется. Чтобы разгадать эту тайну, мы выбрали trap-seq подход к целевой XPP и эндодермальных клеток. Чтобы обогатить клетки в боковой корневой программе, мы имитировали гормональный триггер, экзогенно применяя аналог auxin (1-нафталенеацетической кислоты, NAA)48, который в то же время позволил временно решить начальную фазу бокового образования корня.
Protocol
Representative Results
Discussion
Проверка шаблона локализации RPL18
Решающее значение для того, чтобы избежать неправильного толкования данных из любого эксперимента TRAP, имеет правильную модель выражения помеченного рибосомального подразделения. Таким образом, включение GFP в качестве эпитопа тега RPL18 очень ?…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Мы хотели бы поблагодарить центр генетического разнообразия цюрихского центра генетического разнообразия за важные консультации экспертов на ранней стадии этого проекта. Работа в лаборатории Vermeer была поддержана грантом sNF Professorship (PP00P3_157524) и грантом на оборудование R’EQUIP (316030-164086) от Швейцарского национального научного фонда (SNSF), присужденного JEMV.
Materials
| Sterilization | |||
| bleach, 13% | Sigma | 71696 | |
| beaker | VWR | 214-1172/74/75 | |
| desiccator with porcelaine plate (DURAN) | Sigma/Merck | Z317454-1EA/Z317594-1EA | |
| EtOH, p.a. | Honeywell | 02860-1L | |
| HCl, 37% | Roth | 4625.1 | |
| Tween 20 | Sigma | P9416 | |
| Plate growth + harvesting | |||
| MS salts, basal salt mixture, incl. MES buffer | Duchefa | M0254 | |
| agar plant for cell culture | Applichem/Panreac | A2111.1000 | |
| DMSO | Sigma | D4540 | |
| forcepts | Rubis Switzerland | 5-SA model | |
| KOH | Fluka | 60370 | |
| micropore/surgical tape | 3M | 1530-0 | |
| NAA | Duchefa | N0903 | |
| petri dishes 120×120 mm | Greiner bio-one | 688102 | |
| scalpel | VWR/Swann-Morton | 233-5454 | |
| tissues, neutral, two-layered | any supplier of your choice | ||
| Immunoprecipitation | |||
| GFP-beads: gtma-100 GFP-Trap_MA | Chromotek | e.g. gtma-100 | |
| Brij-35 | Sigma | P1254-500G | |
| centrifuge tubes (in accordance with centrifuge) | Beckman Coulter | 357001 | |
| Chloramphenicol | Applichem | C0378-25G | |
| cotton gloves | VWR | 113-7355 | |
| Cycloheximide, HPLC grade | Sigma | 01810-1G | |
| DEPC | VWR | E174 | might have long delivery times |
| DTT | Fluka | 43815 | |
| EGTA | Sigma | 3054.3 | |
| homogenizers DUALL 23 | KONTES GLASS CO (via VWR) | SCERSP885450-0023 (set) | SCERSP885451-0023 pestle only – SCERSP885452-0023 cylinder only; long delivery times |
| Igepal CA-360 | Sigma | I3021-100ml | |
| KCl | Sigma | 60130 | |
| MgCl2 hexahydrat | Roth | 2189.2 | |
| mortar and pestle | VWR | 470148-960 & 470019-978 | |
| PMSF | Roche | 10 837 091 001 | |
| Polyoxyethylene-(10)-tridecylether/PTE | Sigma | P2393-500G | |
| RNase-free water | Roth | T143.3 | |
| RNAZap | Thermo Fisher | AM9780/AM9782 | for cleaning surfaces |
| Tris, >99.3% | Roth | AE15.3 | |
| Triton X-100 | Fluka | T8787-250ml | |
| Tween 20 | Sigma | P9416-100ml | |
| RNA extraction | |||
| 2-Propanol, p.a. | Sigma | 33539-1L-GL-R | |
| Chloroform, HPLC grade | Scharlau | CL02181000 | |
| EtOH, p.a. | Honeywell | 02860-1L | |
| low-retention microcentrifuge tubes, 1.5 ml | Eppendorf/Sigma | Z666548-250EA | LoBind |
| RNase-free DNase set | Qiagen | 79254 | |
| RNeasy MiniElute Cleanup Kit | Qiagen | 74204 | |
| TRIzol reagent | ThermoFisher/Ambion | 15596018 | |
| Library preparation | |||
| 15/50 mL Tube Magnetic Separator | Abraxis | PN 472250 | |
| AMPure beads | Beckman Coulter | A63881 | |
| Index Kit A | Illumina | FC-131-2001 | |
| Index Kit D | Illumina | FC-131-2004 | |
| neodymium magnets | Amazon/other | 6 x 1.5 mm range: N42 (NdFeB) | |
| Nextera XT kit | Illumina | FC-131-1024/1096 | https://emea.support.illumina.com/ |
| PCR strips | ThermoScientific | AB-0266 | |
| SMARTer v4 kit | Takara Bioscience | 634892 | https://www.takarabio.com/ |
| Bioanalyzer | Agilent | 2100 Bioanalyzer Instrument | specialized equipment for RNA/DNA quality control |
| Tapestation | Agilent | 4200 Tapestation Instrument | specialized equipment for RNA/DNA quality control |
| Fragment Analyzer | Agilent | 5400 Fragment Analyzer System | specialized equipment for RNA/DNA quality control (high throughput) |
| LabChip | PerkinElmer | LabChip GX Touch Nucleic Acid Analyzer | specialized equipment for RNA/DNA quality control (high throughput) |
| Qubit 4 Fluorometer | ThermoFisher | Q33239 | specialized equipment for RNA/DNA concentration determination |
| qRT-PCR | |||
| GATA23 | Microsynth | fwd: AGTGAGAATGAA AGAAGAGAAGGG; rev: GTGGCTGCGAAT AATATGAATACC |
|
| GH3.3 | Microsynth | fwd: CAAACCAATCCT CCAAATGAC; rev: ACTTATCCGCAA CCCGACT |
|
| LBD29 | Microsynth | fwd: TCTCCAACAACA GGTTGTGAAT; rev: AAGGAGCCTTAG TAGTGTCTCCA |
|
| UBC21 | Microsynth | fwd: TGCGACTCAGGG AATCTTCT; rev: TCATCCTTTCTT AGGCATAGCG |
|
| SsoAdvanced Universal SYBR Green | Bio-Rad | #172-5270 | |
| iScript Adv cDNA Kit | Bio-Rad | #172-5038 | |
| miscellaneous | |||
| Falcon tubes 15 ml, Cellstar | Greiner bio-one | 188261 | |
| Falcon tubes 50 ml, Cellstar | Greiner bio-one | 210261 | |
| filter tips 1 ml | Axygen | TF-1000-R-S | |
| filter tips 10 µl | Axygen | TF-10-R-S | |
| filter tips 100 µl | Axygen | TF-100-R-S | |
| filter tips 20 µl | Axygen | TF-20-R-S | |
| filter tips 200 µl | Axygen | TF-200-R-S | |
| microcentrifuge tubes 1.5 ml | SARSTEDT | 72.690.001 | |
| Propidium iodide | Sigma | P4170-100MG | |
| sequencing company | Novogene | en.novogene.com |
References
- Van Verk, M. C., Hickman, R., Corné, M. J., Pieterse, M., Van Wees, S. C. RNA-Seq: Revelation Of The Messengers. Trends In Plant Science. 18 (4), 175-179 (2013).
- Libault, M., Pingault, L., Zogli, P., Schiefelbein, J. Plant Systems Biology At The Single-Cell Level. Trends In Plant Science. 22 (11), 949-960 (2017).
- Mustroph, A., et al. Profiling Translatomes Of Discrete Cell Populations Resolves Altered Cellular Priorities During Hypoxia In Arabidopsis. Proceedings Of The National Academy Of Sciences Of The United States Of America. 106 (44), 18843-18848 (2009).
- Karve, R., Iyer-Pascuzzi, A. S. Digging Deeper: High-Resolution Genome-Scale Data Yields New Insights Into Root Biology. Current Opinion In Plant Biology. 24, 24-30 (2015).
- Warner, J. R., Knopf, P. M., Rich, A. A Multiple Ribosomal Structure In Protein Synthesis. Proceedings of The National Academy of Sciences of The United States of America. 49 (1), 122-129 (1963).
- Gautam, V., Sarkar, A. K. Laser Assisted Microdissection, An Efficient Technique To Understand Tissue Specific Gene Expression Patterns And Functional Genomics In Plants. Molecular Biotechnology. 57 (4), 299-308 (2015).
- Bargmann, B. O. R., Birnbaum, K. D. Fluorescence Activated Cell Sorting Of Plant Protoplasts. Journal of Visualized Experiments. (36), e1673 (2010).
- Deal, R. B., Henikoff, S. The Intact Method For Cell Type-Specific Gene Expression And Chromatin Profiling In Arabidopsis Thaliana. Nature Protocols. 6 (1), 56-68 (2011).
- Dougherty, J. D. The Expanding Toolkit Of Translating Ribosome Affinity Purification. The Journal of Neuroscience: The Official Journal Of The Society For Neuroscience. 37 (50), 12079-12087 (2017).
- Mustroph, A., Juntawong, P., Bailey-Serres, J. Isolation Of Plant Polysomal mRNA By Differential Centrifugation And Ribosome Immunopurification Methods. Methods in Molecular Biology. 553, 109-126 (2009).
- Matsushima, W., et al. SLAM-ITseq: Sequencing Cell Type-Specific Transcriptomes Without Cell Sorting. Development. 145 (13), (2018).
- Basnet, H., et al. Flura-Seq Identifies Organ-Specific Metabolic Adaptations During Early Metastatic Colonization. Elife. 8, (2019).
- Rodriques, S. G., et al. Slide-Seq: A Scalable Technology For Measuring Genome-Wide Expression At High Spatial Resolution. Science. 363 (6434), 1463-1467 (2019).
- Fazal, F. M., et al. Atlas Of Subcellular RNA Localization Revealed By Apex-Seq. Cell. 178 (2), 473-490 (2019).
- Slane, D., Bayer, M., Kaufmann, K., Mueller-Roeber, B. Cell Type-Specific Gene Expression Profiling Using Fluorescence-Activated Nuclear Sorting. Plant Gene Regulatory Networks: Methods And Protocols. , 27-35 (2017).
- Zanetti, M. E., Chang, I. F., Gong, F., Galbraith, D. W., Bailey-Serres, J. Immunopurification Of Polyribosomal Complexes Of Arabidopsis For Global Analysis Of Gene Expression. Plant Physiology. 138 (2), 624-635 (2005).
- King, H. A., Gerber, A. P. Translatome Profiling: Methods For Genome-Scale Analysis Of mRNA Translation. Briefings In Functional Genomics. 15 (1), 22-31 (2016).
- Mašek, T., Valášek, L., Pospíšek, M., Nielsen, H. Polysome Analysis And RNA Purification From Sucrose Gradients. RNA: Methods And Protocols. , 293-309 (2011).
- Heiman, M., et al. A Translational Profiling Approach For The Molecular Characterization Of Cns Cell Types. Cell. 135 (4), 738-748 (2008).
- Halbeisen, R. E., Scherrer, T., Gerber, A. P. Affinity Purification Of Ribosomes To Access The Translatome. Methods. 48 (3), 306-310 (2009).
- Thomas, A., et al. A Versatile Method For Cell-Specific Profiling Of Translated mRNAs In Drosophila. Plos One. 7 (7), e40276 (2012).
- Watson, F. L., et al. Cell Type-Specific Translational Profiling In The Xenopus Laevis Retina. Developmental Dynamics. 241 (12), 1960-1972 (2012).
- Lam, P. Y., Harvie, E. A., Huttenlocher, A. Heat Shock Modulates Neutrophil Motility In Zebrafish. Plos One. 8 (12), e84436 (2013).
- Fang, Y., et al. Translational Profiling Of Cardiomyocytes Identifies An Early Jak1/Stat3 Injury Response Required For Zebrafish Heart Regeneration. Proceedings Of The National Academy Of Sciences Of The United States Of America. 110 (33), 13416-13421 (2013).
- Mustroph, A., Zanetti, M. E., Girke, T., Bailey-Serres, J. Isolation And Analysis Of mRNAs From Specific Cell Types Of Plants By Ribosome Immunopurification. Methods In Molecular Biology. 959, 277-302 (2013).
- Monshausen, G. B., Gilroy, S. Feeling Green: Mechanosensing In Plants. Trends In Cell Biology. 19 (5), 228-235 (2009).
- Day, R. C., Grossniklaus, U., Macknight, R. C. Be More Specific! Laser-Assisted Microdissection Of Plant Cells. Trends In Plant Science. 10 (8), 397-406 (2005).
- Sheen, J. Signal Transduction In Maize And Arabidopsis Mesophyll Protoplasts. Plant Physiology. 127 (4), 1466-1475 (2001).
- Datta, S., et al. Laser Capture Microdissection: Big Data From Small Samples. Histology And Histopathology. 30 (11), 1255-1269 (2015).
- Birnbaum, K., et al. A Gene Expression Map Of The Arabidopsis Root. Science. 302 (5652), 1956 (2003).
- Hamant, O., Haswell, E. S. Life Behind The Wall: Sensing Mechanical Cues In Plants. BMC Biology. 15 (1), 1354 (2017).
- Vragović, K., et al. Translatome Analyses Capture Of Opposing Tissue-Specific Brassinosteroid Signals Orchestrating Root Meristem Differentiation. Proceedings of The National Academy of Sciences of the United States of America. 112 (3), 923-928 (2015).
- Wang, Y., Jiao, Y. Translating Ribosome Affinity Purification (Trap) For Cell-Specific Translation Profiling In Developing Flowers. Methods In Molecular Biology. 1110, 323-328 (2014).
- Sablok, G., Powell, J. J., Kazan, K. Emerging Roles And Landscape Of Translating mRNAs In Plants. Frontiers in Plant Science. 8, 1443 (2017).
- Ron, M., et al. Hairy Root Transformation Using Agrobacterium Rhizogenes As A Tool For Exploring Cell Type-Specific Gene Expression And Function Using Tomato As A Model. Plant Physiology. 166 (2), 455-469 (2014).
- Reynoso, M. A., et al. Evolutionary Flexibility In Flooding Response Circuitry In Angiosperms. Science. 365 (6459), 1291-1295 (2019).
- Dolan, L., et al. Cellular Organisation Of The Arabidopsis Thaliana Root. Development. 119 (1), 71 (1993).
- Ristova, D., Barbez, E. . Root Development. , (2018).
- Shekhar, V., Stӧckle, D., Thellmann, M., Vermeer, J. E. M. The Role Of Plant Root Systems In Evolutionary Adaptation. Current Topics in Developmental Biology. 131, 55-80 (2019).
- Malamy, J. E., Benfey, P. N. Down And Out In Arabidopsis: The Formation Of Lateral Roots. Trends in Plant Science. 2 (10), 390-396 (1997).
- de Smet, I., et al. Bimodular Auxin Response Controls Organogenesis In Arabidopsis. Proceedings of the National Academy of Sciences of The United States of America. 107 (6), 2705-2710 (2010).
- Péret, B., et al. Arabidopsis Lateral Root Development: An Emerging Story. Trends In Plant Science. 14 (7), 399-408 (2009).
- Vilches-Barro, A., Maizel, A. Talking Through Walls: Mechanisms Of Lateral Root Emergence In Arabidopsis Thaliana. Current Opinion in Plant Biology. 23, 31-38 (2015).
- Porco, S., et al. Lateral Root Emergence In Arabidopsis Is Dependent On Transcription Factor Lbd29 Regulation Of Auxin Influx Carrier Lax3. Development. 143 (18), 3340-3349 (2016).
- Stoeckle, D., Thellmann, M., Vermeer, J. E. Breakout-Lateral Root Emergence In Arabidopsis Thaliana. Current Opinion in Plant Biology. 41, 67-72 (2018).
- Banda, J., et al. Lateral Root Formation In Arabidopsis: A Well-Ordered Lrexit. Trends in Plant Science. 24 (9), 826-839 (2019).
- Vermeer, J. E. M., et al. A Spatial Accommodation By Neighboring Cells Is Required For Organ Initiation In Arabidopsis. Science. 343 (6167), 178-183 (2014).
- Vanneste, S., et al. Cell Cycle Progression In The Pericycle Is Not Sufficient For Solitary Root/Iaa14-Mediated Lateral Root Initiation In Arabidopsis Thaliana. The Plant Cell. 17 (11), 3035-3050 (2005).
- Marques-Bueno, M. M., et al. A Versatile Multisite Gateway-Compatible Promoter And Transgenic Line Collection For Cell Type-Specific Functional Genomics In Arabidopsis. The Plant Journal : For Cell and Molecular Biology. 85 (2), 320-333 (2016).
- Shimada, T. L., Shimada, T., Hara-Nishimura, I. A Rapid And Non-Destructive Screenable Marker, Fast, For Identifying Transformed Seeds Of Arabidopsis Thaliana. The Plant Journal : For Cell and Molecular Biology. 61 (3), 519-528 (2010).
- Clough, S. J., Bent, A. F. Floral Dip: A Simplified Method For Agrobacterium Mediated Transformation Of Arabidopsis Thaliana. The Plant Journal. 16 (6), 735-743 (1998).
- Lindsey, B. E., Rivero, L., Calhoun, C. S., Grotewold, E., Brkljacic, J. Standardized Method For High-Throughput Sterilization Of Arabidopsis Seeds. Journal Of Visualized Experiments. (128), e56587 (2017).
- Andersen, T. G., et al. Diffusible Repression Of Cytokinin Signalling Produces Endodermal Symmetry And Passage Cells. Nature. 555, 529-533 (2018).
- Schroeder, A., et al. The Rin: An Rna Integrity Number For Assigning Integrity Values To Rna Measurements. BMC Molecular Biology. 7, 3 (2006).
- Vragović, K., Bartom, E., Savaldi-Goldstein, S. Quantitation Of Cell Type-Specific Responses To Brassinosteroid By Deep Sequencing Of Polysome-Associated Polyadenylated RNA. Methods in Molecular Biology. 1564, 81-102 (2017).
- Bertin, B., Renaud, Y., Aradhya, R., Jagla, K., Junion, G. Trap-Rc, Translating Ribosome Affinity Purification From Rare Cell Populations Of Drosophila Embryos. Journal Of Visualized Experiments. (103), e52985 (2015).
- Livak, K. J., Schmittgen, T. D. Analysis Of Relative Gene Expression Data Using Real-Time Quantitative PCR And The 2(-Delta Delta C(T)) Method. Methods. 25 (4), 402-408 (2001).
- Jiao, Y., Meyerowitz, E. M. Cell-Type Specific Analysis Of Translating Rnas In Developing Flowers Reveals New Levels Of Control. Molecular Systems Biology. 6, 419 (2010).
- Tian, C., et al. A Gene Expression Map Of Shoot Domains Reveals Regulatory Mechanisms. Nature Communications. 10 (1), 141 (2019).
- Townsley, B. T., Covington, M. F., Ichihashi, Y., Zumstein, K., Sinha, N. R. Brad-Seq: Breath Adapter Directional Sequencing: A Streamlined, Ultra-Simple And Fast Library Preparation Protocol For Strand Specific mRNA Library Construction. Frontiers in Plant Science. 6, 366 (2015).
- Song, Y., et al. A Comparative Analysis Of Library Prep Approaches For Sequencing Low Input Translatome Samples. BMC Genomics. 19 (1), 696 (2018).
- Basu, D., Haswell, E. S. Plant Mechanosensitive Ion Channels: An Ocean Of Possibilities. Current Opinion in Plant Biology. 40, 43-48 (2017).
- Brady, S. M., et al. A High-Resolution Root Spatiotemporal Map Reveals Dominant Expression Patterns. Science. 318 (5851), 801 (2007).

