Traduzindo a Purificação da Afinidade ribossófica (TRAP) para investigar o desenvolvimento raiz arabidopsis thaliana em uma escala específica de tipo de célula
Summary
Traduzir purificação de afinidade costela (TRAP) oferece a possibilidade de dissecar programas de desenvolvimento com processamento mínimo de órgãos e tecidos. O protocolo produz RNA de alta qualidade a partir de células direcionadas com uma subunidade ribossômica rotulada como proteína fluorescente verde (GFP). Ferramentas de análise a jusante, como qRT-PCR ou RNA-seq, revelam perfis de expressão específicos de tecido e célula.
Abstract
Neste artigo, damos instruções práticos para obter dados translatome de diferentes tipos de células radiculares Arabidopsis thaliana através do método de purificação de afinidade costelado (TRAP) traduzindo e preparação consecutiva de biblioteca otimizada otimizada.
Como material inicial, empregamos linhas de plantas que expressam a proteína ribossômica RPL18 com gfp de forma específica do tipo celular, utilizando promotores adequados. Antes da imunopurificação e extração de RNA, o tecido é congelado, o que preserva a integridade do tecido e permite simultaneamente a execução de estudos de séries temporais com alta resolução temporal. Notavelmente, as estruturas de parede celular permanecem intactas, o que é uma grande desvantagem em procedimentos alternativos, como abordagens baseadas em triagem celular ativadas por fluorescência que dependem de protoplasting tecidual para isolar populações celulares distintas. Além disso, nenhuma fixação tecidual é necessária como em técnicas baseadas em microdissecção de captura a laser, o que permite obter RNA de alta qualidade.
No entanto, a amostragem de subpopulações de células e apenas o RNA associado ao polisome severamente limita severamente os rendimentos de RNA. É, portanto, necessário aplicar métodos suficientemente sensíveis de preparação de bibliotecas para a aquisição bem-sucedida de dados pelo RNA-seq.
O TRAP oferece uma ferramenta ideal para a pesquisa de plantas, pois muitos processos de desenvolvimento envolvem vias de sinalização mecânica e relacionadas à parede celular. O uso de promotores para atingir populações celulares específicas está fazendo a ponte entre o nível de órgãos e células únicas que, por sua vez, sofrem de pouca resolução ou custos muito altos. Aqui, aplicamos TRAP para estudar a comunicação celular-célula na formação de raízes laterais.
Introduction
Impulsionada pela crescente aplicação de técnicas de sequenciamento de última geração, a resolução espacial na biologia do desenvolvimento poderia ser aumentada. Estudos contemporâneos visam dissecar tecidos para tipos de células especializadas, se não o nível de célulaúnica11,2,,3,4. Para isso, uma infinidade de métodos diferentes foi concebida ao longo dos últimos cinquenta anos (ver Figura 1A)5,6,,7,,688,99,10,,11,,12,,13,,14,15.
Muitas ferramentas em ciência vegetal têm sido adaptações de técnicas que foram pioneiras na pesquisa animal. Este não é o caso para o método que estamos introduzindo em detalhes aqui. Em 2005, equipado com uma forte formação em tradução de proteínas, o Laboratório Bailey-Serres começou a projetar proteínas ribossômicas para posterior purificação de afinidade16. Assim, poderiam evitar o demorado e intensivo em perfil de polisério, que se baseia na ultracentrifugação com um gradiente de sacarose e foi utilizado para avaliar ribossomos traduzidos desde a década de 196017,18. Desde então, o método foi referido como purificação de afinidade de ribossomos traláricos (TRAP)16. Após estudos bem-sucedidos de translatome em plantas, Heiman et al. adaptaram TRAP para animais19 e outros estenderam sua aplicação para leveduras20, Drosophila21, Xenopus22 e zebrafish23,24.
Embora a modificação genética do sistema modelo seja um pré-requisito para trap, que limita sua aplicação a espécies passíveis de transformação genética, pode-se simultaneamente aproveitar essa objeção para atingir subconjuntos de células que são de interesse especial e de outra forma extremamente difíceis de isolar do tecido/órgão intacto25 (por exemplo, células dendríticas altamente ramificadas em um cérebro de camundongos ou hifas fúngicas em tecido vegetal infectado). Nas plantas, todas as células são mantidas no lugar através de paredes celulares que formam a base do esqueleto hidrostático26. Para libertar uma célula vegetal dessa matriz, os cientistas cortaram fisicamente a célula de seu tecido circundante através da microdissecção de captura a laser (LCM)27 ou realizaram digestão enzimática das paredes celulares28. Entre as últimas células, os chamados protoplastos, a população de interesse é fluorescentemente rotulada e pode ser separada por meio da triagem celular ativada por fluorescência (FACS)7. O LCM geralmente requer uma amostra para ser fixada e embutida em cera, o que acaba por deteriorar a qualidade do seu RNA29. Os métodos baseados em FACS produzem RNA de alta qualidade, mas o processo de protoplasting em si introduz diferenças na expressão genética30 e tecidos com paredes de células secundárias modificadas e grossas são notoriamente difíceis de tratar. Além disso, muitos processos de desenvolvimento nas plantas são assumidos para contar com sinais mecanicamente transmitidos e, portanto, a integridade da parede celular é de suma importância31. Dois métodos, que utilizam um atalho para contornar o isolamento celular operando no nível de núcleos, são a triagem nuclear ativada por fluorescência (FANS) e o isolamento de núcleos marcados em tipos de células específicas (INTACT). Como no TRAP, eles usam promotores específicos do tipo celular para marcar núcleos, que posteriormente são enriquecidos através da classificação ou puxar para baixo, respectivamente8,15. Um grande desafio para todas essas abordagens é obter material de RNA suficiente de subconjuntos de células em um tecido. Como o TRAP captura apenas uma fração dos RNAs celulares, a coleta de amostras é um gargalo considerável. Portanto, protocolos especialmente sensíveis de preparação de bibliotecas são necessários para produzir dados de alta qualidade a partir de baixas quantidades de entrada.
Desde a sua criação, o TRAP tem sido usado em combinação com micromatrizes de DNA ou, como os custos de sequenciamento caíram significativamente nos últimos anos, RNA-seq10,32,33. Uma infinidade de questões de pesquisa já foi elucidada como revisada em Sablok et al.34. Estamos convencidos de que mais relatórios serão seguidos nos próximos anos, pois a técnica é muito versátil ao combinar diferentes promotores para atingir tipos específicos de células. Eventualmente, isso será feito mesmo de forma indutível, e pode ser combinado com a sondagem da reação da planta a muitos fatores de estresse bióticos e abióticos. Além disso, quando linhas transgênicas estáveis não estão disponíveis, sistemas de expressão raiz peluda também têm sido usados com sucesso para realizar TRAP em tomate e medicago35,36.
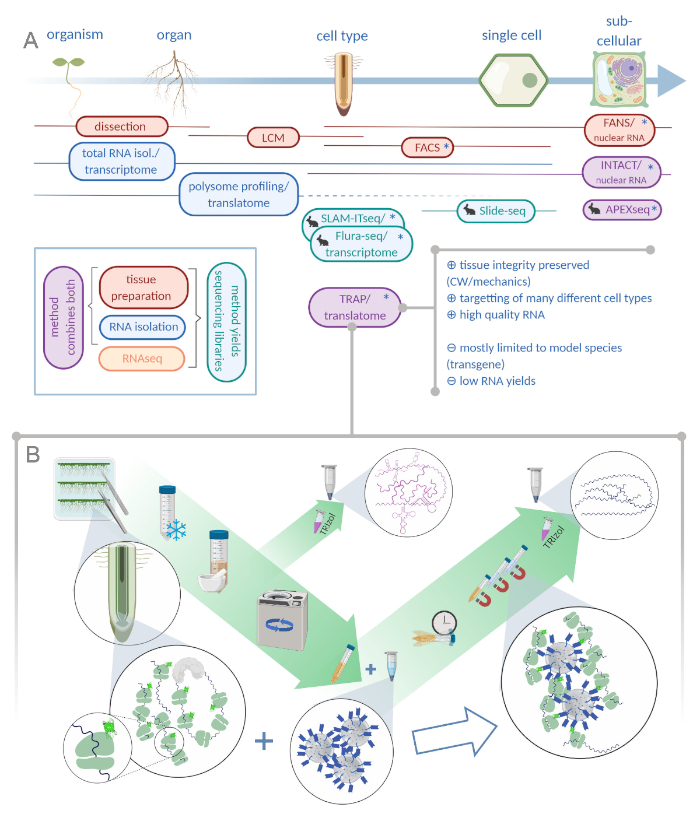
Figura 1: Traduzir purificação de afinidade de ribossomos (TRAP) complementa o portfólio de análise “ômica”. A. O aumento dos níveis de precisão analítica, até a resolução unicelular ou mesmo subcelular, pode ser alcançado por uma infinidade de métodos ou combinações. O esquema dá uma visão geral das ferramentas disponíveis atualmente no campo vegetal e animal. A coleta de tecidos na resolução celular pode ser alcançada por protocolos como LCM ou FACS, que são então acoplados à transcrição padrão ou análise de perfil/tradução polisome. TRAP e INTACT integram tanto a captura de tecido quanto o isolamento do RNA, pois são baseados na marcação de epítopos. No entanto, intacto amostra apenas núcleos celulares e constitui, portanto, um caso especial de análise de transcriptome. Um pequeno ícone de coelho marca métodos recém-desenvolvidos no campo animal: Enquanto SLAM-ITseq e Flura-seq dependem da segmentação metabólica de RNAs nascentes com bases uracil modificadas em células que expressam a enzima permissiva, Slide-seq faz uso de um slide de vidro revestido com códigos de barras de DNA que fornecem informações posicionais na faixa celular. Uma abordagem de rotulagem de proximidade é seguida em APEX-seq para amostrar RNAs em compartimentos subcelulares específicos. Notavelmente, o aumento da resolução muitas vezes requer a geração de material transgênico (asteriscos) e esses métodos são, portanto, predominantemente utilizados para espécies modelo. Trap é especialmente adequado para estudos de ciência vegetal envolvendo parede celular (CW) ou sinalização mecânica, bem como espécies celulares que são difíceis de liberar de sua matriz CW. B. Etapas detalhadas do laboratório molhado do procedimento TRAP: As mudas que expressam proteína ribossômica marcada por GFP em tipos de células distintas (por exemplo, endoderme raiz) são cultivadas em placas de Petri por sete dias e o material raiz colhido pelo congelamento instantâneo. Uma amostra total de controle de RNA é coletada do extrato bruto homogeneizado antes de pelleting os detritos via centrifugação. Contas anti-GFP magnéticas são adicionadas ao extrato limpo para realizar a imunoprecipitação. Após a incubação e três etapas de lavagem, o RNA associado ao pórnio (Trap/Polysome RNA) é obtido diretamente através da extração de fenol-clorofórmio. LCM: microdissecção de captura a laser, FACS/FANS: célula ativada por fluorescência/classificação nuclear, APEX-seq: método baseado na engenharia de ascorbate peroxidase, INTACT: isolamento de núcleos marcados em tipos de células específicas, SLAM-ITseq: alquilação vinculada ao thiol(SH) para o sequenciamento metabólico de RNA no tecido, Flura-seq: sequenciamento de RNA com rótulo fluorouracil (Criado com Biorender.com) Clique aqui para ver uma versão maior desta figura.
O objetivo deste artigo é fornecer uma descrição detalhada do método TRAP, destacar passos críticos e fornecer orientação para um possível método de preparação da biblioteca.
Um experimento trap genérico consistirá essencialmente nas seguintes etapas (ver também Figura 1B): (1) Preparação de material vegetal incluindo clonagem de construção de marcação de ribossomos, produção e seleção de linhas transgênicas, crescimento e acúmulo de sementes, esterilização e revestimento, e aplicação/tratamento de estresse (opcional) e colheita de tecidos; (2) imunopurificação, incluindo homogeneização tecidual e limpeza do extrato bruto, lavagem de abelhas e imunopurificação, e etapas de lavagem; (3) Extração de RNA e avaliação da qualidade; e (4) preparação da biblioteca.
A raiz arabidopsis tem sido um sistema modelo para estudar o desenvolvimento de plantas desde sua introdução como uma planta modelo37,38. Aqui, a aplicação do TRAP é apresentada no contexto do desenvolvimento da raiz lateral vegetal. Nas plantas, o acúmulo de todo o sistema radicular depende da execução deste programa e, portanto, é muito importante para a sobrevivência do organismo39. Em Arabidopsis,as raízes laterais originam-se do tecido periciclo que reside ao lado dos vasos xilem e, portanto, é denominado periciclo do pólo xilem (XPP; ver Figura 2C)40. Algumas células XPP, que estão localizadas no interior da raiz, adquirem uma identidade celular fundadora e, sobre um gatilho hormonal local, começam a se proliferar por inchaço e divisão anticlinalmente41. No entanto, devido à presença de uma matriz de parede celular rígida, este processo exerce estresse mecânico nos tecidos circundantes. Em particular, a endoderme sobreposta é afetada, pois está no caminho do eixo de crescimento da raiz lateral42,43,44. De fato, o primordium recém-formado terá que crescer através da célula endoderme sobreposta (Figura 2C2), enquanto as células córtex e epiderme são apenas deixadas de lado para que o primordium finalmente emerja45,46. Trabalhos recentes em nosso laboratório mostraram que a endoderme está contribuindo ativamente para acomodar a proliferação no periciclo. O bloqueio direcionado da sinalização hormonal endodérmica é suficiente para inibir até mesmo a primeira divisão nas células XPP47. Assim, a comunicação periciclo-endoderme constitui um ponto de verificação muito cedo para o desenvolvimento da raiz lateral na Arabidopsis. No entanto, não se sabe como esse crosstalk é realizado. Para desvendar esse mistério, escolhemos a abordagem TRAP-seq para atingir células XPP e endodérmicas. Para enriquecer para as células no programa raiz lateral, imitamos o gatilho hormonal aplicando exogenously um análogo auxina (1-ácido naftalinacático, NAA)48, que ao mesmo tempo permitiu resolver temporalmente a fase inicial da formação da raiz lateral.
Protocol
Representative Results
Discussion
Verificação do padrão de localização RPL18
Crucial para evitar má interpretação dos dados de qualquer experimento TRAP é o padrão de expressão adequado da subunidade ribossômica marcada. Portanto, a incorporação do GFP como uma tag epítopoa ao RPL18 de forma muito elegante permite a verificação do padrão de expressão desejado e, consecutivamente, a retração da fração polisome do mesmo tecido. Abordagens mais invasivas para assegurar padrões de promotores adequados são seguidas…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Gostaríamos de agradecer a Jean-Claude Walser, do Centro de Diversidade Genética de Zurique, por conselhos cruciais de especialistas na fase inicial deste projeto. O trabalho no laboratório Vermeer foi apoiado por uma bolsa de professor do SNF (PP00P3_157524) e uma bolsa de equipamentos R’EQUIP (316030_164086) da Fundação Nacional de Ciência suíça (SNSF) concedida ao JEMV.
Materials
| Sterilization | |||
| bleach, 13% | Sigma | 71696 | |
| beaker | VWR | 214-1172/74/75 | |
| desiccator with porcelaine plate (DURAN) | Sigma/Merck | Z317454-1EA/Z317594-1EA | |
| EtOH, p.a. | Honeywell | 02860-1L | |
| HCl, 37% | Roth | 4625.1 | |
| Tween 20 | Sigma | P9416 | |
| Plate growth + harvesting | |||
| MS salts, basal salt mixture, incl. MES buffer | Duchefa | M0254 | |
| agar plant for cell culture | Applichem/Panreac | A2111.1000 | |
| DMSO | Sigma | D4540 | |
| forcepts | Rubis Switzerland | 5-SA model | |
| KOH | Fluka | 60370 | |
| micropore/surgical tape | 3M | 1530-0 | |
| NAA | Duchefa | N0903 | |
| petri dishes 120×120 mm | Greiner bio-one | 688102 | |
| scalpel | VWR/Swann-Morton | 233-5454 | |
| tissues, neutral, two-layered | any supplier of your choice | ||
| Immunoprecipitation | |||
| GFP-beads: gtma-100 GFP-Trap_MA | Chromotek | e.g. gtma-100 | |
| Brij-35 | Sigma | P1254-500G | |
| centrifuge tubes (in accordance with centrifuge) | Beckman Coulter | 357001 | |
| Chloramphenicol | Applichem | C0378-25G | |
| cotton gloves | VWR | 113-7355 | |
| Cycloheximide, HPLC grade | Sigma | 01810-1G | |
| DEPC | VWR | E174 | might have long delivery times |
| DTT | Fluka | 43815 | |
| EGTA | Sigma | 3054.3 | |
| homogenizers DUALL 23 | KONTES GLASS CO (via VWR) | SCERSP885450-0023 (set) | SCERSP885451-0023 pestle only – SCERSP885452-0023 cylinder only; long delivery times |
| Igepal CA-360 | Sigma | I3021-100ml | |
| KCl | Sigma | 60130 | |
| MgCl2 hexahydrat | Roth | 2189.2 | |
| mortar and pestle | VWR | 470148-960 & 470019-978 | |
| PMSF | Roche | 10 837 091 001 | |
| Polyoxyethylene-(10)-tridecylether/PTE | Sigma | P2393-500G | |
| RNase-free water | Roth | T143.3 | |
| RNAZap | Thermo Fisher | AM9780/AM9782 | for cleaning surfaces |
| Tris, >99.3% | Roth | AE15.3 | |
| Triton X-100 | Fluka | T8787-250ml | |
| Tween 20 | Sigma | P9416-100ml | |
| RNA extraction | |||
| 2-Propanol, p.a. | Sigma | 33539-1L-GL-R | |
| Chloroform, HPLC grade | Scharlau | CL02181000 | |
| EtOH, p.a. | Honeywell | 02860-1L | |
| low-retention microcentrifuge tubes, 1.5 ml | Eppendorf/Sigma | Z666548-250EA | LoBind |
| RNase-free DNase set | Qiagen | 79254 | |
| RNeasy MiniElute Cleanup Kit | Qiagen | 74204 | |
| TRIzol reagent | ThermoFisher/Ambion | 15596018 | |
| Library preparation | |||
| 15/50 mL Tube Magnetic Separator | Abraxis | PN 472250 | |
| AMPure beads | Beckman Coulter | A63881 | |
| Index Kit A | Illumina | FC-131-2001 | |
| Index Kit D | Illumina | FC-131-2004 | |
| neodymium magnets | Amazon/other | 6 x 1.5 mm range: N42 (NdFeB) | |
| Nextera XT kit | Illumina | FC-131-1024/1096 | https://emea.support.illumina.com/ |
| PCR strips | ThermoScientific | AB-0266 | |
| SMARTer v4 kit | Takara Bioscience | 634892 | https://www.takarabio.com/ |
| Bioanalyzer | Agilent | 2100 Bioanalyzer Instrument | specialized equipment for RNA/DNA quality control |
| Tapestation | Agilent | 4200 Tapestation Instrument | specialized equipment for RNA/DNA quality control |
| Fragment Analyzer | Agilent | 5400 Fragment Analyzer System | specialized equipment for RNA/DNA quality control (high throughput) |
| LabChip | PerkinElmer | LabChip GX Touch Nucleic Acid Analyzer | specialized equipment for RNA/DNA quality control (high throughput) |
| Qubit 4 Fluorometer | ThermoFisher | Q33239 | specialized equipment for RNA/DNA concentration determination |
| qRT-PCR | |||
| GATA23 | Microsynth | fwd: AGTGAGAATGAA AGAAGAGAAGGG; rev: GTGGCTGCGAAT AATATGAATACC |
|
| GH3.3 | Microsynth | fwd: CAAACCAATCCT CCAAATGAC; rev: ACTTATCCGCAA CCCGACT |
|
| LBD29 | Microsynth | fwd: TCTCCAACAACA GGTTGTGAAT; rev: AAGGAGCCTTAG TAGTGTCTCCA |
|
| UBC21 | Microsynth | fwd: TGCGACTCAGGG AATCTTCT; rev: TCATCCTTTCTT AGGCATAGCG |
|
| SsoAdvanced Universal SYBR Green | Bio-Rad | #172-5270 | |
| iScript Adv cDNA Kit | Bio-Rad | #172-5038 | |
| miscellaneous | |||
| Falcon tubes 15 ml, Cellstar | Greiner bio-one | 188261 | |
| Falcon tubes 50 ml, Cellstar | Greiner bio-one | 210261 | |
| filter tips 1 ml | Axygen | TF-1000-R-S | |
| filter tips 10 µl | Axygen | TF-10-R-S | |
| filter tips 100 µl | Axygen | TF-100-R-S | |
| filter tips 20 µl | Axygen | TF-20-R-S | |
| filter tips 200 µl | Axygen | TF-200-R-S | |
| microcentrifuge tubes 1.5 ml | SARSTEDT | 72.690.001 | |
| Propidium iodide | Sigma | P4170-100MG | |
| sequencing company | Novogene | en.novogene.com |
References
- Van Verk, M. C., Hickman, R., Corné, M. J., Pieterse, M., Van Wees, S. C. RNA-Seq: Revelation Of The Messengers. Trends In Plant Science. 18 (4), 175-179 (2013).
- Libault, M., Pingault, L., Zogli, P., Schiefelbein, J. Plant Systems Biology At The Single-Cell Level. Trends In Plant Science. 22 (11), 949-960 (2017).
- Mustroph, A., et al. Profiling Translatomes Of Discrete Cell Populations Resolves Altered Cellular Priorities During Hypoxia In Arabidopsis. Proceedings Of The National Academy Of Sciences Of The United States Of America. 106 (44), 18843-18848 (2009).
- Karve, R., Iyer-Pascuzzi, A. S. Digging Deeper: High-Resolution Genome-Scale Data Yields New Insights Into Root Biology. Current Opinion In Plant Biology. 24, 24-30 (2015).
- Warner, J. R., Knopf, P. M., Rich, A. A Multiple Ribosomal Structure In Protein Synthesis. Proceedings of The National Academy of Sciences of The United States of America. 49 (1), 122-129 (1963).
- Gautam, V., Sarkar, A. K. Laser Assisted Microdissection, An Efficient Technique To Understand Tissue Specific Gene Expression Patterns And Functional Genomics In Plants. Molecular Biotechnology. 57 (4), 299-308 (2015).
- Bargmann, B. O. R., Birnbaum, K. D. Fluorescence Activated Cell Sorting Of Plant Protoplasts. Journal of Visualized Experiments. (36), e1673 (2010).
- Deal, R. B., Henikoff, S. The Intact Method For Cell Type-Specific Gene Expression And Chromatin Profiling In Arabidopsis Thaliana. Nature Protocols. 6 (1), 56-68 (2011).
- Dougherty, J. D. The Expanding Toolkit Of Translating Ribosome Affinity Purification. The Journal of Neuroscience: The Official Journal Of The Society For Neuroscience. 37 (50), 12079-12087 (2017).
- Mustroph, A., Juntawong, P., Bailey-Serres, J. Isolation Of Plant Polysomal mRNA By Differential Centrifugation And Ribosome Immunopurification Methods. Methods in Molecular Biology. 553, 109-126 (2009).
- Matsushima, W., et al. SLAM-ITseq: Sequencing Cell Type-Specific Transcriptomes Without Cell Sorting. Development. 145 (13), (2018).
- Basnet, H., et al. Flura-Seq Identifies Organ-Specific Metabolic Adaptations During Early Metastatic Colonization. Elife. 8, (2019).
- Rodriques, S. G., et al. Slide-Seq: A Scalable Technology For Measuring Genome-Wide Expression At High Spatial Resolution. Science. 363 (6434), 1463-1467 (2019).
- Fazal, F. M., et al. Atlas Of Subcellular RNA Localization Revealed By Apex-Seq. Cell. 178 (2), 473-490 (2019).
- Slane, D., Bayer, M., Kaufmann, K., Mueller-Roeber, B. Cell Type-Specific Gene Expression Profiling Using Fluorescence-Activated Nuclear Sorting. Plant Gene Regulatory Networks: Methods And Protocols. , 27-35 (2017).
- Zanetti, M. E., Chang, I. F., Gong, F., Galbraith, D. W., Bailey-Serres, J. Immunopurification Of Polyribosomal Complexes Of Arabidopsis For Global Analysis Of Gene Expression. Plant Physiology. 138 (2), 624-635 (2005).
- King, H. A., Gerber, A. P. Translatome Profiling: Methods For Genome-Scale Analysis Of mRNA Translation. Briefings In Functional Genomics. 15 (1), 22-31 (2016).
- Mašek, T., Valášek, L., Pospíšek, M., Nielsen, H. Polysome Analysis And RNA Purification From Sucrose Gradients. RNA: Methods And Protocols. , 293-309 (2011).
- Heiman, M., et al. A Translational Profiling Approach For The Molecular Characterization Of Cns Cell Types. Cell. 135 (4), 738-748 (2008).
- Halbeisen, R. E., Scherrer, T., Gerber, A. P. Affinity Purification Of Ribosomes To Access The Translatome. Methods. 48 (3), 306-310 (2009).
- Thomas, A., et al. A Versatile Method For Cell-Specific Profiling Of Translated mRNAs In Drosophila. Plos One. 7 (7), e40276 (2012).
- Watson, F. L., et al. Cell Type-Specific Translational Profiling In The Xenopus Laevis Retina. Developmental Dynamics. 241 (12), 1960-1972 (2012).
- Lam, P. Y., Harvie, E. A., Huttenlocher, A. Heat Shock Modulates Neutrophil Motility In Zebrafish. Plos One. 8 (12), e84436 (2013).
- Fang, Y., et al. Translational Profiling Of Cardiomyocytes Identifies An Early Jak1/Stat3 Injury Response Required For Zebrafish Heart Regeneration. Proceedings Of The National Academy Of Sciences Of The United States Of America. 110 (33), 13416-13421 (2013).
- Mustroph, A., Zanetti, M. E., Girke, T., Bailey-Serres, J. Isolation And Analysis Of mRNAs From Specific Cell Types Of Plants By Ribosome Immunopurification. Methods In Molecular Biology. 959, 277-302 (2013).
- Monshausen, G. B., Gilroy, S. Feeling Green: Mechanosensing In Plants. Trends In Cell Biology. 19 (5), 228-235 (2009).
- Day, R. C., Grossniklaus, U., Macknight, R. C. Be More Specific! Laser-Assisted Microdissection Of Plant Cells. Trends In Plant Science. 10 (8), 397-406 (2005).
- Sheen, J. Signal Transduction In Maize And Arabidopsis Mesophyll Protoplasts. Plant Physiology. 127 (4), 1466-1475 (2001).
- Datta, S., et al. Laser Capture Microdissection: Big Data From Small Samples. Histology And Histopathology. 30 (11), 1255-1269 (2015).
- Birnbaum, K., et al. A Gene Expression Map Of The Arabidopsis Root. Science. 302 (5652), 1956 (2003).
- Hamant, O., Haswell, E. S. Life Behind The Wall: Sensing Mechanical Cues In Plants. BMC Biology. 15 (1), 1354 (2017).
- Vragović, K., et al. Translatome Analyses Capture Of Opposing Tissue-Specific Brassinosteroid Signals Orchestrating Root Meristem Differentiation. Proceedings of The National Academy of Sciences of the United States of America. 112 (3), 923-928 (2015).
- Wang, Y., Jiao, Y. Translating Ribosome Affinity Purification (Trap) For Cell-Specific Translation Profiling In Developing Flowers. Methods In Molecular Biology. 1110, 323-328 (2014).
- Sablok, G., Powell, J. J., Kazan, K. Emerging Roles And Landscape Of Translating mRNAs In Plants. Frontiers in Plant Science. 8, 1443 (2017).
- Ron, M., et al. Hairy Root Transformation Using Agrobacterium Rhizogenes As A Tool For Exploring Cell Type-Specific Gene Expression And Function Using Tomato As A Model. Plant Physiology. 166 (2), 455-469 (2014).
- Reynoso, M. A., et al. Evolutionary Flexibility In Flooding Response Circuitry In Angiosperms. Science. 365 (6459), 1291-1295 (2019).
- Dolan, L., et al. Cellular Organisation Of The Arabidopsis Thaliana Root. Development. 119 (1), 71 (1993).
- Ristova, D., Barbez, E. . Root Development. , (2018).
- Shekhar, V., Stӧckle, D., Thellmann, M., Vermeer, J. E. M. The Role Of Plant Root Systems In Evolutionary Adaptation. Current Topics in Developmental Biology. 131, 55-80 (2019).
- Malamy, J. E., Benfey, P. N. Down And Out In Arabidopsis: The Formation Of Lateral Roots. Trends in Plant Science. 2 (10), 390-396 (1997).
- de Smet, I., et al. Bimodular Auxin Response Controls Organogenesis In Arabidopsis. Proceedings of the National Academy of Sciences of The United States of America. 107 (6), 2705-2710 (2010).
- Péret, B., et al. Arabidopsis Lateral Root Development: An Emerging Story. Trends In Plant Science. 14 (7), 399-408 (2009).
- Vilches-Barro, A., Maizel, A. Talking Through Walls: Mechanisms Of Lateral Root Emergence In Arabidopsis Thaliana. Current Opinion in Plant Biology. 23, 31-38 (2015).
- Porco, S., et al. Lateral Root Emergence In Arabidopsis Is Dependent On Transcription Factor Lbd29 Regulation Of Auxin Influx Carrier Lax3. Development. 143 (18), 3340-3349 (2016).
- Stoeckle, D., Thellmann, M., Vermeer, J. E. Breakout-Lateral Root Emergence In Arabidopsis Thaliana. Current Opinion in Plant Biology. 41, 67-72 (2018).
- Banda, J., et al. Lateral Root Formation In Arabidopsis: A Well-Ordered Lrexit. Trends in Plant Science. 24 (9), 826-839 (2019).
- Vermeer, J. E. M., et al. A Spatial Accommodation By Neighboring Cells Is Required For Organ Initiation In Arabidopsis. Science. 343 (6167), 178-183 (2014).
- Vanneste, S., et al. Cell Cycle Progression In The Pericycle Is Not Sufficient For Solitary Root/Iaa14-Mediated Lateral Root Initiation In Arabidopsis Thaliana. The Plant Cell. 17 (11), 3035-3050 (2005).
- Marques-Bueno, M. M., et al. A Versatile Multisite Gateway-Compatible Promoter And Transgenic Line Collection For Cell Type-Specific Functional Genomics In Arabidopsis. The Plant Journal : For Cell and Molecular Biology. 85 (2), 320-333 (2016).
- Shimada, T. L., Shimada, T., Hara-Nishimura, I. A Rapid And Non-Destructive Screenable Marker, Fast, For Identifying Transformed Seeds Of Arabidopsis Thaliana. The Plant Journal : For Cell and Molecular Biology. 61 (3), 519-528 (2010).
- Clough, S. J., Bent, A. F. Floral Dip: A Simplified Method For Agrobacterium Mediated Transformation Of Arabidopsis Thaliana. The Plant Journal. 16 (6), 735-743 (1998).
- Lindsey, B. E., Rivero, L., Calhoun, C. S., Grotewold, E., Brkljacic, J. Standardized Method For High-Throughput Sterilization Of Arabidopsis Seeds. Journal Of Visualized Experiments. (128), e56587 (2017).
- Andersen, T. G., et al. Diffusible Repression Of Cytokinin Signalling Produces Endodermal Symmetry And Passage Cells. Nature. 555, 529-533 (2018).
- Schroeder, A., et al. The Rin: An Rna Integrity Number For Assigning Integrity Values To Rna Measurements. BMC Molecular Biology. 7, 3 (2006).
- Vragović, K., Bartom, E., Savaldi-Goldstein, S. Quantitation Of Cell Type-Specific Responses To Brassinosteroid By Deep Sequencing Of Polysome-Associated Polyadenylated RNA. Methods in Molecular Biology. 1564, 81-102 (2017).
- Bertin, B., Renaud, Y., Aradhya, R., Jagla, K., Junion, G. Trap-Rc, Translating Ribosome Affinity Purification From Rare Cell Populations Of Drosophila Embryos. Journal Of Visualized Experiments. (103), e52985 (2015).
- Livak, K. J., Schmittgen, T. D. Analysis Of Relative Gene Expression Data Using Real-Time Quantitative PCR And The 2(-Delta Delta C(T)) Method. Methods. 25 (4), 402-408 (2001).
- Jiao, Y., Meyerowitz, E. M. Cell-Type Specific Analysis Of Translating Rnas In Developing Flowers Reveals New Levels Of Control. Molecular Systems Biology. 6, 419 (2010).
- Tian, C., et al. A Gene Expression Map Of Shoot Domains Reveals Regulatory Mechanisms. Nature Communications. 10 (1), 141 (2019).
- Townsley, B. T., Covington, M. F., Ichihashi, Y., Zumstein, K., Sinha, N. R. Brad-Seq: Breath Adapter Directional Sequencing: A Streamlined, Ultra-Simple And Fast Library Preparation Protocol For Strand Specific mRNA Library Construction. Frontiers in Plant Science. 6, 366 (2015).
- Song, Y., et al. A Comparative Analysis Of Library Prep Approaches For Sequencing Low Input Translatome Samples. BMC Genomics. 19 (1), 696 (2018).
- Basu, D., Haswell, E. S. Plant Mechanosensitive Ion Channels: An Ocean Of Possibilities. Current Opinion in Plant Biology. 40, 43-48 (2017).
- Brady, S. M., et al. A High-Resolution Root Spatiotemporal Map Reveals Dominant Expression Patterns. Science. 318 (5851), 801 (2007).

