細胞型特異的スケールでのシロイヌナズナ・タリアナの開発を調査するためのリボソーム親和性精製(TRAP)の翻訳
Summary
リボソームアフィニティー精製(TRAP)を翻訳すると、臓器や組織の最小限の処理で開発プログラムを解剖する可能性があります。このプロトコルは、緑色蛍光タンパク質(GFP)標識リボソームサブユニットを標的とした細胞から高品質のRNAを生成します。qRT-PCR や RNA-seq などのダウンストリーム解析ツールは、組織および細胞の種類特異的発現プロファイルを明らかにします。
Abstract
本稿では、リボソーム親和精製(TRAP)法と連続して最適化された低入力ライブラリ調製を通じて、異なるシロイヌナナの根細胞型から翻訳データを取得するための実践的な指示を与える。
原料として、適切なプロモーターを用いることで、細胞型特異的な方法でGFPタグリボソームタンパク質RPL18を発現する植物ラインを採用しています。免疫精製とRNA抽出の前に、組織は凍結され、組織の完全性を維持し、同時に高い時間分解能で時系列研究を実行することができます。特に、細胞壁構造はそのまま残っており、これは、異なる細胞集団を単離するために組織をプロトパロントに依存する蛍光活性化細胞選別ベースのアプローチのような代替手順の大きな欠点である。さらに、レーザー捕捉マイクロ解剖ベースの技術のように組織固定は必要なく、高品質のRNAを得ることができます。
しかし、細胞の亜集団からのサンプリングと、ポリソーム関連RNAの単離のみがRNAの収量を著しく制限します。したがって、RNA-seqによるデータ取得を成功させるためには十分に感受性の高いライブラリ調製方法を適用する必要がある。
TRAPは、多くの発達プロセスが細胞壁関連および機械的シグナル伝達経路を伴うため、植物研究に理想的なツールを提供します。特定の細胞集団を標的とするプロモーターの使用は、臓器と単一細胞レベルの間のギャップを埋めており、その結果、解像度の低下や非常に高いコストに苦しんでいます。ここでは、トラップを適用して、横根形成における細胞細胞通信を研究する。
Introduction
次世代シーケンシング技術の応用の増加によって、発生生物学における空間分解能が増強される可能性があります。現代の研究は、単一細胞レベル,,1、2、3、42ではないにしても、特殊な細胞タイプに組織1を解剖することを目的としています。34この処理のために、過去 50 年間に多数の異なる方法が考案されています (図1A)5、6、,7,、8 、9、109、11、12、13、1414、15.
植物科学の多くのツールは、動物研究で開拓された技術の適応されています。これは、ここで詳しく紹介する方法には当てはまらない。2005年、タンパク質翻訳の強力な背景を備えたBailey-Serres Labは、その後の親和性精製のためにリボソームタンパク質の設計に着手しました。したがって、スクロース勾配を有する超遠心分離に基づく時間と労力を要するポリソームプロファイリングを回避することができ、1960年代17、18,18年以来リボソームの翻訳を評価するために使用された。この方法は、その後、翻訳リボソーム親和性精製(TRAP)16と呼ばれている。植物の翻訳研究に成功した後、Heimanらの動物19と他の人のためのTRAPを適応させた酵母20、ショウジョウバエ21、ゼノプス22およびゼブラフィッシュ23、24,24への応用を拡張した。
モデルシステムの遺伝子改変はTRAPの前提条件であり、その適用を遺伝的変換に適した種に限定しますが、この異議を、特別な関心を持ち、無傷の組織/器官25から分離することが非常に困難な細胞のサブセット(例えば、マウス脳内の高度に分岐した樹状細胞または感染した植物組織の真菌性催眠細胞)に同時に利用することができます。植物では、全ての細胞が、静水性骨格26の基礎となる細胞壁を介して所定の位置に保持される。このマトリックスから植物細胞を解放するために、科学者たちはレーザー捕捉マイクロディション(LCM)27を介して周囲の組織から細胞を物理的27に切断するか、または細胞壁28の酵素消化を行った。後者の細胞の中でも、いわゆるプロトプラストは、目的の集団が蛍光標識され、蛍光活性化細胞選別(FACS)7を介して分離することができる7。LCMは通常、試料を固定してワックスに埋め込む必要があり、最終的にはRNA29の品質が低下する。FACSベースの方法は高品質のRNAを生み出すが、プロプロトバテナのプロセス自体が遺伝子発現30の違いを導入し、改変された厚い二次細胞壁を有する組織は治療が困難であると悪名高い。さらに、植物における多くの発達過程は機械的に伝達される信号に依存すると仮定され、したがって細胞壁の完全性は最も重要である31。核のレベルで動作することによって細胞単離を回避するためのショートカットを使用する2つの方法は、蛍光活性化核選別(FANS)および特定の細胞タイプ(INTACT)でタグ付けされた核の単離である。TRAPのように、彼らは細胞型特異的プロモーターを使用して核をマークし、その後、並べ替えまたはプルダウンを介して濃縮され、それぞれ8,8、15。これらすべてのアプローチの主要な課題は、組織中の細胞のサブセットから十分なRNA材料を得るということです。TRAP は細胞RNAのほんの一部しか捕捉しなかね、サンプル収集は大きなボトルネックです。したがって、特に、低入力量から高品質のデータを生成するためには、機密性の高いライブラリ準備プロトコルが必要です。
その設立以来、TRAPはDNAマイクロアレイと組み合わせて使用されているか、または、シーケンシングコストが近年大幅に低下したため、RNA-seq1010、32、3332,33が使用されている。Sablokら34でレビューされた多数の研究の質問が既に解明されています。我々は、特定の細胞タイプを標的に異なるプロモーターを組み合わせる場合、この技術は非常に汎用性が高いため、今後数年間でより多くの報告が続くと確信しています。最終的には、これは誘導可能な方法でも行われ、多くの生物的および無生物的ストレス要因に対する植物の反応を調査することと組み合わされる可能性があります。さらに、安定したトランスジェニックラインが利用できないところでは、毛深い根発現系もトマトおよびメディカゴ35,36,36でTRAPを行うためにもうまく使用されている。
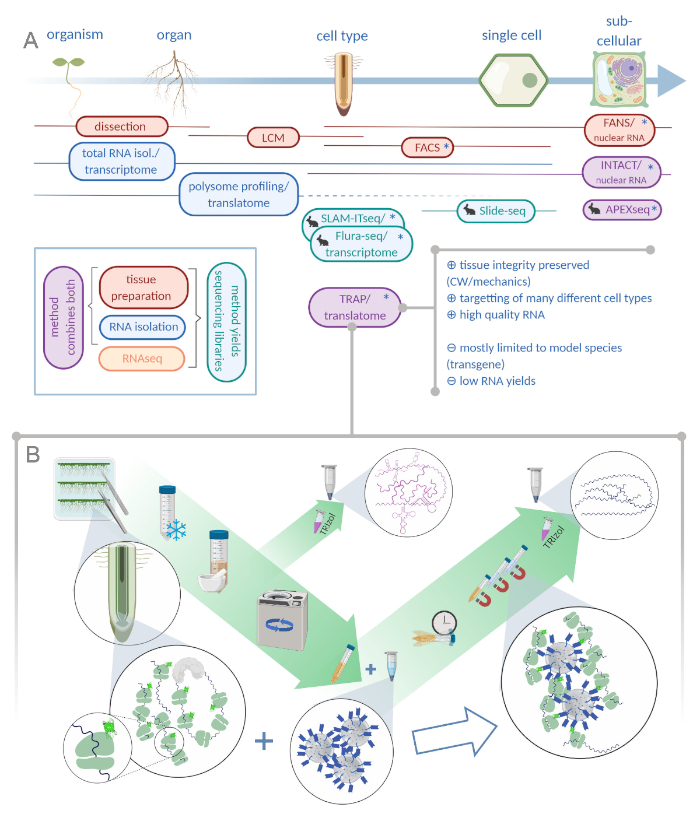
図1:リボソームアフィニティー精製(TRAP)の翻訳は、「オミックス」分析ポートフォリオを補完します。A.分析精度のレベルを上げると、単一細胞または細胞内の分解能まで、多くの方法またはそれらの組み合わせによって達成できます。このスキームは、植物および動物分野で現在利用可能なツールの概要を示します。細胞分解能での組織の収集は、LCMやFACSのようなプロトコルによって達成され、標準のトランスクリプトームまたはポリソームプロファイリング/翻訳分析に結合されます。TRAPとINTACTは、エピトープタグに基づいているため、組織捕捉とRNAの分離の両方を統合します。しかしながら、INTACTサンプルは細胞核のみを構成し、したがって、トランスクリプトーム分析の特別な場合を構成する。小さなウサギのアイコンは、動物分野で新たに開発された方法を示しています:SLAM-ITseqとFlura-seqは、寛容な酵素を発現する細胞内の改変ウラシル塩基を持つ新生RNAの代謝ターゲッティングに依存していますが、Slide-seqは、細胞範囲内の位置情報を提供するDNAバーコード付きコーティングガラススライドを使用しています。特定の細胞内区画で RNA をサンプリングするために、APEX-seq では近接標識アプローチが続きます。特に、分解能の向上にはトランスジェニック材料(アスタリスク)の生成が必要なことが多く、モデル種に対してこれらの方法が主に使用される。TRAPは、細胞壁(CW)やメカニックシグナリング、およびCWマトリックスから放出することが困難な細胞種を含む植物科学研究に特に適しています。B. TRAP手順の詳細なウェットラボステップ:異なる細胞タイプ(例えば根内胚葉)でGFPタグ付きリボソームタンパク質を発現する苗は、7日間ペトリ皿で栽培され、根材料はスナップ凍結によって収穫される。全RNA制御サンプルは、遠心分離によって破片をペレットする前に、均質化された粗抽出物から収集されます。磁性抗GFPビーズをクリアした抽出物に添加して、免疫沈降を行います。インキュベートと3つの洗浄工程の後、ポリソーム関連RNA(TRAP/ポリソームRNA)はフェノール-クロロホルム抽出を介して直接得られます。LCM:レーザーキャプチャマイクロディスセクション、FACS/ファン:蛍光活性化細胞/核選別、 APEX-seq:工学的アスコルビンペルオキシダーゼに基づく方法、INTACT:特定の細胞型にタグ付けされた核の単離、SLAM-ITseq:チオール(SH)結合アルキル化組織中のRNAの代謝シーケンシングに対する、Flura-seq:フルオロシル標識RNAシーケンシング(フルオロシル標識RNAシーケンシング Biorender.com)をクリックしてください。
この記事の目的は、TRAP メソッドの詳細な説明を提供し、重要な手順を強調し、可能なライブラリ準備方法のガイダンスを提供することです。
一般的なTRAP実験は、基本的に次のステップで構成されます(図1Bも参照):(1)リボソームタグ構造のクローニング、トランスジェニックラインの生産と選択、種子の成長と増量、ストレス適用/処理(オプション)および組織収穫を含む植物材料の調製。(2)粗抽出物の組織均質化および除去、ビーズ洗浄および免疫精製、および洗浄工程を含む免疫精製;(3) RNA抽出および品質評価(4)図書館の準備。
シロイヌナズナルートは、モデルプラント37,38として導入されて以来、38植物開発を研究するモデルシステムとなっています。ここでは、TRAPの適用は植物側面根開発の文脈で紹介される。植物では、全体の根系の蓄積は、このプログラムの実行に依存し、したがって、生物の生存のために非常に重要である39.シロイヌナズナシスでは、横根はxylem血管の隣に存在するペリサイクル組織に由来し、したがってxylem極ペリサイクル(XPP;図2Cを参照)40と呼ばれます。根の奥深くに位置する一部のXPP細胞は、創始者の細胞の同一性を獲得し、局所ホルモンの引き金に基づいて、反クリン41を腫脹して分裂させることによって増殖し始める。しかし、硬質細胞壁マトリックスの存在により、このプロセスは周囲の組織に機械的なストレスを与えます。特に、上方の内皮は、根根成長軸42、43、44,43,の邪魔であるように影響を受ける。実際、新たに形成されたプリモジウムは、上層の内皮細胞(図2C2)を介して成長する必要がありますが、皮質および表皮細胞は、プリモジウムが最終的に45、46,46を出現させるために脇に押し出されるだけです。私たちの研究室での最近の研究は、内皮が積極的にペリサイクルの増殖に対応するために貢献していることを示しています。内皮ホルモンシグナル伝達の標的遮断は、XPP細胞47における非常に最初の分裂を阻害するのに十分である。したがって、ペリサイクル-子宮内皮通信は、シロイヌナズナの横根開発のための非常に初期のチェックポイントを構成する。しかし、このクロストークがどのように行われるかは分かっていません。この謎を解明するために、XPP細胞と内皮細胞を標的とするTRAP-seqアプローチを選択しました。横根プログラムの細胞を濃縮するために、オーキシンアナログ(1-ナフタレ酢酸、NAA)48を外因的に適用することによってホルモントリガーを模倣し、同時に側根形成の48初期段階を時間的に解決することを可能にした。
Protocol
Representative Results
Discussion
RPL18ローカリゼーションパターンの検証
TRAP実験のデータの誤解釈を避けるために重要なのは、タグ付きリボソームサブユニットの適切な発現パターンです。従って、RPL18へのエピトープタグとしてのGFPの組み込みは、所望の発現パターンの検証を非常に優雅に可能にし、連続して、同じ組織からポリソーム分画のプルダウンを可能にする。適切なプロモーターパターンを保証?…
Disclosures
The authors have nothing to disclose.
Acknowledgements
このプロジェクトの初期段階で、チューリッヒ遺伝ダイバーシティセンターのジャン=クロード・ウォルサーに重要な専門家のアドバイスをしてくれたことに感謝します。フェルメール研究所での研究は、スイス国立科学財団(SNSF)からSNF教授助成(PP00P3_157524)とR’EQUIP機器助成金(316030_164086)によって支援されました。
Materials
| Sterilization | |||
| bleach, 13% | Sigma | 71696 | |
| beaker | VWR | 214-1172/74/75 | |
| desiccator with porcelaine plate (DURAN) | Sigma/Merck | Z317454-1EA/Z317594-1EA | |
| EtOH, p.a. | Honeywell | 02860-1L | |
| HCl, 37% | Roth | 4625.1 | |
| Tween 20 | Sigma | P9416 | |
| Plate growth + harvesting | |||
| MS salts, basal salt mixture, incl. MES buffer | Duchefa | M0254 | |
| agar plant for cell culture | Applichem/Panreac | A2111.1000 | |
| DMSO | Sigma | D4540 | |
| forcepts | Rubis Switzerland | 5-SA model | |
| KOH | Fluka | 60370 | |
| micropore/surgical tape | 3M | 1530-0 | |
| NAA | Duchefa | N0903 | |
| petri dishes 120×120 mm | Greiner bio-one | 688102 | |
| scalpel | VWR/Swann-Morton | 233-5454 | |
| tissues, neutral, two-layered | any supplier of your choice | ||
| Immunoprecipitation | |||
| GFP-beads: gtma-100 GFP-Trap_MA | Chromotek | e.g. gtma-100 | |
| Brij-35 | Sigma | P1254-500G | |
| centrifuge tubes (in accordance with centrifuge) | Beckman Coulter | 357001 | |
| Chloramphenicol | Applichem | C0378-25G | |
| cotton gloves | VWR | 113-7355 | |
| Cycloheximide, HPLC grade | Sigma | 01810-1G | |
| DEPC | VWR | E174 | might have long delivery times |
| DTT | Fluka | 43815 | |
| EGTA | Sigma | 3054.3 | |
| homogenizers DUALL 23 | KONTES GLASS CO (via VWR) | SCERSP885450-0023 (set) | SCERSP885451-0023 pestle only – SCERSP885452-0023 cylinder only; long delivery times |
| Igepal CA-360 | Sigma | I3021-100ml | |
| KCl | Sigma | 60130 | |
| MgCl2 hexahydrat | Roth | 2189.2 | |
| mortar and pestle | VWR | 470148-960 & 470019-978 | |
| PMSF | Roche | 10 837 091 001 | |
| Polyoxyethylene-(10)-tridecylether/PTE | Sigma | P2393-500G | |
| RNase-free water | Roth | T143.3 | |
| RNAZap | Thermo Fisher | AM9780/AM9782 | for cleaning surfaces |
| Tris, >99.3% | Roth | AE15.3 | |
| Triton X-100 | Fluka | T8787-250ml | |
| Tween 20 | Sigma | P9416-100ml | |
| RNA extraction | |||
| 2-Propanol, p.a. | Sigma | 33539-1L-GL-R | |
| Chloroform, HPLC grade | Scharlau | CL02181000 | |
| EtOH, p.a. | Honeywell | 02860-1L | |
| low-retention microcentrifuge tubes, 1.5 ml | Eppendorf/Sigma | Z666548-250EA | LoBind |
| RNase-free DNase set | Qiagen | 79254 | |
| RNeasy MiniElute Cleanup Kit | Qiagen | 74204 | |
| TRIzol reagent | ThermoFisher/Ambion | 15596018 | |
| Library preparation | |||
| 15/50 mL Tube Magnetic Separator | Abraxis | PN 472250 | |
| AMPure beads | Beckman Coulter | A63881 | |
| Index Kit A | Illumina | FC-131-2001 | |
| Index Kit D | Illumina | FC-131-2004 | |
| neodymium magnets | Amazon/other | 6 x 1.5 mm range: N42 (NdFeB) | |
| Nextera XT kit | Illumina | FC-131-1024/1096 | https://emea.support.illumina.com/ |
| PCR strips | ThermoScientific | AB-0266 | |
| SMARTer v4 kit | Takara Bioscience | 634892 | https://www.takarabio.com/ |
| Bioanalyzer | Agilent | 2100 Bioanalyzer Instrument | specialized equipment for RNA/DNA quality control |
| Tapestation | Agilent | 4200 Tapestation Instrument | specialized equipment for RNA/DNA quality control |
| Fragment Analyzer | Agilent | 5400 Fragment Analyzer System | specialized equipment for RNA/DNA quality control (high throughput) |
| LabChip | PerkinElmer | LabChip GX Touch Nucleic Acid Analyzer | specialized equipment for RNA/DNA quality control (high throughput) |
| Qubit 4 Fluorometer | ThermoFisher | Q33239 | specialized equipment for RNA/DNA concentration determination |
| qRT-PCR | |||
| GATA23 | Microsynth | fwd: AGTGAGAATGAA AGAAGAGAAGGG; rev: GTGGCTGCGAAT AATATGAATACC |
|
| GH3.3 | Microsynth | fwd: CAAACCAATCCT CCAAATGAC; rev: ACTTATCCGCAA CCCGACT |
|
| LBD29 | Microsynth | fwd: TCTCCAACAACA GGTTGTGAAT; rev: AAGGAGCCTTAG TAGTGTCTCCA |
|
| UBC21 | Microsynth | fwd: TGCGACTCAGGG AATCTTCT; rev: TCATCCTTTCTT AGGCATAGCG |
|
| SsoAdvanced Universal SYBR Green | Bio-Rad | #172-5270 | |
| iScript Adv cDNA Kit | Bio-Rad | #172-5038 | |
| miscellaneous | |||
| Falcon tubes 15 ml, Cellstar | Greiner bio-one | 188261 | |
| Falcon tubes 50 ml, Cellstar | Greiner bio-one | 210261 | |
| filter tips 1 ml | Axygen | TF-1000-R-S | |
| filter tips 10 µl | Axygen | TF-10-R-S | |
| filter tips 100 µl | Axygen | TF-100-R-S | |
| filter tips 20 µl | Axygen | TF-20-R-S | |
| filter tips 200 µl | Axygen | TF-200-R-S | |
| microcentrifuge tubes 1.5 ml | SARSTEDT | 72.690.001 | |
| Propidium iodide | Sigma | P4170-100MG | |
| sequencing company | Novogene | en.novogene.com |
References
- Van Verk, M. C., Hickman, R., Corné, M. J., Pieterse, M., Van Wees, S. C. RNA-Seq: Revelation Of The Messengers. Trends In Plant Science. 18 (4), 175-179 (2013).
- Libault, M., Pingault, L., Zogli, P., Schiefelbein, J. Plant Systems Biology At The Single-Cell Level. Trends In Plant Science. 22 (11), 949-960 (2017).
- Mustroph, A., et al. Profiling Translatomes Of Discrete Cell Populations Resolves Altered Cellular Priorities During Hypoxia In Arabidopsis. Proceedings Of The National Academy Of Sciences Of The United States Of America. 106 (44), 18843-18848 (2009).
- Karve, R., Iyer-Pascuzzi, A. S. Digging Deeper: High-Resolution Genome-Scale Data Yields New Insights Into Root Biology. Current Opinion In Plant Biology. 24, 24-30 (2015).
- Warner, J. R., Knopf, P. M., Rich, A. A Multiple Ribosomal Structure In Protein Synthesis. Proceedings of The National Academy of Sciences of The United States of America. 49 (1), 122-129 (1963).
- Gautam, V., Sarkar, A. K. Laser Assisted Microdissection, An Efficient Technique To Understand Tissue Specific Gene Expression Patterns And Functional Genomics In Plants. Molecular Biotechnology. 57 (4), 299-308 (2015).
- Bargmann, B. O. R., Birnbaum, K. D. Fluorescence Activated Cell Sorting Of Plant Protoplasts. Journal of Visualized Experiments. (36), e1673 (2010).
- Deal, R. B., Henikoff, S. The Intact Method For Cell Type-Specific Gene Expression And Chromatin Profiling In Arabidopsis Thaliana. Nature Protocols. 6 (1), 56-68 (2011).
- Dougherty, J. D. The Expanding Toolkit Of Translating Ribosome Affinity Purification. The Journal of Neuroscience: The Official Journal Of The Society For Neuroscience. 37 (50), 12079-12087 (2017).
- Mustroph, A., Juntawong, P., Bailey-Serres, J. Isolation Of Plant Polysomal mRNA By Differential Centrifugation And Ribosome Immunopurification Methods. Methods in Molecular Biology. 553, 109-126 (2009).
- Matsushima, W., et al. SLAM-ITseq: Sequencing Cell Type-Specific Transcriptomes Without Cell Sorting. Development. 145 (13), (2018).
- Basnet, H., et al. Flura-Seq Identifies Organ-Specific Metabolic Adaptations During Early Metastatic Colonization. Elife. 8, (2019).
- Rodriques, S. G., et al. Slide-Seq: A Scalable Technology For Measuring Genome-Wide Expression At High Spatial Resolution. Science. 363 (6434), 1463-1467 (2019).
- Fazal, F. M., et al. Atlas Of Subcellular RNA Localization Revealed By Apex-Seq. Cell. 178 (2), 473-490 (2019).
- Slane, D., Bayer, M., Kaufmann, K., Mueller-Roeber, B. Cell Type-Specific Gene Expression Profiling Using Fluorescence-Activated Nuclear Sorting. Plant Gene Regulatory Networks: Methods And Protocols. , 27-35 (2017).
- Zanetti, M. E., Chang, I. F., Gong, F., Galbraith, D. W., Bailey-Serres, J. Immunopurification Of Polyribosomal Complexes Of Arabidopsis For Global Analysis Of Gene Expression. Plant Physiology. 138 (2), 624-635 (2005).
- King, H. A., Gerber, A. P. Translatome Profiling: Methods For Genome-Scale Analysis Of mRNA Translation. Briefings In Functional Genomics. 15 (1), 22-31 (2016).
- Mašek, T., Valášek, L., Pospíšek, M., Nielsen, H. Polysome Analysis And RNA Purification From Sucrose Gradients. RNA: Methods And Protocols. , 293-309 (2011).
- Heiman, M., et al. A Translational Profiling Approach For The Molecular Characterization Of Cns Cell Types. Cell. 135 (4), 738-748 (2008).
- Halbeisen, R. E., Scherrer, T., Gerber, A. P. Affinity Purification Of Ribosomes To Access The Translatome. Methods. 48 (3), 306-310 (2009).
- Thomas, A., et al. A Versatile Method For Cell-Specific Profiling Of Translated mRNAs In Drosophila. Plos One. 7 (7), e40276 (2012).
- Watson, F. L., et al. Cell Type-Specific Translational Profiling In The Xenopus Laevis Retina. Developmental Dynamics. 241 (12), 1960-1972 (2012).
- Lam, P. Y., Harvie, E. A., Huttenlocher, A. Heat Shock Modulates Neutrophil Motility In Zebrafish. Plos One. 8 (12), e84436 (2013).
- Fang, Y., et al. Translational Profiling Of Cardiomyocytes Identifies An Early Jak1/Stat3 Injury Response Required For Zebrafish Heart Regeneration. Proceedings Of The National Academy Of Sciences Of The United States Of America. 110 (33), 13416-13421 (2013).
- Mustroph, A., Zanetti, M. E., Girke, T., Bailey-Serres, J. Isolation And Analysis Of mRNAs From Specific Cell Types Of Plants By Ribosome Immunopurification. Methods In Molecular Biology. 959, 277-302 (2013).
- Monshausen, G. B., Gilroy, S. Feeling Green: Mechanosensing In Plants. Trends In Cell Biology. 19 (5), 228-235 (2009).
- Day, R. C., Grossniklaus, U., Macknight, R. C. Be More Specific! Laser-Assisted Microdissection Of Plant Cells. Trends In Plant Science. 10 (8), 397-406 (2005).
- Sheen, J. Signal Transduction In Maize And Arabidopsis Mesophyll Protoplasts. Plant Physiology. 127 (4), 1466-1475 (2001).
- Datta, S., et al. Laser Capture Microdissection: Big Data From Small Samples. Histology And Histopathology. 30 (11), 1255-1269 (2015).
- Birnbaum, K., et al. A Gene Expression Map Of The Arabidopsis Root. Science. 302 (5652), 1956 (2003).
- Hamant, O., Haswell, E. S. Life Behind The Wall: Sensing Mechanical Cues In Plants. BMC Biology. 15 (1), 1354 (2017).
- Vragović, K., et al. Translatome Analyses Capture Of Opposing Tissue-Specific Brassinosteroid Signals Orchestrating Root Meristem Differentiation. Proceedings of The National Academy of Sciences of the United States of America. 112 (3), 923-928 (2015).
- Wang, Y., Jiao, Y. Translating Ribosome Affinity Purification (Trap) For Cell-Specific Translation Profiling In Developing Flowers. Methods In Molecular Biology. 1110, 323-328 (2014).
- Sablok, G., Powell, J. J., Kazan, K. Emerging Roles And Landscape Of Translating mRNAs In Plants. Frontiers in Plant Science. 8, 1443 (2017).
- Ron, M., et al. Hairy Root Transformation Using Agrobacterium Rhizogenes As A Tool For Exploring Cell Type-Specific Gene Expression And Function Using Tomato As A Model. Plant Physiology. 166 (2), 455-469 (2014).
- Reynoso, M. A., et al. Evolutionary Flexibility In Flooding Response Circuitry In Angiosperms. Science. 365 (6459), 1291-1295 (2019).
- Dolan, L., et al. Cellular Organisation Of The Arabidopsis Thaliana Root. Development. 119 (1), 71 (1993).
- Ristova, D., Barbez, E. . Root Development. , (2018).
- Shekhar, V., Stӧckle, D., Thellmann, M., Vermeer, J. E. M. The Role Of Plant Root Systems In Evolutionary Adaptation. Current Topics in Developmental Biology. 131, 55-80 (2019).
- Malamy, J. E., Benfey, P. N. Down And Out In Arabidopsis: The Formation Of Lateral Roots. Trends in Plant Science. 2 (10), 390-396 (1997).
- de Smet, I., et al. Bimodular Auxin Response Controls Organogenesis In Arabidopsis. Proceedings of the National Academy of Sciences of The United States of America. 107 (6), 2705-2710 (2010).
- Péret, B., et al. Arabidopsis Lateral Root Development: An Emerging Story. Trends In Plant Science. 14 (7), 399-408 (2009).
- Vilches-Barro, A., Maizel, A. Talking Through Walls: Mechanisms Of Lateral Root Emergence In Arabidopsis Thaliana. Current Opinion in Plant Biology. 23, 31-38 (2015).
- Porco, S., et al. Lateral Root Emergence In Arabidopsis Is Dependent On Transcription Factor Lbd29 Regulation Of Auxin Influx Carrier Lax3. Development. 143 (18), 3340-3349 (2016).
- Stoeckle, D., Thellmann, M., Vermeer, J. E. Breakout-Lateral Root Emergence In Arabidopsis Thaliana. Current Opinion in Plant Biology. 41, 67-72 (2018).
- Banda, J., et al. Lateral Root Formation In Arabidopsis: A Well-Ordered Lrexit. Trends in Plant Science. 24 (9), 826-839 (2019).
- Vermeer, J. E. M., et al. A Spatial Accommodation By Neighboring Cells Is Required For Organ Initiation In Arabidopsis. Science. 343 (6167), 178-183 (2014).
- Vanneste, S., et al. Cell Cycle Progression In The Pericycle Is Not Sufficient For Solitary Root/Iaa14-Mediated Lateral Root Initiation In Arabidopsis Thaliana. The Plant Cell. 17 (11), 3035-3050 (2005).
- Marques-Bueno, M. M., et al. A Versatile Multisite Gateway-Compatible Promoter And Transgenic Line Collection For Cell Type-Specific Functional Genomics In Arabidopsis. The Plant Journal : For Cell and Molecular Biology. 85 (2), 320-333 (2016).
- Shimada, T. L., Shimada, T., Hara-Nishimura, I. A Rapid And Non-Destructive Screenable Marker, Fast, For Identifying Transformed Seeds Of Arabidopsis Thaliana. The Plant Journal : For Cell and Molecular Biology. 61 (3), 519-528 (2010).
- Clough, S. J., Bent, A. F. Floral Dip: A Simplified Method For Agrobacterium Mediated Transformation Of Arabidopsis Thaliana. The Plant Journal. 16 (6), 735-743 (1998).
- Lindsey, B. E., Rivero, L., Calhoun, C. S., Grotewold, E., Brkljacic, J. Standardized Method For High-Throughput Sterilization Of Arabidopsis Seeds. Journal Of Visualized Experiments. (128), e56587 (2017).
- Andersen, T. G., et al. Diffusible Repression Of Cytokinin Signalling Produces Endodermal Symmetry And Passage Cells. Nature. 555, 529-533 (2018).
- Schroeder, A., et al. The Rin: An Rna Integrity Number For Assigning Integrity Values To Rna Measurements. BMC Molecular Biology. 7, 3 (2006).
- Vragović, K., Bartom, E., Savaldi-Goldstein, S. Quantitation Of Cell Type-Specific Responses To Brassinosteroid By Deep Sequencing Of Polysome-Associated Polyadenylated RNA. Methods in Molecular Biology. 1564, 81-102 (2017).
- Bertin, B., Renaud, Y., Aradhya, R., Jagla, K., Junion, G. Trap-Rc, Translating Ribosome Affinity Purification From Rare Cell Populations Of Drosophila Embryos. Journal Of Visualized Experiments. (103), e52985 (2015).
- Livak, K. J., Schmittgen, T. D. Analysis Of Relative Gene Expression Data Using Real-Time Quantitative PCR And The 2(-Delta Delta C(T)) Method. Methods. 25 (4), 402-408 (2001).
- Jiao, Y., Meyerowitz, E. M. Cell-Type Specific Analysis Of Translating Rnas In Developing Flowers Reveals New Levels Of Control. Molecular Systems Biology. 6, 419 (2010).
- Tian, C., et al. A Gene Expression Map Of Shoot Domains Reveals Regulatory Mechanisms. Nature Communications. 10 (1), 141 (2019).
- Townsley, B. T., Covington, M. F., Ichihashi, Y., Zumstein, K., Sinha, N. R. Brad-Seq: Breath Adapter Directional Sequencing: A Streamlined, Ultra-Simple And Fast Library Preparation Protocol For Strand Specific mRNA Library Construction. Frontiers in Plant Science. 6, 366 (2015).
- Song, Y., et al. A Comparative Analysis Of Library Prep Approaches For Sequencing Low Input Translatome Samples. BMC Genomics. 19 (1), 696 (2018).
- Basu, D., Haswell, E. S. Plant Mechanosensitive Ion Channels: An Ocean Of Possibilities. Current Opinion in Plant Biology. 40, 43-48 (2017).
- Brady, S. M., et al. A High-Resolution Root Spatiotemporal Map Reveals Dominant Expression Patterns. Science. 318 (5851), 801 (2007).

