RNAi-gemedieerde controle van Aflatoxines in Peanut: Methode om Mycotoxine Productie en transgenexpressie Analyseer de Peanut /<em> Aspergillus</em> Pathosystem
Summary
We tonen een werkwijze voor de analyse van aflatoxinen en transgenexpressie in pindazaden dat RNA-stoorsignalen bevatten voor silencing aflatoxine-synthese genen in de schimmel Aspergillus flavus. RNAi-gemedieerde regulering van mycotoxinen in planten is niet eerder gerapporteerd.
Abstract
De Voedsel- en Landbouworganisatie van de Verenigde Naties schat dat 25% van de voedselgewassen in de wereld zijn besmet met aflatoxinen. Dat vertegenwoordigt 100 miljoen ton voedsel wordt vernietigd of omgeleid naar niet-menselijke consumptie per jaar. Aflatoxinen zijn krachtige carcinogenen normaal opgebouwd door de schimmel Aspergillus flavus en A. parasiticus in granen, noten, wortelgewassen en andere landbouwproducten. Silencing van vijf aflatoxine-synthese genen door RNA interferentie (RNAi) in pinda planten werd gebruikt voor aflatoxine accumulatie na inenting met A. controle flavus. Voorheen bestond er geen methode om de effectiviteit van RNAi in individuele pinda transgene gebeurtenissen te analyseren, omdat deze meestal bij weinig zaden en traditionele grote veldexperimenten onder aflatoxine-bevorderlijk omstandigheden werden geen optie. In het veld, de waarschijnlijkheid om natuurlijk besmette zaden vaak 1/100 tot 1/1,000. Bovendien wordt aflatoxinebesmetting niet uniform verdeeld. Onze werkwijze gebruikt weinig zaden per transgene gebeurtenis met stukjes verwerkt voor real-time PCR (RT-PCR) of kleine RNA-sequencing en voor de analyse van aflatoxine accumulatie van ultra-vloeistofchromatografie (UPLC). RNAi-expressie pinda lijnen 288-72 en 288-74, kwamen tot 100% reductie (p≤0.01) in aflatoxine B 1 en B2 ten opzichte van de controle die tot opgelopen tot 14.000 ng. G -1 van aflatoxine B 1 bij geïnoculeerd met A. aflatoxigenic flavus. Als referentie, de maximale totaal van aflatoxines toegestane voor menselijke consumptie in de Verenigde Staten is 20 ng. G -1. Dit protocol beschrijft de toepassing van RNAi-gemedieerde controle van aflatoxines in transgene pinda zaden en methoden voor de evaluatie. Wij zijn van mening dat de toepassing ervan in de fokkerij van de pinda en andere gewassen snelle vooruitgang zal brengen op dit belangrijke gebied van de wetenschap, Medicijnen en menselijke voeding, en zal een belangrijke bijdrage leveren aan de internationale inspanningen om aflatoxines, en mogelijk andere mycotoxinen te controleren in grote voedselgewassen.
Introduction
Ongeveer 4,5 miljard mensen chronisch blootgesteld aan aflatoxines 1, de meest krachtige kankerverwekkende stoffen in de natuur 2 bekend. Deze verontreinigen mycotoxinen 25% van de voedselgewassen van de wereld 3, zoals maïs, cassave, rijst, noten, granen en kruiden. 4. Aflatoxines oorzaak dwerggroei bij kinderen van 5, aantasting van het immuunsysteem 6, aanwezig in 58% van hepatocellulaire carcinomen-zijn in de menselijke biopsies 7,8, en honderden mensen te doden tijdens de periodieke uitbarstingen van aflatoxicose 9,10. Aflatoxinen zijn polyketide afgeleid mycotoxinen normaal geproduceerd door Aspergillus flavus en A. parasiticus; aflatoxinen B 1 en B2 worden geproduceerd door A. flavus, terwijl A. parasiticus produceert G 1 en G2. De chemische structuur van deze verbindingen en een chromatogram dat de scheiding door UPLC zijn weergegeven in figuur 1. </strong>
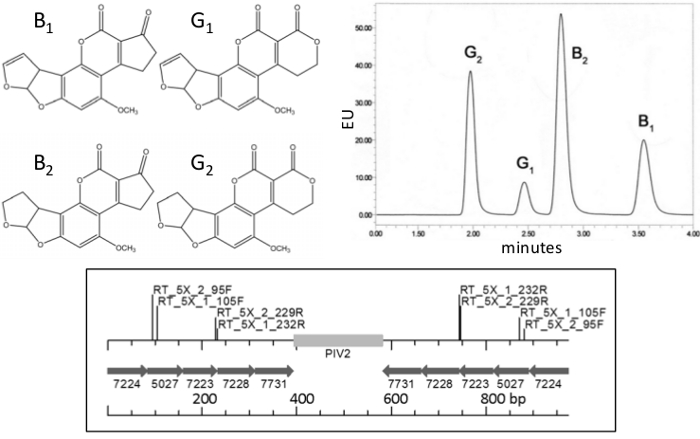
Figuur 1. aflatoxinen en RNAi invoegen Top. Chemische structuur (links) en voorbeeld chromatogram (rechts) van de vier meest voorkomende polyketide afgeleid aflatoxinen: B 1, B 2, G 1 en G2, geproduceerd door Aspergillus parasiticus, A . flavus produceert B 1 en B 2 Bodem: Schema van genfragmenten in de RNAi construct p5XCAPD gebruikt voor pinda transformatie, getallen onder pijlen zijn gen-fragment toetreding nummers in de Aspergillus flavus genoom;. PIV2: aardappel intron; bp: basenparen; RT_5X_1 en RT_5X_2. Real-Time PCR-primer locaties Klik hier om een grotere versie van deze figuur te bekijken.
Economische verliezen in de export als gevolg van aflatoxines in pinda alleen meer dan $ 450.000.000 $ indien berekend op basis van de 4 ng. G -1 limiet van aflatoxine toegestaan voor menselijke consumptie in de Europese Unie 11. Aflatoxinen zijn bekend voor 60 jaar 12; Echter, hoewel veel landbouwpraktijken werden ontwikkeld om hun effect, met inbegrip van de toepassing van andere schimmelstammen 13,14 te beperken, geen consistente methode zeggenschap bestaat, en resistente plantenrassen zijn niet beschikbaar. Testfabriek kiemplasma voor resistentie tegen aflatoxines is bijzonder moeilijk, omdat zelfs onder omstandigheden bevorderlijk voor pathogeen invasie, mycotoxine accumulatie is onvoorspelbaar en volgt niet een normale verdeling. Zo experimenten vereisen gewoonlijk grote aanplant gebieden, honderden zaden en meerdere monsters van 100-1,700 g tot variabiliteit van de gegevens 15,16 verminderen.
RNA interferentie wasontdekt in 1998 17; en de voordelen van 'silencing' worden momenteel onderzocht in een aantal nieuwe toepassingen, bijv., in humane therapieën tegen uitgezaaide borstkanker 18, 19 leverkanker, myeloïde leukemie 20 en in bestrijdingsmiddelen tegen insecten en nematoden 22 21. In planten, kunnen RNA interferentie signalen cel reizen naar cel, met kleine interfererende RNA (siRNA) en een hoog moleculair gewicht RNA die verantwoordelijk is voor de systemische posttranscriptionele gene silencing 23,24, zelfs binnen schimmels die in nauw contact met waardplant 25. De effectiviteit van RNAi op plant- silencing van fungale pathogenen genen is beschreven in enkele installatie pathosystems, deze, visueel onderzoek symptomen in de bovengrondse delen van de planten (blad) toegestaan ziekte kwantificering, dwz oomyceet Bremia in sla 26 , Puccinia in tarwe <sup> 27 en Fusarium in bananen 28. Veel moeilijker is om RNAi effectiviteit mycotoxinen in planten, met name aflatoxinen in grondnoten controle evalueren bladeren vertonen geen symptomen van infectie, de organen binnengedrongen (zaden) onder enkele centimeters van de bodem, het voorkomen van infectie is onvoorspelbaar, en slechts chemische analyse kan de aanwezigheid van aflatoxine te bepalen. Bovendien heeft elke transgene gebeurtenis in pinda normaal produceert weinig zaden (4-6 per plant); Daarom traditionele testen voor een no-aflatoxine accumulatie eigenschap in grote proefvelden, duurzame hele bijsnijden seizoenen, en het gebruik van honderden zaden is niet haalbaar. Een werkwijze wordt beschreven om te analyseren in minder dan een week, RNAi pindazaden aanwezigheid van transgene en een no-aflatoxine accumulatie eigenschap, met slechts weinig zaden.
Protocol
Representative Results
Discussion
Plant-gastheer RNAi-gemedieerde silencing van genen in schimmels aangetoond 27,43, maar er zijn geen publicaties tonen de haalbaarheid van RNAi-gemedieerde regulering van mycotoxine accumulatie in planten. Een beperkende factor voor deze studies pinda was het ontbreken van een werkwijze om een niet-aflatoxine accumulatie fenotype van individuele planten te evalueren, zoals bladeren vertonen geen symptomen van schimmelinfectie ondergrondse peulen. Bovendien zijn de niet-normaal accumulatie van aflatoxinen, en de noodzaak van grote monsters voor chemische analyse 15,16 de kwantificering van potentiële RNAi effect belemmerd op een enkele plant. De hier gepresenteerde methode omvat 72 uur experimenten met vijf zaden drie 24 h-interval bemonsteringen in drievoud (tabel 1, figuur 7) voeren. In vergelijking met de typische analyse van aflatoxinen dat niet minder dan 100 g zaad nodig, onze werkwijze is bijzonder geschikt voor individual transgene gebeurtenissen van pinda planten die aanvankelijk produceren niet meer dan twee of drie peulen.
RNA-gemedieerde silencing van aflatoxine synthese is aangetoond door genetische transformatie van Aspergillus flavus en A. parasiticus. Aangezien AFLR is een belangrijke regulator van aflatoxine productie in A. flavus en A. parasiticus 44,45, wordt het een interessant doelwit voor RNA-gemedieerde silencing bij planten. Echter, genetische variaties in AFLR aangetoond onder Aspergillus species 46 en die genetische varianten kon ontsnappen silencing indien er geen perfecte sequentie overeenkomen met RNAi signaal geproduceerd in de plantengastheer. Zo AFLR was een van de doelstellingen voor silencing in vector p5XCAPD, maar was niet de enige. Omgekeerde herhalingen van de AFLR gen ingebracht in A. flavus en A. parasiticus door transformatie resulteerde in zwijgen en minimale of geen production van aflatoxinen 47 (McDonald et al., 2005b). Ook silencing AFLD gen voorkomen aflatoxine productie met 98% in A. flavus en A. parasiticus in directe transformatie 48. Om de kans op succes in het systeem te verhogen, werd getransformeerd met pinda geïnverteerde repeat fragmenten van vijf genen betrokken bij aflatoxine productie A. flavus. Hier wordt aangetoond dat het gebruik van p5XCAPD dat doelstellingen voor silencing verschillende genen in het aflatoxine synthese route, 90% -100% lager gehalte aan aflatoxine B 1 en B 2 in lijn 288-72 werden bereikt, en 60-100% lagere niveaus opgebouwd in lijn 288-74 vergeleken met de controle, terwijl half zaadlobben werden geïnoculeerd met A. flavus, Figuren 4, 7. Het belangrijkste gedetecteerde deze werkwijze statistisch significante verschillen in aflatoxine accumulatie door lijnen 288-72, 288-74 vs. controle gedurende het experiment door toepassing van parametrische statistics, figuur 7. Gezien de kleine steekproef, is het belangrijk om de noodzaak benadrukken het gebruik van een krachtige methode voor aflatoxinen, deze experimenten werden geanalyseerd met UPLC met een hoge resolutie, vijf-voudige hogere prestaties en drie maal hogere gevoeligheid heeft dan detecteert HPLC 49.
Expressie van de RNAi insert in 288-74 werd pas ontdekt in onvolwassen zaadlobben (geel) bij 24 uur incubatie. De RNAi insert werd niet gedetecteerd door RT-PCR op rijpe zaadlobben van 288-74 op 24 uur, of op een looptijd groep op 48 uur, figuur 6. Ditzelfde fenomeen werd waargenomen in andere RNAi transgene pinda lijnen (Arias, RS, 2015 ongepubliceerd), waarbij gewoonlijk RNAi transcripten alleen werden gedetecteerd op onrijpe zaadlobben in 24 uur. RNA-monsters werden behandeld met DNAse voor cDNA-synthese, de gegevens werden genormaliseerd op het niveau van actine expressie en geen bewijs van DNA verontreiniging waargenomen. Indien DNA in de monsters aanwezig zijn, het moetzijn gedetecteerd in het 48 hr samples, maar dat was niet altijd het geval. Expressie onder controle van de 35S-promotor niet altijd uniform; het kan worden beïnvloed door omgevingsfactoren 50, type weefsel en het ontwikkelingsstadium 51,52. Op hetzelfde moment, in het pad van RNA-interferentie, de snelheid van mRNA verval en de snelheid van siRNA verval aanzienlijk 53 variëren. Het is mogelijk dat een snelle afbraak van het mRNA door het mechanisme van RNA interferentie mRNA-detectie kunnen voorkomen bij 48 uur incubatie. Of afwezigheid van expressie bij 48 uur was het gevolg van lage 35S-promoter gedreven transcriptie, of een snelle afbraak van dsRNA door Dicer nog worden beantwoord. Zo zou de detectie van kleine RNA's door high throughput sequencing een beter inzicht in de processen die plaatsvinden door middel van RNAi 54 geven in deze experimenten. Echter, omdat RNA-silencing verspreidt systemisch, voornamelijk via het floëem van photosynthaat bronnen putten sucrose (in casu pindazaden) 55, het zwijgen van aflatoxine-synthese kan plaatsvinden in zaden zonder lokale expressie van RNAi insert. Veel onderzoek moet nog worden gedaan om de drempelwaarde van kleine interfererende RNA's (siRNA's) die nodig zijn om aflatoxine accumulatie in zaden te voorkomen vast te stellen. Het is belangrijk te benadrukken dat zowel mRNA expressie van de RNAi construct (figuur 6), en accumulatie van aflatoxinen B 1 en B2 (figuur 7) vertoonde verschillende resultaten van onrijpe (geel) vs. volwassen (bruin) zaadlobben. Pinda planten onbepaalde groei, dat wil presenteren ze bij oogst verschillende looptijd pods, figuur 2. Bovendien zaden van verschillende rijpheid groepen verschillen in hun chemische samenstelling, bijv., 2,4% sucrose in onrijpe zaden, en 1,9% in rijpe zaden onder dezelfde veldomstandigheden 56,57. Dus, om de werkelijke efficienc begrijpeny van RNA-gemedieerde regulering van aflatoxine accumulatie, is het belangrijk om volwassenheid groepen afzonderlijk te onderzoeken.
Een natuurlijke afweer van pindazaden is de productie van fytoalexinen, die varieert in de verscheidenheid van verbindingen geproduceerd en hun relatieve hoeveelheden afhankelijk van de looptijd van de zaden en milieuomstandigheden 58-61, en het is bijzonder hoger in vergelijking met embryo zaadlobben 62. Embryo's ook significant hogere concentraties van nucleïnezuren, zowel DNA als RNA dan de zaadlobben (RS Arias, ongepubliceerd). Zoals pinda zaden rijpen, veranderingen in hun fysiologie en chemische samenstelling optreden 63. Fenolische antioxidanten in pinda testa vorm gecondenseerde tannines met fungistatische activiteit 64; Dit blijkt ook uit de mesocarp kleur die volwassenheid fasen, geel tot zwart 35 weerspiegelt, zoals de inhoud van tannines en fenolen verhoogt met een looptijd van 65 jaar. Aldus aanwezigheid van tESTA of embryo's in het experiment, gezien hun antimicrobiële eigenschappen kunnen schimmelgroei beperkt en derhalve overschat het effect van RNAi derhalve werden zij verwijderd. Ook het verwijderen van testa en embryo helpt beperken de bronnen van variatie in de analyse, zoals helft zaadlob dat draagt het embryo zal fytoalexinen meer RNA inhoud.
Naast de analyse naar looptijd groepen en verwijdering van testa en embryo deze experimenten, is het belangrijk te benadrukken enkele waarnemingen: a) hoewel de resultaten worden getoond voor maximaal 96 uur incubatie, is het raadzaam om niet meer dan 72 hr om consistente resultaten te verkrijgen, als zaden word afgebroken door 96 uur; en b) dat de helft zaadlobben van dezelfde zaad, hoewel steekproefsgewijs, niet volledig onafhankelijke steekproeven vormen, RT-PCR en aflatoxine accumulatie in transgene events toonden minimale verschillen tussen de zaden. Ook een nauwkeurige schimmelsporen mee volume inoculums van 2 pi, en toepassing van sporen op het snijvlak van de zaadlobben vermijden druipt aan de zijkanten belangrijk dat de gekiemde sporen worden blootgesteld aan het plantenweefsel. Het water / agar op de platen moet op 1,5% (w / v), zachte agar veroorzaakt afspoeling van sporen zoals op het laatste frame van Figuur 4 (onderste). Moet de beschikbaarheid zaad van een bepaalde transgene geval beperkt zijn, kan de bemonstering worden uitgevoerd in tweevoud in plaats van drievoudige verkrijgen van vergelijkbare resultaten (dat wil zeggen, figuur 7); zal echter drievoudige monsters helpen standaardfout verminderen. De enige beperking van deze werkwijze is dat het een uiterst gevoelig systeem (UPLC) aflatoxine detectie / kwantificering vereist, maar tevens teneinde de kans overschatten het effect van RNAi aflatoxinen mag niet worden gedetecteerd door minder gevoelige methoden.
Samenvattend biedt deze methode voor het eerst een betrouwbare benadering van de studieeffect van RNAi in de controle van aflatoxinen. Verminderen van de tijd voor een experiment van een hele teeltperiode tot minder dan een week, zal deze methode enorm versnellen van het onderzoek op RNAi-pinda / Aspergillus pathosystem naar de mitigatie en / of eliminatie van aflatoxines.
Disclosures
The authors have nothing to disclose.
Acknowledgements
This work received the financial support of USDA-ARS CRIS project 6604-21000-004-00D, CRIS project 6604-42000-008-00D, and USAID Feed-the-Future program Agreement number 58-0210-3-012. We thank Valerie Orner, LaTanya Johnson, Joseph Powell and Kathy Gray for their technical assistance. Mention of trade names or commercial products in this article is solely for the purpose of providing specific information and does not imply recommendation or endorsement by the US Department of Agriculture.
Materials
| Primers, oligonucleotides | DNA Technologies, Coralville, IA, USA | n/a | |
| Dneasy Plant Mini Kit | Qiagen, Valencia, CA | 69106 | |
| Czapek Dox agar medium | Oxoid, by Thermo Fisher Scientific, Waltham, MA | CM0095 | |
| Agar | Thermo Fisher Scientific, Waltham, MA | BP 1423 | |
| Freezer -80°C | n/a | n/a | |
| Aluminum Oxide, Al2O3 | Fisher Scientific | A941 | |
| SPE Reservoirs 1.5 mL | Grace Davison Discovery Scientific | 210011 | |
| Frits for 1.5 mL SPE reservoir | Grace Davison Discovery Scientific | 211401 | |
| Autosampler vials | Waters Corporation, Milford, MA | 186005221 | |
| Waters Acquity Ultra-Performance Liquid-Chromatography (UPLC) instrument; UPLC-H-Class Quaternary Solvent Manager; UPLC Sample Manager; UPLC Fluorescent detector (FLR); UPLC BEH C18 2.1mmx50mm, 1.7mm column | Waters Corporation, Milford, MA | ||
| Finnigan LCQ Advantage MAX ion trap mass spectrometer, with Xcalibur version 1.4 software | Thermo Electron Corp., San Jose, CA | ||
| Aflatoxin standards, B1, B2, G1 and G2 | Sigma-Aldrich, St. Louis, MO | A6636; A9887; A0138; A0263 | |
| Systat Software 12.2 | SYSTAT Software Inc., Point Richmond, CA | ||
| Trizol reagent | Invitrogen, CA | 15596-018 | |
| SuperScript III First Strand Synthesis Super Mix | Invitrogen, CA | 11752-050 | |
| ABI 7500 Real-Time PCR | Lifetechnologies, Grand Island, NY | 4406984 | |
| Luria Broth-Miller | Fisher Scientific | R453642 | |
| pENTR1A | Invitrogen, CA | A10462 | |
| LR Clonase II enzyme mix | Invitrogen, CA | 11791-020 | |
| T4 DNA Ligase | NEB Biolabs | M0202L | |
| Gelrite | Sigma-Aldrich, St. Louis, MO | G1919 | |
| Acetosyringone | Sigma-Aldrich, St. Louis, MO | D134406 | |
| QIAcube robot workstation | Qiagen, Valencia, CA | 9001292 | |
| Antibiotics: kanamycin, cefotaxime, gentamicin; streptomycin | Goldbio, St. Louis, MO | cef.: C-104-25; kan: K-120-5; gent.: G-400-1; strep.: S-150-50 | |
| Platinum Taq DNA Polymerase High Fidelity | Invitrogen, CA | 11304-029 |
References
- Williams, J. H., et al. Human aflatoxicosis in developing countries: a review of toxicology, exposure, potential health consequences, and interventions. The American Journal of Clinical Nutrition. 80, 1106-1122 (2004).
- American Association for Cancer Research: AACR. An evaluation of chemicals and industrial processes associated with cancer in humans based on human and animal data: IARC Monographs Volumes 1 to 20. Cancer Research. 40, 1-12 (1980).
- Turner, P. C. The molecular epidemiology of chronic aflatoxin driven impaired child growth. Scientifica. , (2013).
- Rasooly, R., Hernlem, B., He, X., Friedman, M. Non-linear relationships between aflatoxin B1 levels and the biological response of monkey kidney vero cells. Toxins (Basel). 5, 1447-1461 (2013).
- Gong, Y. Y., et al. Determinants of aflatoxin exposure in young children from Benin and Togo, West Africa: the critical role of weaning. International Journal of Epidemiology. 32, 556-562 (2003).
- Eaton, D. L., Groopman, J. D. . The toxicology of aflatoxins: human health, veterinary, and agricultural significance. , (1994).
- Murugavel, K. G., et al. Prevalence of aflatoxin B1 in liver biopsies of proven hepatocellular carcinoma in India determined by an in-house immunoperoxidase test. Journal of Medical Microbiology. 56, 1455-1459 (2007).
- Wang, J. S., et al. Hepatocellular carcinoma and aflatoxin exposure in Zhuqing Village, Fusui County, People’s Republic of China. Cancer Epidemiology, Biomarkers & Prevention. 10, 143-146 (2001).
- Azziz-Baumgartner, E., et al. Case-control study of an acute aflatoxicosis outbreak, Kenya, 2004. Environmental Health Perspectives. 113, 1779-1783 (2005).
- Lye, M. S., Ghazali, A. A., Mohan, J., Alwin, N., Nair, R. C. An outbreak of acute hepatic encephalopathy due to severe aflatoxicosis in Malaysia. American Journal of Tropical Medicine and Hygiene. 53, 68-72 (1995).
- Villers, P. Aflatoxins and safe storage. Frontiers in Microbiology. 5, 158 (2014).
- Kensler, T. W., Roebuck, B. D., Wogan, G. N., Groopman, J. D. Aflatoxin: A 50-year odyssey of mechanistic and translational toxicology. Toxicological Sciences. 120, S28-S48 (2011).
- Dorner, J. W., Cole, R. J., Wicklow, D. T. Aflatoxin reduction in corn through field application of competitive fungi. Journal of Food Protection. 62, 650-656 (1999).
- Cotty, P. J., Bhatnagar, D. Variability among atoxigenic Aspergillus flavus strains in ability to prevent aflatoxin contamination and production of aflatoxin biosynthetic-pathway enzymes. Applied and Environmental Microbiology. 60, 2248-2251 (1994).
- Whitaker, T. B. Standardisation of mycotoxin sampling procedures: an urgent necessity. Food Control. 14, 233-237 (2003).
- Whitaker, T. B., Dorner, J. W., Giesbrecht, F. G., Slate, A. B. Variability among aflatoxin test results on runner peanuts harvested from small field plots. Peanut Science. 31, 59-63 (2004).
- Fire, A., et al. Potent and specific genetic interference by double-stranded RNA in Caenorhabditis elegans. Nature. 391, 806-811 (1998).
- Rafael, D., et al. EMT blockage strategies: Targeting Akt dependent mechanisms for breast cancer metastatic behaviour modulation. Current Gene Therapy. , (2015).
- Li, G., Chang, H., Zhai, Y. P., Xu, W. Targeted silencing of inhibitors of apoptosis proteins with siRNAs: a potential anti-cancer strategy for hepatocellular carcinoma. Asian Pacific. Journal of Cancer Prevention: APJCP. 14, 4943-4952 (2013).
- Koldehoff, M. Targeting bcr-abl transcripts with siRNAs in an imatinib-resistant chronic myeloid leukemia patient: challenges and future directions. Methods in Molecular Biology. 1218, 277-292 (2015).
- Zhang, J., et al. Pest control. Full crop protection from an insect pest by expression of long double-stranded RNAs in plastids. Science. 347, 991-994 (2015).
- Ajjappala, H., Chung, H. Y., Sim, J. S., Choi, I., Hahn, B. S. Disruption of prefoldin-2 protein synthesis in root-knot nematodes via host-mediated gene silencing efficiently reduces nematode numbers and thus protects plants. Planta. 241, 773-787 (2015).
- Jose, A. M., Hunter, C. P. Transport of sequence-specific RNA interference information between cells. Annual Review of Genetics. 41, 305-330 (2007).
- Vazquez, F., Hohn, T. Biogenesis and biological activity of secondary siRNAs in plants. Scientifica. , (2013).
- Tinoco, M. L. P., Dias, B. B. A., Dall’Astta, R. C., Pamphile, J. A., Aragao, F. J. L. In vivo trans-specific gene silencing in fungal cells by in planta expression of a double-stranded RNA. BMC Biology. 8, (2010).
- Govindarajulu, M., Epstein, L., Wroblewski, T., Michelmore, R. W. Host-induced gene silencing inhibits the biotrophic pathogen causing downy mildew of lettuce. Plant Biotechnology Journal. , (2014).
- Yin, C., Jurgenson, J. E., Hulbert, S. H. Development of a host-induced RNAi system in the wheat stripe rust fungus Puccinia striiformis f. sp. tritici. Molecular Plant-Microbe Interactions. 24, 554-561 (2011).
- Ghag, S. B., Shekhawat, U. K., Ganapathi, T. R. Host-induced post-transcriptional hairpin RNA-mediated gene silencing of vital fungal genes confers efficient resistance against Fusarium. wilt in banana. Plant Biotechnology Journal. 12, 541-553 (2014).
- Filichkin, S. A., et al. Efficiency of gene silencing repeats vs. transitive RNAi in Arabidopsis: direct inverted vectors. Plant Biotechnology Journal. 5, 615-626 (2007).
- Sciaky, D., Montoya, A. L., Chilton, M. D. Fingerprints of Agrobacterium Ti Plasmids. Plasmid. 1, 238-253 (1978).
- Clark, D. J., Maaloe, O. DNA Replication and Division Cycle in Escherichia coli. Journal of Molecular Biology. 23, 99-112 (1967).
- Murashige, T., Skoog, F. A revised medium for rapid growth and bio assays with tobacco tissue cultures. Physiol Plantarum. 15, 473-497 (1962).
- Srinivasan, T., Kumar, K. R. R., Kirti, P. B. Establishment of efficient and rapid regeneration system for some diploid wild species of Arachis. Plant Cell Tissue and Organ Culture. 101, 303-309 (2010).
- Gomes, A. L. V., et al. Single-tube nested PCR using immobilized internal primers for the identification of dengue virus serotypes. Journal of Virology Methods. 145, 76-79 (2007).
- Williams, E. J., Drexler, J. S. A non-destructive method for determining peanut pod maturity. Peanut Science. 8, 134-141 (1981).
- Sobolev, V. S., Dorner, J. W. Cleanup procedure for determination of aflatoxins in major agricultural commodities by liquid chromatography. Journal of AOAC International. 85, 642-645 (2002).
- Biselli, S., Hartig, L., Wegner, H., Hummert, C. Analysis of Fusarium. toxins using LC-MS-MS: Application to various food and feed matrices. LC GC North America. 23, 404-413 (2005).
- Arias, R. S., Sobolev, V. S., Orner, V. A., Dang, P. M., Lamb, M. C. Potential involvement of Aspergillus flavus laccases in peanut invasion at low water potential. Plant Pathology. 63, 353-363 (2014).
- Dang, P. M., Chen, C. Y., Holbrook, C. C. Evaluation of five peanut (Arachis hypogaea) genotypes to identify drought responsive mechanisms utilising candidate-gene approach. Functional Plant Biology. 40, 1323-1333 (2013).
- Schmittgen, T. D., Livak, K. J. Analyzing real-time PCR data by the comparative C-T method. Nature Protocols. 3, 1101-1108 (2008).
- Amaike, S., Keller, N. P. Aspergillus flavus. Annual Review of Phytopathology. 49, 107-133 (2011).
- Nowara, D., et al. HIGS: Host-induced gene silencing in the obligate biotrophic fungal pathogen Blumeria graminis. Plant Cell. 22, 3130-3141 (2010).
- Woloshuk, C. P., et al. Molecular characterization of aflR, a regulatory locus for aflatoxin biosynthesis. Applied and Environmental Microbiology. 60, 2408-2414 (1994).
- Price, M. S., et al. The aflatoxin pathway regulator AflR induces gene transcription inside and outside of the aflatoxin biosynthetic cluster. FEMS Microbiology Letters. 255, 275-279 (2006).
- Ehrlich, K. C., Montalbano, B. G., Cotty, P. J. Sequence comparison of aflR from different Aspergillus. species provides evidence for variability in regulation of aflatoxin production. Fungal Genetics and Biology. 38, 63-74 (2003).
- McDonald, T., Brown, D., Keller, N. P., Hammond, T. M. RNA silencing of mycotoxin production in Aspergillus and Fusarium species. Molecular Plant Microbe Interactions. 18, 539-545 (2005).
- Abdel-Hadi, A. M., Caley, D. P., Carter, D. R., Magan, N. Control of aflatoxin production of Aspergillus flavus. and Aspergillus parasiticus. using RNA silencing technology by targeting aflD. (nor-1) gene. Toxins (Basel). 3, 647-659 (2011).
- Swartz, M. E. Ultra performance liquid chromatography (UPLC): An introduction: Separation Science Redefined. LCGC North America. , 8-14 (2005).
- Maghuly, F., Khan, M. A., Fernandez, E. B., Druart, P., Watillon, B., Laimer, M. Stress regulated expression of the GUS-marker gene (uidA) under the control of plant calmodulin and viral 35S promoters in a model fruit tree rootstock: Prunus incisa x serrula. Journal of Biotechnology. 135, 105-116 (2008).
- de Mesa, M. C., Santiago-Doménech, N., Pliego-Alfaro, F., Quesada, M. A., Mercado, J. A. The CaMV 35S promoter is highly active on floral organs and pollen of transgenic strawberry plants. Plant Cell Reports. 23, 32-38 (2004).
- Sunilkumar, G., Mohr, L., Lopata-Finch, E., Emani, C., Rathore, K. S. Developmental and tissue-specific expression of CaMV 35S promoter in cotton as revealed by GFP. Plant Molecular Biology. 50, 463-474 (2002).
- Groenenboom, M. A. C., Maree, A. F. M., Hogeweg, P. The RNA silencing pathway: The bits and pieces that matter. PLoS Computational Biology. 1, 155-165 (2005).
- Zhao, D., Song, G. Q. High-throughput sequencing as an effective approach in profiling small RNAs derived from a hairpin RNA expression vector in woody plants. Plant Science: an International Journal of Experimental Plant Biology. 228, 39-47 (2014).
- Kamthan, A., Chauduri, A., Kamthan, M., Datta, A. Small RNAs in plants: recent development and application for crop improvement. Frontiers in Plant Science. 6, 208 (2015).
- Manda, A., Bodapati, P. N., Rachaputi, N. C., Wright, G., Fukai, S. . Aflatoxins and their relationship with sugars in peanut (Arachis hypogaea L). , (2004).
- Uppala, S. S. . Factors affecting pre-harvest aflatoxin contamination of peanut (Arachis hypogaea L). , (2011).
- Sobolev, V. S. Localized production of phytoalexins by peanut (Arachis hypogaea) kernels in response to invasion by Aspergillus species. Journal of Agricultural and Food Chemistry. 56, 1949-1954 (2008).
- Sobolev, V. S., Guo, B. Z., Holbrook, C. C., Lynch, R. E. Interrelationship of phytoalexin production and disease resistance in selected peanut genotypes. Journal of Agricultural and Food Chemistry. 55, 2195-2200 (2007).
- Sobolev, V. S., Neff, S. A., Gloer, J. B. New stilbenoids from peanut (Arachis hypogaea) seeds challenged by an Aspergillus caelatus strain. Journal of Agricultural and Food Chemistry. 57, 62-68 (2009).
- Dorner, J. W., Cole, R. J., Sanders, T. H., Blankenship, P. D. Interrelationship of kernel water activity, soil temperature, maturity, and phytoalexin production in preharvest aflatoxin contamination of drought-stressed peanuts. Mycopathologia. 105, 117-128 (1989).
- Sobolev, V. S. Production of phytoalexins in peanut (Arachis hypogaea) seed elicited by selected microorganisms. Journal of Agricultural and Food Chemistry. 61, 1850-1858 (2013).
- Basha, S. M. M., Cherry, J. P., Young, C. T. Changes in free amino acids, carbohydrates, and proteins of maturing seeds from various peanut (Arachis hypogaea L.) cultivars. Cereal Chemistry. 53, 586-596 (1976).
- Lansden, J. A. Aflatoxin inhibition and fungistasis by peanut tannins. Peanut Science. 9, 17-20 (1982).
- Yen, G. C., Duh, P. D., Tsai, C. L. Relationships between antioxidant activity and maturity of peanut hulls. Journal of Agricultural and Food Chemistry. 41, 67-70 (1993).

